Science:利用源自宿主细胞的非典型crRNA可实现Cas9的多重RNA检测
来源:本站原创 2021-05-04 15:05
2021年5月4日讯/生物谷BIOON/---CRISPR-Cas免疫系统通过CRISPR RNA(crRNA)的引导降解外来遗传物质。crRNA作为间隔重复序列单元被编码在这种系统的CRISPR阵列中。每个crRNA通常由对CRISPR阵列转录而来的前体进行加工而成,然后与这种系统的Cas效应核酸酶合作,直接裂解靶核酸。在作为Cas9核酸酶和许多CRISP
2021年5月4日讯/生物谷BIOON/---CRISPR-Cas免疫系统通过CRISPR RNA(crRNA)的引导降解外来遗传物质。crRNA作为间隔重复序列单元被编码在这种系统的CRISPR阵列中。每个crRNA通常由对CRISPR阵列进行转录而来的前体进行加工而成,然后与这种系统的Cas效应核酸酶(比如Cas9)合作,直接裂解靶核酸。在作为Cas9核酸酶和许多CRISPR技术的来源的II型系统中,crRNA加工和随后Cas9的DNA靶向需要反式激活crRNA(trans-activating crRNA, tracrRNA)。tracrRNA与转录的CRISPR阵列中每个crRNA的“重复序列”部分杂交。然后,宿主的RNase III裂解形成的RNA茎,产生加工后的crRNA:tracrRNA双链,以供Cas9使用。目前还不清楚的是crRNA是否局限于CRISPR-Cas位点,还是可以从基因组的其他地方获得。
在一项新的研究中,来自德国维尔茨堡大学等研究机构的研究人员发现crRNA可以来自CRISPR-Cas位点以外的宿主RNA,这促进他们开一种新的基于Cas9的诊断平台,允许在一次测试中可扩展地检测多种生物标志物。相关研究结果于2021年4月27日在线发表在Science期刊上,论文标题为“Noncanonical crRNAs derived from host transcripts enable multiplexable RNA detection by Cas9”。

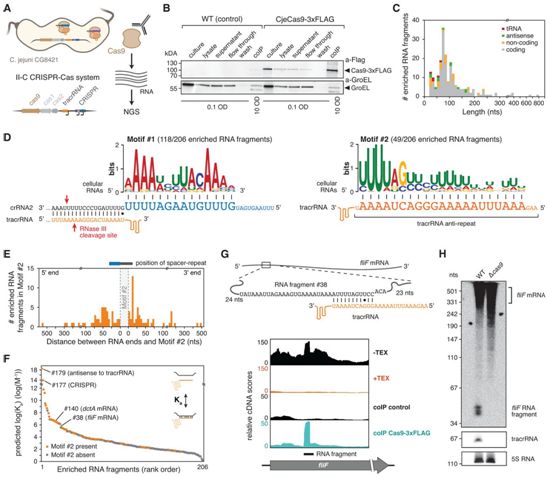
这些作者利用RIP-seq技术和MEME在线工具寻找揭示来自空肠弯曲菌(Campylobacter jejuni)的Cas9(CjeCas9)可以结合的细胞宿主RNA。他们发现两个显著的序列基序:基序#1和基序#2。基序#1与crRNA2引导部分的13个核苷酸互补。令人惊讶的是,基序#2与tracrRNA反重复结构域(anti-repeat domain)的21个核苷酸互补。由于作为crRNA生物发生的一部分,该结构域通常与crRNA重复序列杂交,基序#2提出了这些细胞RNA与tracrRNA杂交从而潜在地成为具有类似crRNA功能的RNA的有趣可能性。
对于含有基序#2的富集RNA片段,这些作者将每个RNA片段中的基序#2与crRNA的间隔序列-重复序列进行比对,并测量每个RNA片段中对应于crRNA的间隔序列或重复序列的长度。最常见的是,基序#2的间隔序列部分比典型的crRNA间隔序列长15个核苷酸,而基序#2的重复序列部分与典型的crRNA重复序列大小相同,类似于crRNA的略微延伸版本。其次,他们预测了每个RNA片段如何与tracrRNA反重复结构域进行碱基配对。预测具有基序#2的RNA片段的结合亲和力明显高于没有基序#2的RNA片段(p = 3×10-7)。然而,尽管缺乏基序#2,多个RNA片段据预测与tracrRNA反重复结构域发生强烈配对,这可能是由于所形成的RNA双链中的隆起造成基序#2的不连续。对于这些RNA片段和那些含有基序#2的RNA片段,每个RNA和tracrRNA反重复结构域的预测相互作用始终包含不完美配对的RNA双链,在反重复结构域的3′端附近配对最广泛。他们对基序#1进行了类似的观察。
这些类似crRNA的RNA提出了一个问题,即这些相同的RNA是否存在于这些作者先前对空肠弯曲菌菌株NCTC11168的RIP-seq分析中。他们发现96个富集的RNA片段中有7个被预测为比至少一个crRNA更紧密地结合tracrRNA反重复结构域。其中的两个RNA片段(来源于fliF mRNA和dctA mRNA)与空肠弯曲菌菌株CG8421中发现的RNA片段相匹配。来自fliF mRNA的RNA片段可以通过Northern印迹在总RNA和RIP-seq样品中检测到,但在cas9缺失后消失了。dctA RNA片段只在一个菌株中被微弱地检测到。虽然在标准生长条件下,cas9的缺失并没有明显扰乱体内FliF蛋白的水平,但在NCTC11168中剔除CRISPR阵列会增加fliF RNA片段的水平。fliF RNA片段是一种加工产物,这一点用Terminator外切酶处理后得到了证实。因此,这些类似crRNA的RNA也存在于空肠弯曲菌菌株NCTC11168中,并且基于fliF中共享的tracrRNA结合位点,可能存在于其他的空肠弯曲菌菌株中。
空肠弯曲菌中Cas9结合的细胞RNA片段类似于crRNA。图片来自Science, 2021, doi:10.1126/science.abe7106。
这些富集的RNA片段的Cas9结合、预测的tracrRNA配对和长度分布表明,tracrRNA与内源性RNA配对,形成了非典型的crRNA(ncrRNA)。因此,预计ncrRNA可以将Cas9引导到两侧为前间区序列邻近基序(protospacer-adjacent motif, PAM)的互补DNA靶标上,类似于典型的crRNA。由于产生检测到的ncrRNA的所有基因都没有正确放置的PAM,因此预计ncrRNA不能引导Cas9裂解它们的起始基因组位点。
从细菌病原体空肠弯曲菌的天然CRISPR-Cas9系统的特征出发,这些作者发现细胞的RNA可以通过与tracrRNA的杂交成为ncrRNA的来源。这一发现将ncrRNA添加到了自然界中发现的向导RNA(gRNA)清单中,包括crRNA、与tracrRNA反重复结构域类似地配对的scaRNA以及在tracrRNA的上游转录形成的“天然”sgRNA。重要的是,这些先前的例子都是在CRISPR-Cas位点内编码的。到目前为止,仍不清楚ncrRNA是否在空肠弯曲菌中起到生理作用。因此,未来的研究将有助于澄清来自CRISPR阵列之外的ncrRNA是否是宿主可耐受的虚假脱靶产物,或者它们是否赋予了尚未发现的适应性免疫之外的功能。
这些作者进一步证实,重编程的tracrRNA(reprogrammed tracrRNA, Rptr)可以将感兴趣的RNA的存在与Cas9的序列特异性DNA靶向联系起来。这种能力可以使Cas9在体内的应用(比如多重转录记录或转录依赖的编辑)成为可能。最直接的应用涉及通过LEOPARD(Leveraging Engineered tracrRNAs and On-target DNAs for PArallel RNA Detection, 利用工程tracrRNA和在靶DNA进行平行RNA检测)在体外进行多重RNA检测。
目前的CRISPR诊断主要依靠Cas12a或Cas13寻找样品中的双链DNA或RNA靶标,在此过程中,靶标识别引起非特异性的单链DNA或RNA裂解荧光报告分子。非特异性荧光读取实际上将一次测试限制为针对一个靶序列。相反,重编程tracrRNA将识别到的RNA转化为ncrRNA,这将引导Cas9靶向匹配的DNA。Cas9结合或裂解匹配的DNA序列将表明样品中存在识别到的RNA。由于每个DNA靶标的序列都是独一无二的,大量的靶序列可以在一次测试中被平行监测。这些作者将由此产生的诊断平台称为LEOPARD。
为了开始评估LEOPARD,这些作者使用T7 RNA聚合酶转录的RNA、市售的SpyCas9蛋白(来自产脓链球菌的Cas9)和线性DNA靶标进行了简化的体外反应。他们从对应于空肠弯曲菌CJ8421_04975中的一个合成ncrRNA位点的RNA开始(SpyCas9下的n4)。在没有加入RNase III或RNase A的情形下,引入退火步骤使重编程的tracrRNA与T7 RNA聚合酶转录的ncrRNA杂交,产生了DNA靶向裂解。裂解效率也与同等的crRNA:tracrRNA对相似,即使ncrRNA序列在它的任一端发生延长,也是如此。退火步骤的时间长度也可以通过快速冷却样品来最小化。通过退火步骤,在酵母总RNA过量100倍的情况下发生了有效的裂解,但只有在ncrRNA存在的情况下才会发生裂解。因此,LEOPARD可以根据DNA靶标的裂解指示特定感兴趣的RNA的存在,并可以通过进一步的优化来简化。
LEOPARD是对现有的主要基于Cas12a或Cas13的CRISPR诊断平台的补充,同时在单一反应中提供可扩展的多重检测,这种多重检测可实现单碱基分辨率。他们基于凝胶电泳或生物分析仪提供了多重检测的原则性证明,尽管这两种方法都很难实现检测十几个以上的靶标。相反,结合微阵列或下一代测序,通过将特定RNA的存在与标记的Cas9的结合或标记的DNA靶标在芯片上的特定位置的裂解联系起来,有可能监测多达数百万个靶标。微阵列和下一代测序也可以提高检测的灵敏度,因为在一个给定的簇集中DNA分子的数量有限,有可能规避对RNA预扩增的需要。还可以开发更简单的涉及横向流动检测的设置,与其他CRISPR诊断法相平行使用。
随着进一步的开发,LEOPARD可能成为一个强大的诊断工具,不仅用于检测单个核苷酸存在差异的病毒变体;它还可能用于其他应用,如筛选癌症突变,识别病原体和抗生素耐药性标志物,或确定药物敏感性的基因表达谱。此外,将重编程的tracrRNA扩展到V型CRISPR系统内的tracrRNA依赖性核酸酶,可能有助于整入它们的独特属性,如信号放大或可编程的转座。
在系统地扰乱标准的crRNA:tracrRNA双链时,这些作者发现许多偏差---特别是在重复序列的5′端之外--被Cas9所容忍,并且仍然靶向结合设计的DNA靶标。然而,尽管有这种非特异性结合,但这种靶向仍由上游的向导序列和对侧翼PAM的需求决定。因此,反重复结构域杂交和向导序列依赖性的DNA靶向都可能限制脱靶活性。虽然这些作者在体外或体内没有观察到任何可检测到的脱靶现象,但未来的研究工作可能在考虑到潜在的脱靶以及靶向活性时制定出重编程tracrRNA的设计规则,类似于现有的sgRNA设计算法。反过来,这些规则将有助于推进任何RNA的利用,使其成为CRISPR技术的序列特异性指南。(生物谷 Bioon.com)
参考资料:
Chunlei Jiao et al. Noncanonical crRNAs derived from host transcripts enable multiplexable RNA detection by Cas9. Science, 2021, doi:10.1126/science.abe7106.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


