《Cell》重磅:北京大学张泽民课题组与勃林格殷格翰联合发表关于单细胞测序刻画肝癌免疫微环境动态特征的研究
来源:生物谷 2019-11-01 16:19
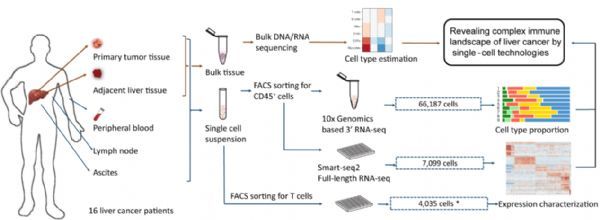
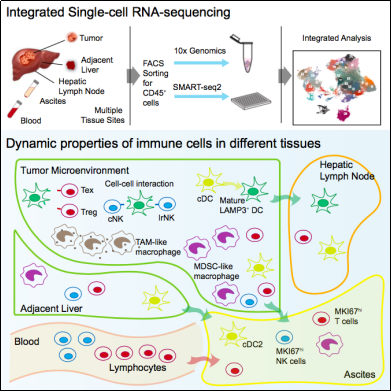
北京大学生命科学学院、北京未来基因诊断高精尖创新中心(ICG)、生物医学前沿创新中心(BIOPIC)张泽民课题组、首都医科大学附属北京世纪坛医院彭吉润课题组以及德国药企勃林格殷格翰(Boehringer Ingelheim)公司肿瘤免疫与免疫调节部门多位科学家,在国际期刊Cell上发表了题为Landscape and Dynamics of Single Immune Cells in Hepat
1. Forner A., Reig M. and Bruix J., Hepatocellular carcinoma, Lancet 391, 2018, 1301–1314.
2. Gnjatic S., Bronte V., Brunet L.R., Butler M.O., Disis M.L., Galon J., Hakansson L.G., Hanks B.A., Karanikas V., Khleif S.N., et al., Identifying baseline immune-related biomarkers to predict clinical outcome of immunotherapy, J. Immunother. Cancer 5, 2017, 44.
3. Ziegenhain C., Vieth B., Parekh S., Reinius B., Guillaumet-Adkins A., Smets M., Leonhardt H., Heyn H., Hellmann I. and Enard W., Comparative Analysis of Single-Cell RNA Sequencing Methods, Mol. Cell 65, 2017, 631–643.e4.
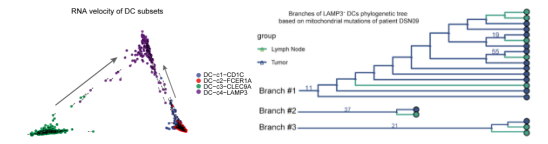
4. La Manno G., Soldatov R., Zeisel A., Braun E., Hochgerner H., Petukhov V., Lidschreiber K., Kastriti M.E., Lönnerberg P., Furlan A., et al., RNA velocity of single cells, Nature 560, 2018, 494–498.
5. Ludwig L.S., Lareau C.A., Ulirsch J.C., Christian E., Muus C., Li L.H., Pelka K., Ge W., Oren Y., Brack A., et al., Lineage Tracing in Humans Enabled by Mitochondrial Mutations and Single-Cell Genomics, Cell 176, 2019, 1325–1339.e22.
6. Michea P., Noël F., Zakine E., Czerwinska U., Sirven P., Abouzid O., Goudot C., Scholer-Dahirel A., Vincent-Salomon A., Reyal F., et al., Adjustment of dendritic cells to the breast-cancer microenvironment is subset specific, Nat. Immunol. 19, 2018, 885–897.
7. Zilionis R., Engblom C., Pfirschke C., Savova V., Zemmour D., Saatcioglu H.D., Krishnan I., Maroni G., Meyerovitz C.V., Kerwin C.M., et al., Single-Cell Transcriptomics of Human and Mouse Lung Cancers Reveals Conserved Myeloid Populations across Individuals and Species, Immunity 50, 2019, 1317–1334.e10.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。