师从诺奖得主David Baker,杨为博士全职加入深圳医学科学院,聚焦于从头设计蛋白质药物
来源:生物世界 2025-07-05 15:07
研究团队描述了螺旋凹面支架的开发,这种支架是为通常参与免疫受体相互作用的凸面靶位点量身定制的。
近日,华盛顿大学蛋白质设计研究所博士后杨为全职加入深圳医学科学院,成立蛋白质药物设计课题组。
杨为, 2019 年于清华大学获得生物学博士学位,导师为国内著名计算机辅助药物设计专家、蛋白质从头设计领军人物来鲁华教授。2019-2025 年在华盛顿大学蛋白质设计研究所从事博士后研究工作,合作导师为蛋白质从头设计先驱、2024 年诺贝尔化学奖得主 David Baker 教授。杨为博士专注于开发全新蛋白质-蛋白质相互作用计算设计方法,及其在免疫调控和肿瘤免疫治疗领域的应用。
蛋白质药物设计课题组将密切结合最新蛋白质计算设计方法发展前沿,利用平台和技术优势,形成算法开发-高通量验证-迭代优化的闭环聚焦于:
1)开发全新药用蛋白质设计方法及验证平台,为全新蛋白质药物开发提供技术支撑;
2)基于开发的算法设计高选择性生物大分子,用于癌症免疫治疗,提高靶向性,降低系统毒性。
2025 年 2 月 26 日,杨为博士作为第一作者,在 Nature Communications 期刊发表了题为:Design of high-affinity binders to immune modulating receptors for cancer immunotherapy 的研究论文。我们通过这篇论文了解下杨为博士的研究方向。

免疫细胞和肿瘤细胞上表达的受体对于维持体内平衡至关重要,它们通过上调或下调免疫反应来发挥作用。在过去的几十年里,癌症免疫疗法通过延长癌症患者的生存期彻底改变了肿瘤学领域。
诸如 CTLA-4、PD-1、LAG3 和 PD-L1 等抑制性受体以及诸如 IL2-R、IL10-R 和 TGFbRII 等细胞因子受体,它们分别调节免疫反应和控制免疫细胞的增殖与分化,已成为癌症免疫治疗的重要治疗靶点。
计算蛋白质设计的发展使得我们能够创造出靶向人类细胞表面受体的从头(de novo)蛋白质结合剂,这些结合剂具有高度的模块化、亲和力和稳定性。鉴于免疫调控网络的复杂性,以及联合疗法中靶向多个受体的兴趣日益浓厚,靶向免疫治疗靶点的高亲和力且性能良好的蛋白质结合剂可能会非常有用。这些受体中的许多都含有免疫球蛋白(Ig)折叠结构域,其表面凸起,因此,难以设计结合剂来靶向结合这些结构域。
研究团队推断,一组形状经过专门设计以与多种含凸面免疫球蛋白折叠结构的靶点相互作用的 5 螺旋凹面支架(5HCS),能够为设计靶向免疫治疗靶点的高稳定性和高亲和力结合剂提供起点。研究团队着手开发此类支架,并利用它们来设计靶向三种关键癌症免疫治疗相关受体(TGFbRII、CTLA-4 和 PD-L1)的高亲和力结合剂。
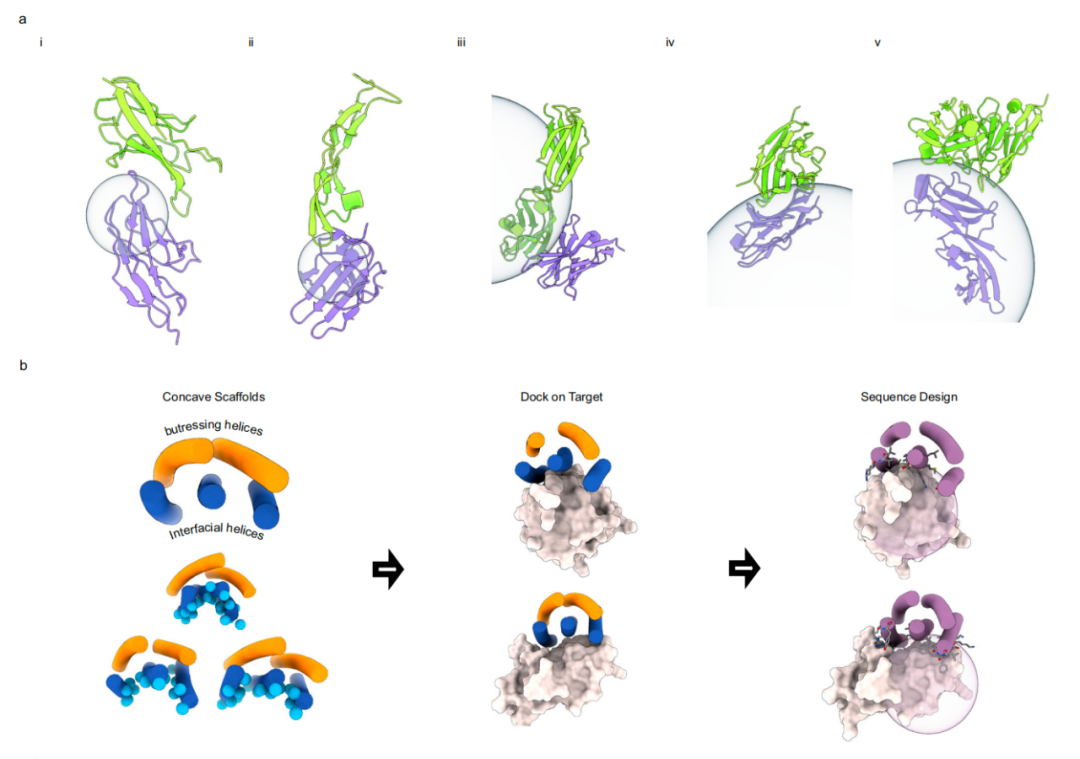
靶向免疫球蛋白样靶点凸界面的5HCS支架的设计
在这项新研究中,研究团队描述了螺旋凹面支架的开发,这种支架是为通常参与免疫受体相互作用的凸面靶位点量身定制的。他们利用这些支架设计出能与 TGFβRII、CTLA-4 和 PD-L1 结合的蛋白质,经过实验优化后,实现了低纳摩尔至皮摩尔级的高亲和力以及强大的生物学活性。进一步晶体结构分析显示,设计的 TGFβRII 和 CTLA-4 的结合剂蛋白与其各自受体结合的蛋白复合物的共晶结构与设计模型高度吻合。这些设计对于下游的治疗应用应具有相当大的实用性。
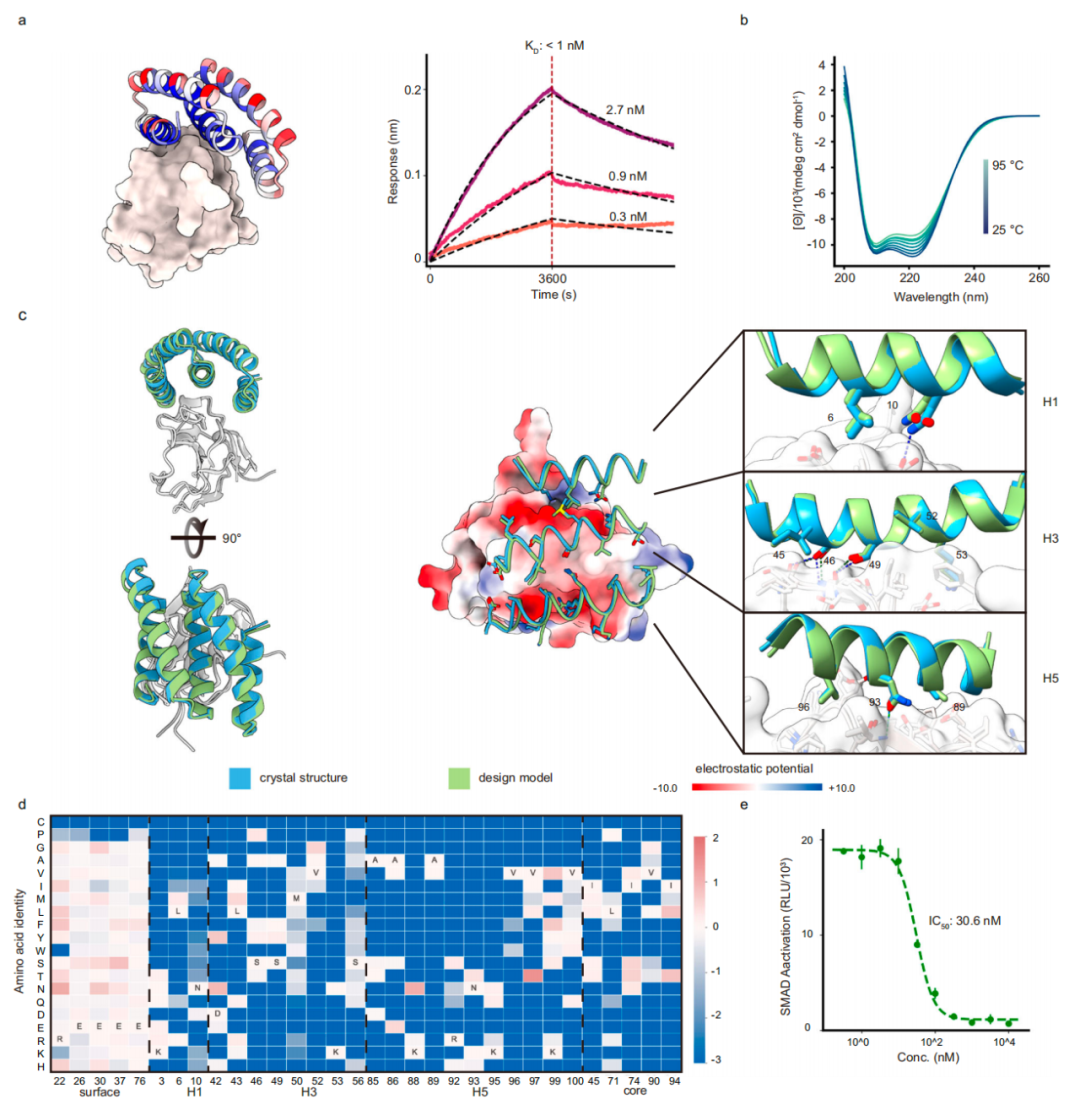
靶向结合 TGFβRII 的凹面 5HCS 结合剂
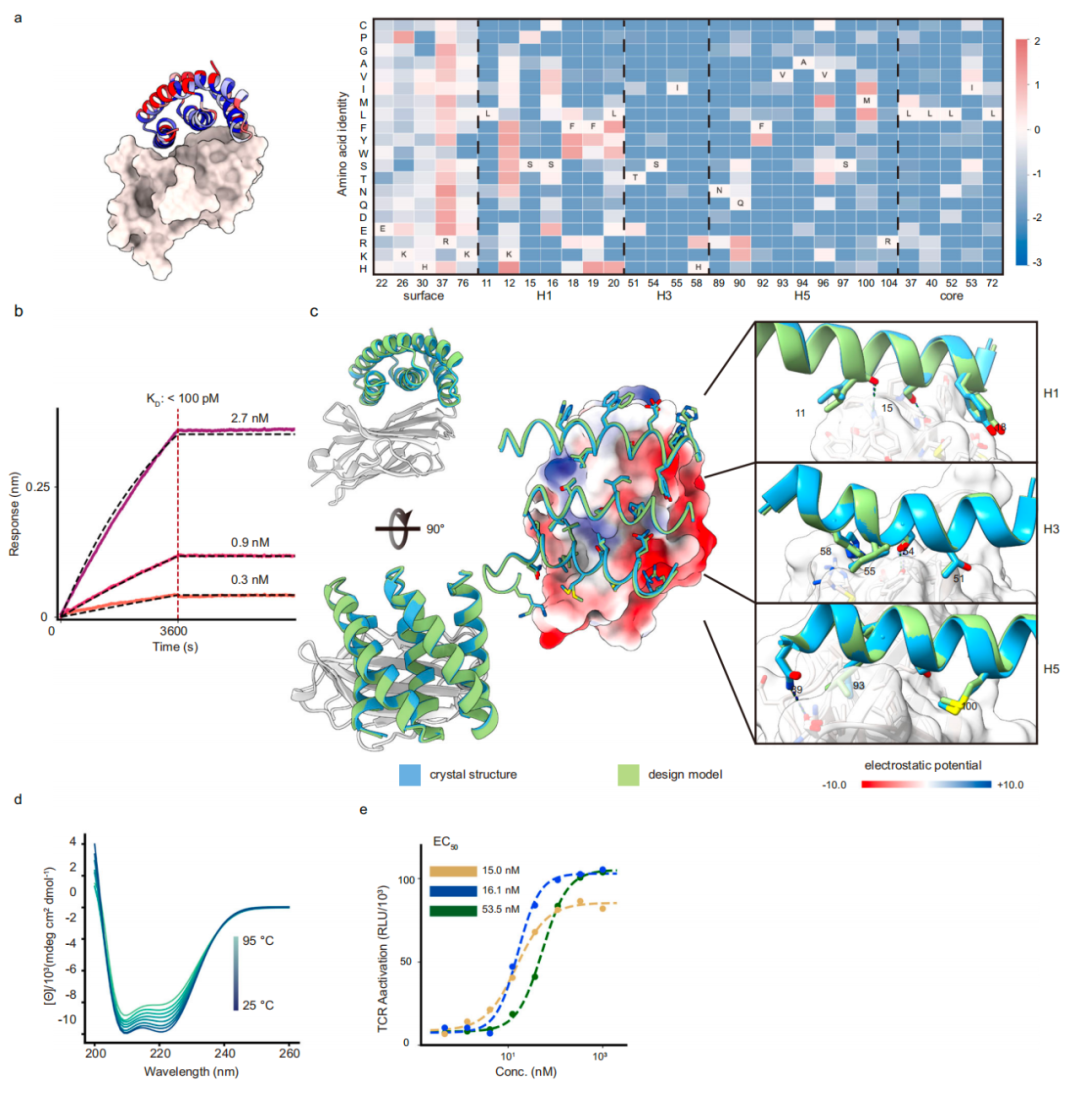
靶向结合 CTLA4 的 5HCS 结合剂
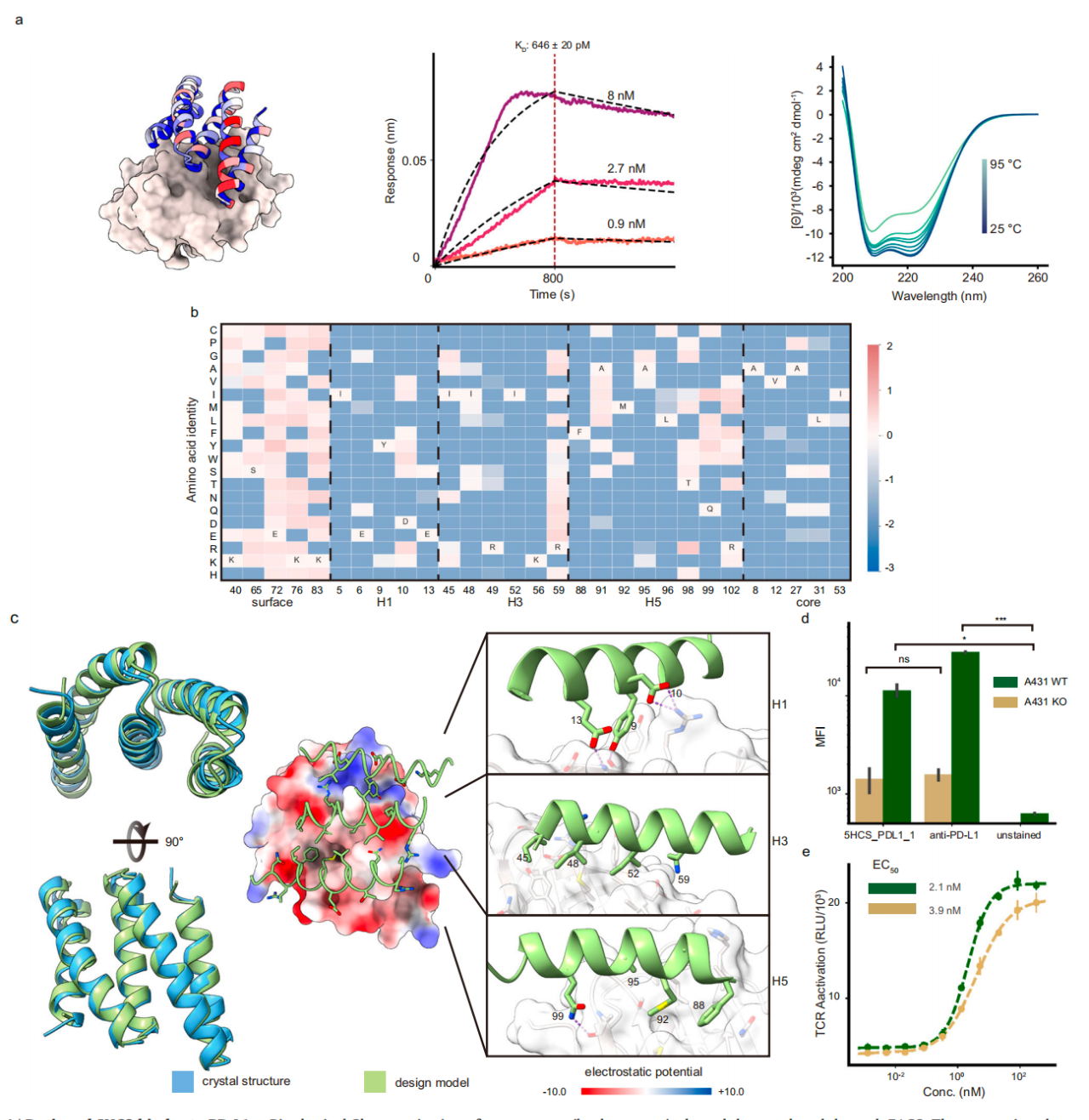
靶向结合 PD-L1 的 5HCS 结合剂
主要论文:
1. Yang, W., Sun, X., Zhang, C. & Lai, L. Discovery of novel helix binding sites at protein-protein interfaces. Comput. Struct. Biotechnol. J. 17, 1396–1403 (2019).
2. Yang, W. et al. Computational design and optimization of novel d-peptide TNFα inhibitors. FEBS Lett. 593, 1292–1302.
3. Yang, W. et al. Design of high-affinity binders to immune modulating receptors for cancer immunotherapy. Nat. Commun. 16, 2001 (2025).
4. Cao, L. et al. Design of protein-binding proteins from the target structure alone. Nature 605, 551–560 (2022).
5. Wang, J. et al. Scaffolding protein functional sites using deep learning. Science 377, 387–394 (2022).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


