Science:开发出一种创新技术,可在单分子水平上分析复杂生物过程的动态变化
来源:生物谷原创 2024-08-28 10:00
这种新开发的方法克服了单分子荧光显微镜领域的一个重大局限,而在此之前,由于一次分析一个样品较为费时费力,该领域一直受到低通量的限制。
在一项新的研究中,由乌普萨拉大学 Sebastian Deindl 教授领导的一个研究团队开发出一种开创性的方法,大大提高了在单分子水平上观察和分析复杂生物过程的能力。相关研究结果发表在2024年8月23日的Science期刊上,论文标题为“Massively parallel analysis of single-molecule dynamics on next-generation sequencing chips”。
Deindl 教授说,“通过我们的新技术,我们如今可以将单分子生物物理学扩展到基因组尺度。这一进展有望大大加深我们对与核酸相互作用的蛋白如何在健康和疾病中发挥作用的理解。”
这种方法被称为 MUSCLE(MUltiplexed Single-molecule Characterization at the Library scalE,库级多重单分子表征),它为更准确、更全面地研究生物系统打开了大门,而在生物系统中,了解分子行为的全貌至关重要。预计它将对作为序列或化学空间的功能的复杂分子动力学研究产生深远影响,使得人们能够探索生物学中以前未知的领域。
这种新开发的方法克服了单分子荧光显微镜领域的一个重大局限,而在此之前,由于一次分析一个样品较为费时费力,该领域一直受到低通量的限制。传统方法仅限于研究少量代表性样本,这可能会导致偏差,错失在大型分子文库中发现新见解的机会。
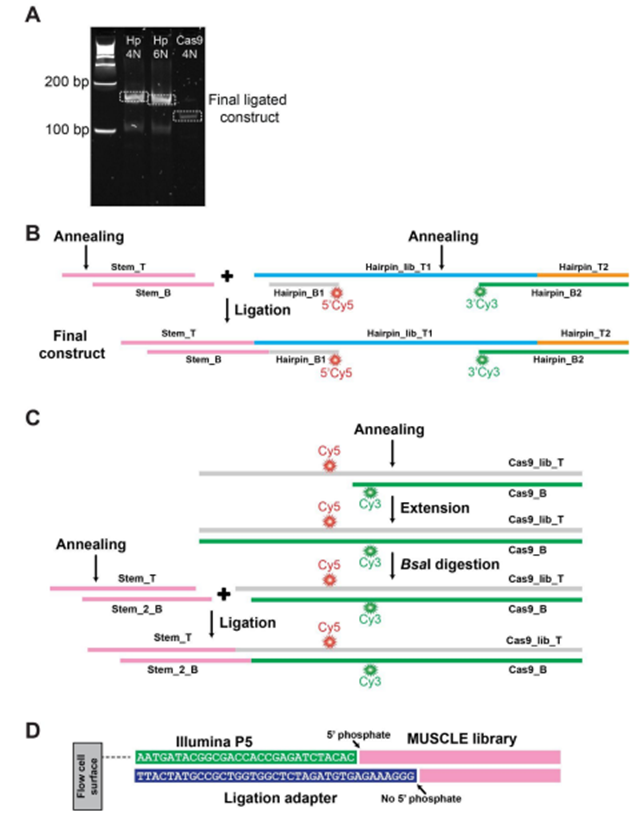
MUSCLE 将单分子荧光显微镜的机理研究与新一代测序的高通量能力相结合,解决了这一难题。这个工作流程的第一步是将荧光标记分子文库附着在称为 Illumina MiSeq 流动池的表面上。然后使用 3D 打印适配器将该流动池放置到单分子荧光显微镜上,这样就可以在多个视野中观察单个分子的实时动态。成像后,对流动池进行标准的 Illumina 测序,从先前观察到的分子中生成相同拷贝的簇。然后,根据这些簇在流动池上的位置,将其与相应的分子进行匹配。
论文共同第一作者Anton Sabantsev博士说,“对单分子成像和 Illumina 测序数据进行空间配准极具挑战性,但如今这个问题已经解决了。”
这种创新方法使得人们能够同时分析大量样本的动态变化,从而更全面地了解复杂的生物过程。Deindl 教授说,“我们的方法允许直接观察大量文库中的动态分子行为,大大提高了我们发现一般趋势、离群行为和独特动态特征的能力,否则这些特征将被隐藏起来。它将改变我们研究生物分子复杂动态的方式,并在分子生物学、遗传学和药物发现领域得到广泛应用。”
图片来自Science, 2024, doi:10.1126/science.adn5371
论文共同第一作者Javier Aguirre Rivera博士、Guanzhong Mao博士、Sabantsev博士和M. Panfilov分别为这种新技术的开发和验证做出了重要贡献。Mao博士说,“这项极具挑战性的持续多年的研究的关键在于我们成员之间出色的团队合作。每个人都带来了不同的东西,这对我们克服所面临的技术障碍至关重要。”
论文共同作者Magnus Lindell的专业知识在将下一代测序整合到 MUSCLE 工作流程中发挥了重要作用。在最初的实验中,研究团队应用该方法分析了DNA发夹动态和Cas9诱导的DNA解旋/重绕。他们的发现揭示了某些目标序列中意想不到的行为,凸显了该方法揭示新生物学观点的潜力。
由于该方法依赖于广泛使用的荧光显微镜和MiSeq仪器,而且利用3D打印技术很容易制造出必要的适配器,因此该方法对更广泛的科学界来说非常容易获得。它可用于研究与核酸相互作用的各种蛋白,以及 DNA 条形编码的蛋白、化合物或配体。(生物谷Bioon.com)
参考资料:
J. Aguirre Rivera et al. Massively parallel analysis of single-molecule dynamics on next-generation sequencing chips. Science, 2024, doi:10.1126/science.adn5371.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


