攻克新生儿的头号遗传病杀手,中国科学家厥功至伟
来源:奇点糕 2022-09-23 09:27
从SMA相关基因的发现到模式动物的构建,从SMN2基因剪接机制的研究到针对外显子7剪接药物的设计和筛选,从ASO技术的建立到MOE等关键化学修饰技术的发明
脊髓性肌萎缩症(SMA)是一种致死性的常染色体隐性遗传病。
SMA曾夺走了无数婴儿的生命,即使存活的患者也须终生接受生活护理,给患者家庭带来了巨大的精神压力和经济负担。据最新估算,我国现存患者约2-3万,并以每年2000例的速度新增。
2016年12月,美国FDA批准了第一款治疗SMA的特效药Nusinersen,为全球的SMA患者带来曙光。该药于2019年进入中国,中文名诺西那生钠,当时是名副其实的天价药。幸运的是,2021年12月它被纳入医保,实质性地造福了中国的SMA患儿。
临床上,SMA可以根据发病时间和严重程度被分为I-IV四种类型:I型是严重型,患者不能坐立,出生后6个月前发病,若无药物治疗将在2岁前因呼吸衰竭死亡;II型是中间型,发病时间为出生后6至18个月,患者能够坐下,但不能独立行走,伴随严重的并发症;III型属温和型,发病较晚,病人能实现独立行走;IV型也被称为成年型,于成年时发病,无危及生命的并发症。遗憾的是,流行病统计学调查显示,SMA病例中I型患者占大部分[1]。
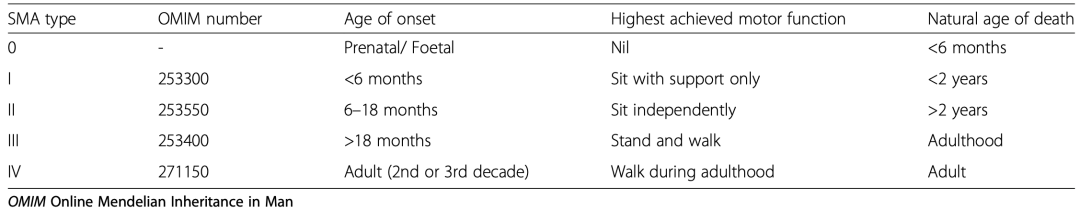
▲ SMA的分型[1]
现在我们已经知道,SMA的致病基因定位在5号染色体长臂,位于5q13区域,病因是运动神经元存活1基因(SMN1)的突变或者缺失导致无法编码SMN蛋白。SMN是一个分布广泛的持家蛋白,已知的功能是参与富U的小核糖核蛋白(U snRNP)的组装和协助轴突mRNA的运输等。
研究表明,5q13是一个几乎对称的区域,由大片段倒转复制形成,SMN1位于端粒一侧。在该区域的中心粒端存在一个SMN1的平行同源基因,称为SMN2,仅可以编码少量有功能的SMN蛋白,这不足以补偿SMN1缺失带来的SMN减少。
SMN1和SMN2极为相似,只有11处碱基不同,至关重要的差别在于它们7号外显子的第六个碱基,SMN1是C,而SMN2是T。这单个碱基的差别并不影响氨基酸的编码,但会影响7号外显子的剪接,结果是SMN1基因大部分产物都是包含外显子7的全长mRNA,而SMN2基因产物大部分是外显子7跳跃(Δ7)的mRNA。Δ7 mRNA的终止密码子发生了位移,产生的SMN是没有功能的截短蛋白,且极不稳定。因此,提高SMN2外显子7的列入水平是治疗SMA的策略之一。
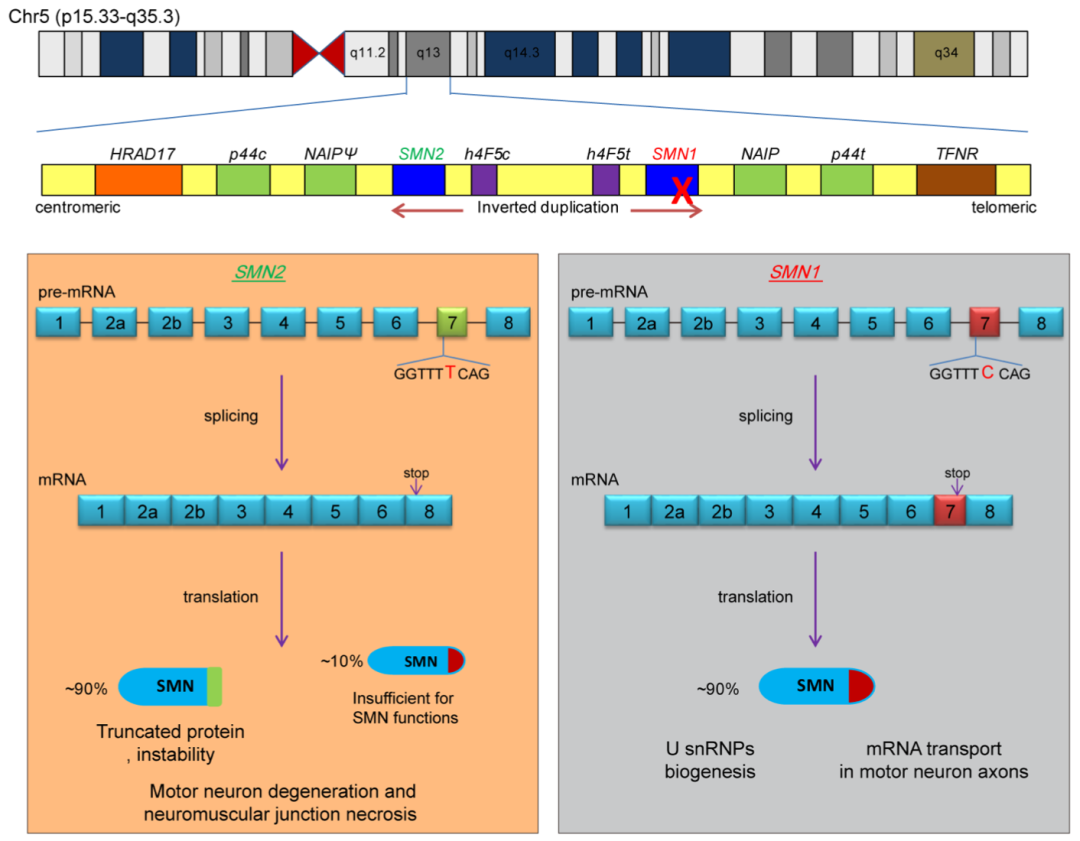
▲ SMA的发病机制
诺西那生钠正是通过纠正SMN2基因的错误剪接实现SMN蛋白水平的大幅提高。诺西那生钠的有效成分是一种反义寡核苷酸(ASO),由18个被特殊化学修饰的寡核苷酸聚合而成(磷酸二酯键被改为硫代磷酸酯键,核糖2号碳的羟基被替换为甲氧乙基(MOE))。
在作用机制方面,诺西那生钠与SMN2内含子7的第10-27个碱基精确碱基互补配对,形成空间占位效应,阻止剪接抑制因子HnRNPs的结合,从而提高外显子7的列入水平。
从1995年SMA的致病基因被明确,直至2016年被FDA批准,首款SMA药物的研发历程跨越20多年时间。
实际上,诺西那生钠的问世是由医学、遗传学、生物学等学科的重大科学发现逐步堆积起来的,是众多科学家和医生精诚合作的成果。需要指出的是,中国科学家华益民博士在诺西那生钠的研发中做出了重要贡献。本文按照时间顺序,对诺西那生钠研发过程中几项举足轻重的研究,及其对本领域的贡献做客观总结。
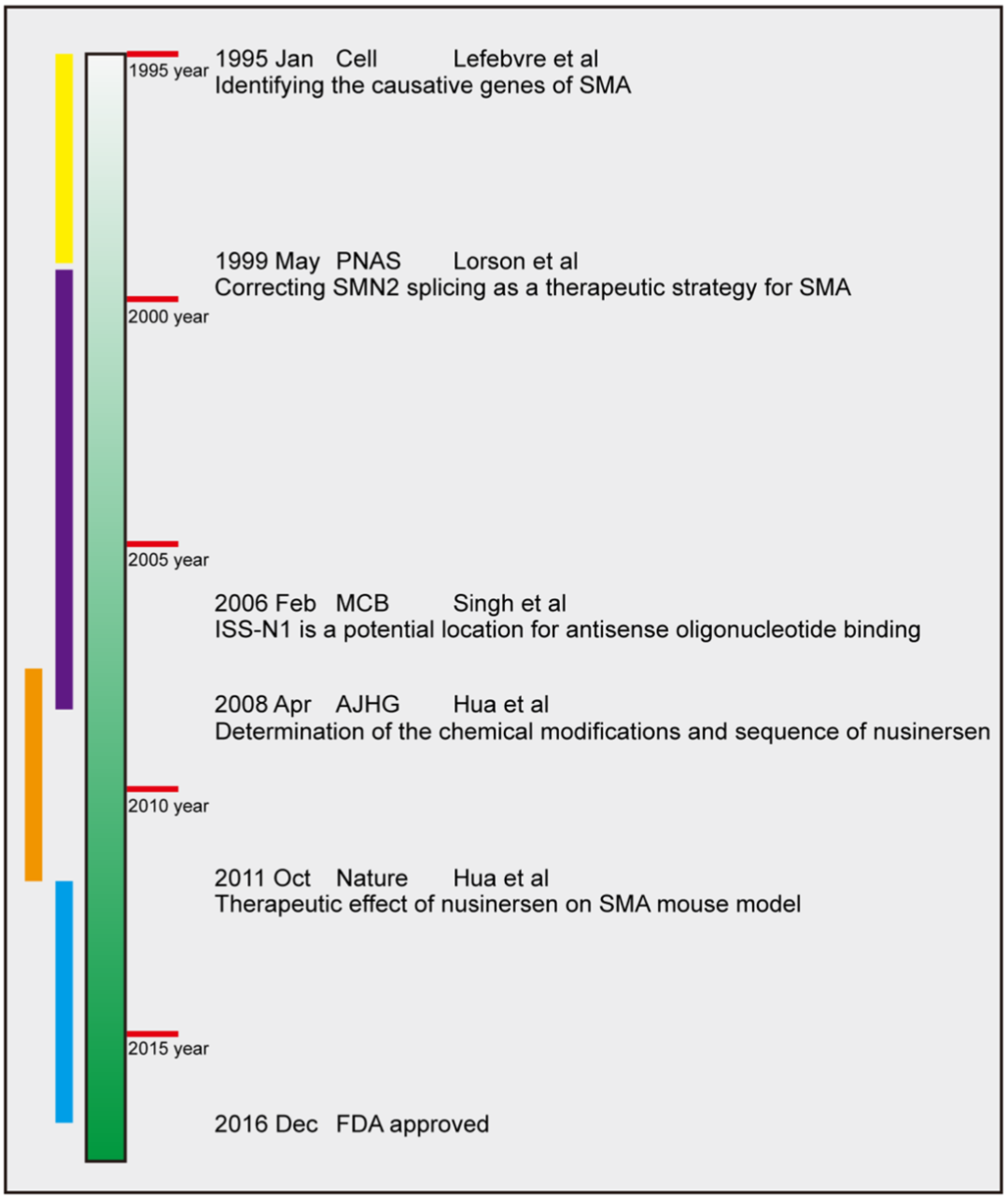
▲ 诺西那生钠研发时间线
20世纪90年代时,受限于分子克隆以及DNA测序技术,当时学界只知道SMA病人5q13区域有大规模缺失,并不能确定具体的致病基因。
1995年,法国科学家Suzie Lefebvre以及同事发现5q13区域是一个倒转复制区,该区域的端粒端包含一个称之为运动神经元存活的基因(命名为SMN基因),编码一个包含294个氨基酸的新蛋白,是SMA的决定基因[2]。
这项研究确定了SMA的致病基因,使这种致死性的疾病有了明确的研究目标,为后续的SMN功能研究和SMA病理机制奠定了分子基础。后续,该团队继续发文证实SMN蛋白定位于一种新的核结构(该结构被Qing Liu和Gideon Dreyfuss命名为Gem小体[3]),与多种RNA结合蛋白相互作用,SMA病人的SMN蛋白是缺乏的[4]。

▲ SMA病人的SMN蛋白是缺乏的[4]
尽管已经清楚位于倒转复制区的SMN1和SMN2极为相似且都可以编码SMN蛋白,但当时并不理解为什么只有SMN1突变而不是SMN2会导致SMA,更没有科学家将SMN2基因当作一个治疗SMA的靶点。
最早回答上述问题的是塔夫茨大学医学院的Elliot J. Androphy和他的博士后Christian L. Lorson。1998年,他们与波恩大学的Brunhilde Wirth合作提出SMN1和SMN2基因编码的主要产物在功能上并不相同,SMN1编码的SMN包含一个SMN自身聚集所必需的结构域,称为YG box,而SMN2编码的SMN不包含该结构域因此不能发生自身聚集[5]。
他们在后续的研究中对SMN1和SMN2基因的几个不同的碱基进行了系统评估,发现二者的剪接差异是由外显子7上第六号位碱基C到T(C6T)转换决定的,并由此推导出SMN2剪接缺陷的机制,即由于C6T转换使得SMN2的7号外显子大规模跳跃不能编码功能性SMN,这无法完全补偿SMN1突变引起的SMN蛋白减少、导致了SMA的发生。更重要的是,他们在论文中明确指出防止外显子7跳跃的策略具有治疗SMA的潜力[6]。该研究使我们更进一步了解SMA的发病机制,为诺西那生钠的研发提供了理论基础。

▲ SMN1和SMN2基因的剪接差异是由外显子7上第六号位碱基C到T(C6T)转换决定的
几乎同时,由Elliot J. Androphy实验室参与的另一篇论文也提出,C6T转换会影响二基因的剪接模式,使得SMN2的蛋白产物截短[7]。有趣的是,近20年后,华益民博士在苏州大学的实验室于2017年发现内含子6的G-44A转换,也在SMN1到SMN2剪接恶化中起着一定的作用[8]。
发病机制的阐明促进了SMA动物模型的发展。2002年,中国台湾的李鸿团队,敲除了小鼠的SMN1,并分别敲入了不同拷贝数的人源SMN2(hSMN2),成功构建了不同严重程度的转基因模式小鼠,严重型具有运动障碍而温和型则伴有远端组织坏死,然而只有4拷贝转基因模型稳定遗传下来。
后来多个实验室在此基础上构建了2拷贝转基因的严重型小鼠,寿命约10天。这些小鼠一般被称为“台湾鼠”,是使用最为广泛的SMA小鼠模型之一[9]。值得一提的是,这是历史上第一次由中国科学家向全世界提供人类疾病的动物遗传模型。
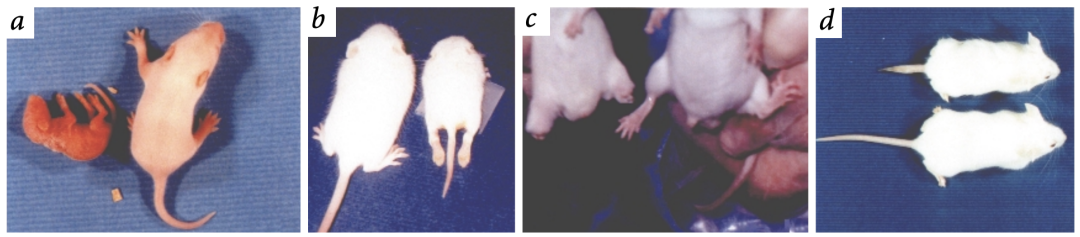
▲ SMA小鼠模型[9]
同年,俄亥俄州立大学的Arthur H.M. Burghes实验室发文报道了另一种严重型SMA小鼠模型,该鼠携带两个hSMN2 拷贝(Smn1-/-, SMN2+/+),但寿命比严重型台湾鼠更短[10]。Burghes团队后续在严重型台湾鼠的基础上插入SMN Δ7编码序列,构建了一种寿命约2周的SMA小鼠模型(Smn1-/-, SMN22/0, SMNΔ7+/+),被称为“Δ7”SMA小鼠[11]。这些动物模型的研发为后续SMA药物的体内动力学研究奠定了基础。
SMA发病机制阐明后的研究重点是如何纠正SMN2外显子7的列入,以及如何利用该策略治疗SMA。2001年李鸿实验室率先发现了能促进SMN2外显子7列入的小分子化合物丁酸钠,并在SMA小鼠中观察到病理症状的改善和寿命的延长[12]。
随后多个实验室开展了纠正SMN2外显子7剪接的小分子化合物的筛选工作。Elliot实验室的助理教授华裔科学家周建华博士等,巧妙地设计了带有绿色荧光蛋白(GFP)或荧光素酶(luciferase)标记的小基因系统。他们通过在外显子7上插入单个碱基控制开放阅读框,将Exon7的列入/跳跃信号转化为荧光或者酶活等化学信号。这是最早的SMN2剪接依赖的高通量药物筛选体系,并成功筛到了几种有效的小分子化合物[13]。
尽管早期的小分子药并没有在临床试验中取得成功,这种利用报告基因进行高通量筛选的思想,对剪接调控类药物的研发具有重要指导意义,诺西那生钠和另一种被批准的剪接调控药物Risdiplam,皆是由类似的报告系统筛选获得。
研发药物的另一种策略是使用ASO纠正SMN2的剪接。2001年,加州大学Klemens J. Hertel实验室的Sharlene R. Lim发现,作用于内含子7的3’剪接位点的ASO,可以少量促进外显子7的列入,其原理是ASO抑制了内含子7的3’剪接位点的识别,竞争性地增强了内含子6的3’剪接位点的识别[14]。
日本科学家Kazunori Imaizumi团队报道了SMN2内含子6存在一个内含子剪接沉默子,称为element 1,与element 1结合的ASO能少量增加外显子7的列入[15]。冷泉港实验室Adrian R. Krainer团队设计了肽核酸(PNA),该策略将寡核苷酸和短肽偶联,通过一段与SMN2碱基互补的寡核苷酸序列提高PNA对SMN2的靶向性,通过包含剪接激活结构域的短肽实现对剪接的调控,该策略在细胞中有效地提高了SMN2外显子7的列入[16]。
伦敦帝国理工学院的Francesco Muntoni团队采用了双功能ASO的策略(b-ASO),它包括与靶外显子互补的反义部分和负责招募SMN2外显子7剪接激活子的尾部组成,用b-ASO处理SMA病人的细胞后,可以观察到明显的SMN蛋白水平提高和Gem小体数量增多[17]。然而后续的动物试验中,b-ASO对SMA小鼠的寿命提升十分有限[18]。
一项由麻省大学医学院Ravindra N. Singh主导的研究在诺西那生钠研发历程中是不能不提的。Ravindra N. Singh曾是Elliot J. Androphy实验室在塔夫茨大学时期聘用的助理教授,跟随Androphy实验室搬至麻省大学医学院逐步建立独立实验室。
Ravindra N. Singh的博士后Nirmal K Singh发现,在SMN2内含子7的5’端存在一段对外显子7列入有强抑制作用的序列,命名为内含子剪接沉默子N1(ISS-N1)。所在单位于2005年12月正式申请ISS-N1的专利保护,并于2010年11月获专利许可,相关论文2006年发表。重要的是,靶向ISS-N1的ASO能大幅提高SMN2外显子7的列入,具有治疗潜力[19]。
Nirmal K Singh等使用的ASO几乎是诺西那生钠的雏形,其结合的位置是内含子7的7-25,不同于诺西那生钠的10-27,使用的化学修饰是2’甲氧基也不同于诺西那生钠的2’MOE。遗憾的是,他们并未进行ASO的优化工作。后续的在体试验结果表明,这种ASO在Δ7 SMA小鼠中仅能小幅增加SMN的蛋白水平,在温和型台湾鼠模型中不仅对外显子7的列入没有影响,反而会引起炎症反应[20, 21]。
以上这些ASO的早期探索均未取得预期效果,其原因在于两方面:1)缺乏系统性研究,不清楚最优的ASO结合位置是什么;2)不清楚哪种化学修饰最易于成药。
2004年6月,华益民博士在SMA基金会资助下,由麻省大学Elliot实验室加入冷泉港实验室Krainer团队,开展了针对SMN2剪接的ASO药物筛选工作。他首次采用步移法(ASO walk)大规模、系统地筛选不同长度ASO,靶点覆盖从内含子6到外显子8的整个区域,并与加州的ISIS制药公司(2015年改名Ionis)合作分析了多种ASO化学修饰的成药性,发现ASO10-27(即诺西那生钠)及其加长版本在单体经过糖基2号位MOE修饰后具有最强反义活性,在较低浓度时即能有效地纠正SMN2的剪接缺陷[22, 23]。专利于2006年6月正式申请,于2013年1月获批。
ASO10-27靶向上文提到的ISS-N1。华益民博士和Krainer教授进一步阐明了ASO10-27的作用机制,即该位置存在两个剪接抑制因子hnRNP A1/2的结合位点,ASO通过空间占位效应阻止了hnRNP A1/2的结合,从而提高了外显子7的列入水平[23]。这篇论文的发表标志着诺西那生钠的主成分ASO10-27正式确立。
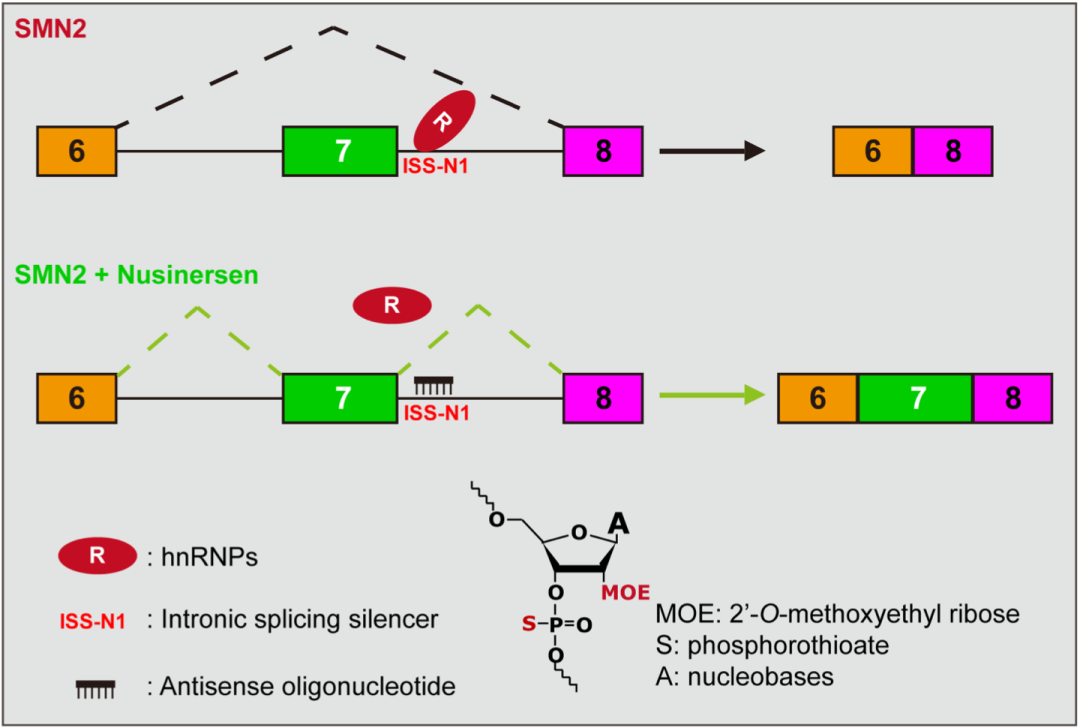
▲ 诺西那生钠的作用机制
在诺西那生钠发现期间从试管、细胞中ASO的筛选,到动物试验工作包括设计、执行等主要由华益民博士完成,ISIS负责这一项目的是C Frank Bennet,其助理包括Timothy Vickers和Brenda Baker,早期负责提供各种化学修饰的ASO,在动物试验期间提供了ASO相关的技术支持,比如ASO特异性抗体等。诺西那生钠最初专利的发明人为Brenda Baker、Adrian R. Krainer和Yimin Hua[24]。
此时,诺西那生钠研发的主题已经发生了本质转变,由“发现”转向“证实”。在哺乳类和非人灵长类动物模型中测试ASO10-27的有效性和毒性,是确定其是否能进一步进入临床试验的关键步骤。ISIS公司已经在ASO的生物分布、药代动力学以及毒理学方面积累了大量研究基础,他们发现MOE修饰的ASO可随血液循环分布于全身,在肝脏等外周组织吸收较好,也证实ASO具有良好的生物安全性[25-27]。
华益民博士2008年在冷泉港实验室基于台湾4拷贝转基因小鼠构建了2拷贝的严重型模型,每窝新生小鼠中有50%的SMA小鼠,加速了动物试验的进程。华博士同时制备了抗人SMN蛋白的特异性抗体,能在小鼠组织中识别人SMN2转基因来源的SMN蛋白,为在模式小鼠蛋白水平精确检测ASO的效果奠定了基础[21]。
诺西那生钠的发明不是一帆风顺的,2009年Gordon J. Lutz实验室在SMA基金会支持下,发现ASO注射进脑室虽然能提高神经组织SMN蛋白水平,但并不能延长模式小鼠的寿命[20]。由于SMA是脊髓运动神经元退变疾病,直接给药到脑脊液是一种合理的治疗策略。华益民博士在2008年底已获得严重型台湾鼠模型,通过侧脑室在新生小鼠给药后,也没有观察到动物病理症状的显著改善,寿命仅延长数天,当时心情非常沮丧。
的确,从2008年到2009上半年SMA学术界被ASO技术无效的氛围笼罩着。突破发生在一次偶然的实验疏漏,华博士有两只小鼠在脑室注射时药物部分进入了皮下,小鼠没有被丢弃,结果这两只小鼠存活了2个月以上,华博士在2009年冷泉港实验室内部会议上报告了这一激动人心的结果,著名科学家DNA双螺旋发明人之一James D Watson听了报告后当场啧啧称奇并予以鼓励。
这一发现使华博士意识到,严重型模式小鼠可能存在严重的外周组织缺陷,这一点可能与人类SMA患者有差异。结果改用简单的皮下注射后,小鼠中位寿命从10天延长到248天,部分小鼠能活2年以上,组织缺陷获得修复,运动能力显著改善[28]。这一轰动性的成果发表在2011年《自然》杂志上,不仅对于SMA的治疗,这也是ASO技术迈向成功的一个标志性事件。

▲ 论文首页截图
非人灵长类动物缺乏SMN2,不能通过剪接测试或蛋白表达检验ASO的治疗效果。向成年或幼年食蟹猴的中枢神经系统注射高剂量的ASO10-27(1-7mg),可以观察到ASO在脊髓的所有区域都有分布,并不会引起死亡和其他不良反应[29]。这些数据表明ASO10-27已具备成为SMA药物的关键品质,为诺西那生钠进入临床试验建立了基础。
2011年,在Ionis Pharmaceuticals的支持下,ASO10-27更名为ISIS-SMNRx,在全球多家医院开展临床试验,随后改名为Ionis-SMNRx及诺西那生钠。Biogen(渤健)在临床二期时加入了诺西那生钠的研发,并购买了诺西那生钠的相关专利。
对于SMA患者,诺西那生钠表现出良好的耐受性,广泛的脑脊液分布和>6个月的半衰期,其安全性和临床疗效也得到了系统的评估。随机、双盲假对照的三期临床试验结果显示,接受治疗的患儿比对照组更有可能存活且运动功能显著改善。由于治疗效果优异,临床试验提前终止,FDA于2016年12月加速批准了该药物入市,渤健给予商业名Spinraza。
▲ FDA批准上市页截图
诺西那生钠的给药方案为12mg(5ml)剂量当量,在前2个月内进行4次鞘内注射,然后每4个月重新补注一次。药物治疗2年后,患有Ⅰ型或Ⅱ型SMA的婴儿表现出运动能力的显著改善。值得注意的是,症状前治疗的疗效远好于症状后治疗。因此,加强新生儿筛查和基因诊断对于现阶段SMA治疗至关重要。起初,诺西那生钠整个疗程的治疗费用约为200万美元,这让许多SMA患者望而却步。幸运的是,越来越多的国家正在将其纳入医疗保险,这将大大增加诺西那生钠的受益群体。
综上所述,从SMA相关基因的发现到模式动物的构建,从SMN2基因剪接机制的研究到针对外显子7剪接药物的设计和筛选,从ASO技术的建立到MOE等关键化学修饰技术的发明,多个科研团队和企业在推动国际第一款核酸重磅药诺西那生钠研发进程中作出了贡献。
诺西那生钠的发明是多个国家和地区科学家共同努力的结果,包括美国、欧洲、中国台湾,其中我国科学家华益民博士在留美期间的工作发挥了关键的作用。同时也不能忘记多个基金会和私人团体在推广SMA知识和资金支持中作出的贡献,包括SMA基金会、SMA家庭/救治SMA、NIH和MDA。
参考文献
1.Verhaart IEC, Robertson A, Wilson IJ, Aartsma-Rus A, Cameron S, Jones CC, Cook SF, Lochmuller H: Prevalence, incidence and carrier frequency of 5q-linked spinal muscular atrophy - a literature review. Orphanet J Rare Dis 2017, 12(1):124.
2.Lefebvre S, Burglen L, Reboullet S, Clermont O, Burlet P, Viollet L, Benichou B, Cruaud C, Millasseau P, Zeviani M et al: Identification and characterization of a spinal muscular atrophy-determining gene. Cell 1995, 80(1):155-165.
3.Liu Q, Dreyfuss G: A novel nuclear structure containing the survival of motor neurons protein. EMBO J 1996, 15(14):3555-3565.
4.Lefebvre S, Burlet P, Liu Q, Bertrandy S, Clermont O, Munnich A, Dreyfuss G, Melki J: Correlation between severity and SMN protein level in spinal muscular atrophy. Nat Genet 1997, 16(3):265-269.
5.Lorson CL, Strasswimmer J, Yao JM, Baleja JD, Hahnen E, Wirth B, Le T, Burghes AH, Androphy EJ: SMN oligomerization defect correlates with spinal muscular atrophy severity. Nat Genet 1998, 19(1):63-66.
6.Lorson CL, Hahnen E, Androphy EJ, Wirth B: A single nucleotide in the SMN gene regulates splicing and is responsible for spinal muscular atrophy. Proc Natl Acad Sci U S A 1999, 96(11):6307-6311.
7.Monani UR, Lorson CL, Parsons DW, Prior TW, Androphy EJ, Burghes AH, McPherson JD: A single nucleotide difference that alters splicing patterns distinguishes the SMA gene SMN1 from the copy gene SMN2. Hum Mol Genet 1999, 8(7):1177-1183.
8.Wu X, Wang SH, Sun J, Krainer AR, Hua Y, Prior TW: A-44G transition in SMN2 intron 6 protects patients with spinal muscular atrophy. Hum Mol Genet 2017, 26(14):2768-2780.
9.Hsieh-Li HM, Chang JG, Jong YJ, Wu MH, Wang NM, Tsai CH, Li H: A mouse model for spinal muscular atrophy. Nat Genet 2000, 24(1):66-70.
10.Monani UR, Sendtner M, Coovert DD, Parsons DW, Andreassi C, Le TT, Jablonka S, Schrank B, Rossoll W, Prior TW et al: The human centromeric survival motor neuron gene (SMN2) rescues embryonic lethality in Smn(-/-) mice and results in a mouse with spinal muscular atrophy. Hum Mol Genet 2000, 9(3):333-339.
11.Le TT, Pham LT, Butchbach ME, Zhang HL, Monani UR, Coovert DD, Gavrilina TO, Xing L, Bassell GJ, Burghes AH: SMNDelta7, the major product of the centromeric survival motor neuron (SMN2) gene, extends survival in mice with spinal muscular atrophy and associates with full-length SMN. Hum Mol Genet 2005, 14(6):845-857.
12.Chang JG, Hsieh-Li HM, Jong YJ, Wang NM, Tsai CH, Li H: Treatment of spinal muscular atrophy by sodium butyrate. Proc Natl Acad Sci U S A 2001, 98(17):9808-9813.
13.Zhang ML, Lorson CL, Androphy EJ, Zhou J: An in vivo reporter system for measuring increased inclusion of exon 7 in SMN2 mRNA: potential therapy of SMA. Gene Ther 2001, 8(20):1532-1538.
14.Lim SR, Hertel KJ: Modulation of survival motor neuron pre-mRNA splicing by inhibition of alternative 3' splice site pairing. J Biol Chem 2001, 276(48):45476-45483.
15.Miyajima H, Miyaso H, Okumura M, Kurisu J, Imaizumi K: Identification of a cis-acting element for the regulation of SMN exon 7 splicing. J Biol Chem 2002, 277(26):23271-23277.
16.Cartegni L, Krainer AR: Correction of disease-associated exon skipping by synthetic exon-specific activators. Nat Struct Biol 2003, 10(2):120-125.
17.Skordis LA, Dunckley MG, Yue B, Eperon IC, Muntoni F: Bifunctional antisense oligonucleotides provide a trans-acting splicing enhancer that stimulates SMN2 gene expression in patient fibroblasts. Proc Natl Acad Sci U S A 2003, 100(7):4114-4119.
18.Baughan TD, Dickson A, Osman EY, Lorson CL: Delivery of bifunctional RNAs that target an intronic repressor and increase SMN levels in an animal model of spinal muscular atrophy. Hum Mol Genet 2009, 18(9):1600-1611.
19.Singh NK, Singh NN, Androphy EJ, Singh RN: Splicing of a critical exon of human Survival Motor Neuron is regulated by a unique silencer element located in the last intron. Mol Cell Biol 2006, 26(4):1333-1346.
20.Williams JH, Schray RC, Patterson CA, Ayitey SO, Tallent MK, Lutz GJ: Oligonucleotide-mediated survival of motor neuron protein expression in CNS improves phenotype in a mouse model of spinal muscular atrophy. J Neurosci 2009, 29(24):7633-7638.
21.Hua Y, Sahashi K, Hung G, Rigo F, Passini MA, Bennett CF, Krainer AR: Antisense correction of SMN2 splicing in the CNS rescues necrosis in a type III SMA mouse model. Genes Dev 2010, 24(15):1634-1644.
22.Hua Y, Vickers TA, Baker BF, Bennett CF, Krainer AR: Enhancement of SMN2 exon 7 inclusion by antisense oligonucleotides targeting the exon. PLoS Biol 2007, 5(4):e73.
23.Hua Y, Vickers TA, Okunola HL, Bennett CF, Krainer AR: Antisense masking of an hnRNP A1/A2 intronic splicing silencer corrects SMN2 splicing in transgenic mice. Am J Hum Genet 2008, 82(4):834-848.
24.Baker BF, Krainer AR, Hua Y: Compositions and methods for modulation of SMN2 splicing. PCT/US2006/024469.
25.Geary RS, Yu RZ, Watanabe T, Henry SP, Hardee GE, Chappell A, Matson J, Sasmor H, Cummins L, Levin AA: Pharmacokinetics of a tumor necrosis factor-alpha phosphorothioate 2'-O-(2-methoxyethyl) modified antisense oligonucleotide: comparison across species. Drug Metab Dispos 2003, 31(11):1419-1428.
26.Geary RS, Yu RZ, Levin AA: Pharmacokinetics of phosphorothioate antisense oligodeoxynucleotides. Curr Opin Investig Drugs 2001, 2(4):562-573.
27.Butler M, Crooke RM, Graham MJ, Lemonidis KM, Lougheed M, Murray SF, Witchell D, Steinbrecher U, Bennett CF: Phosphorothioate oligodeoxynucleotides distribute similarly in class A scavenger receptor knockout and wild-type mice. J Pharmacol Exp Ther 2000, 292(2):489-496.
28.Hua Y, Sahashi K, Rigo F, Hung G, Horev G, Bennett CF, Krainer AR: Peripheral SMN restoration is essential for long-term rescue of a severe spinal muscular atrophy mouse model. Nature 2011, 478(7367):123-126.
29.Passini MA, Bu J, Richards AM, Kinnecom C, Sardi SP, Stanek LM, Hua Y, Rigo F, Matson J, Hung G et al: Antisense oligonucleotides delivered to the mouse CNS ameliorate symptoms of severe spinal muscular atrophy. Sci Transl Med 2011, 3(72):72ra18.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


