研究揭示A-to-I RNA编辑的趋同适应性演化
来源:北京大学 2020-12-25 12:22
北京大学生命科学学院陆剑研究员课题组与特拉维夫大学Eli Eisenberg教授合作在iScience发表了题为“A-to-I RNA editing in honeybees shows signals of adaptation and convergent evolution”的论文。该工作以蜜蜂为研究对象,解析了蜜蜂A-to-
北京大学生命科学学院陆剑研究员课题组与特拉维夫大学Eli Eisenberg教授合作在iScience发表了题为“A-to-I RNA editing in honeybees shows signals of adaptation and convergent evolution”的论文。该工作以蜜蜂为研究对象,解析了蜜蜂A-to-I RNA编辑的适应性演化,以及蜜蜂和果蝇里RNA编辑的趋同适应现象。
由ADAR(adenosine deaminase acting on RNA)蛋白介导的腺嘌呤到次黄嘌呤(A-to-I)的RNA编辑是后生动物中广泛存在的转录后修饰。由于I会被识别为G,因此A-to-I RNA编辑在不改变基因组序列的情况下,时空特异性地增加了转录组和蛋白组的多样性。陆剑课题组之前的工作已经报道了果蝇中存在大量改变氨基酸的非同义RNA编辑位点(Nonsyn),这些编辑位点呈现出适应性信号,受到正向自然选择(Duanet al., 2017, PLoS Genetics),并且这些成簇分布的、具有适应性的非同义RNA编辑事件倾向于连锁在相同mRNA分子上,也就是同时被编辑(Duanet al., 2018, Molecular Biology and Evolution)。
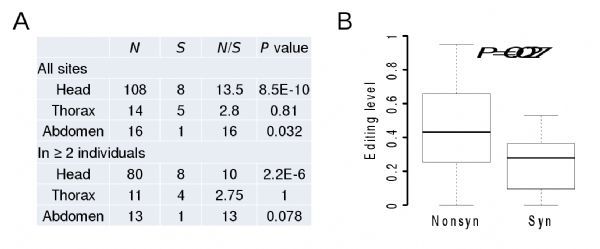
为了在更大的进化尺度上探究A-to-I RNA编辑是否具有适应性,该课题组以西方蜜蜂(Apis mellifera)为材料进行RNA编辑组研究。这项研究充分利用了雄蜂(drone)是单倍体这一优势,通过对四个雄蜂个体的基因组测序和不同组织的转录组测序,系统地鉴定出407个高可信度的A-to-I编辑位点。在407个高可信度编辑位点中,111个是改变氨基酸的非同义编辑位点(Nonsyn,改变蛋白质序列),而只有九个是同义编辑位点(Syn,不改变蛋白质序列)。编辑位点在头里面丰度最高,包括了108个Nonsyn与八个Syn位点,这与头中Adar基因表达量高是相符合的。与Syn位点相比,Nonsyn位点呈现出非常强的适应性演化信号:(1)Nonsyn在编码区中出现的频率显着高于中性(随机)情况下的预期值(图1A);(2)Nonsyn比Syn位点有更高的编辑水平(图1B)。该现象与之前在果蝇中观测到的结果非常一致(Duanet al., 2017, PLoS Genetics)。
研究人员探究了西方蜜蜂和黑腹果蝇的保守编辑位点,发现保守的位点较少,在编码区中只有四个Nonsyn与1个Syn位点在蜜蜂和果蝇中保守。虽然位置不保守,但很多编辑位点却发生在相同的基因上。例如,基因Adar上有一个改变氨基酸的“自身编辑位点”,发生编辑之后会对ADAR蛋白的活性产生影响,对整体RNA编辑活性形成反馈调节。而在果蝇和蜜蜂两个进化枝上独立演化出了这个具有调控作用的自身编辑位点(图2A)。结合之前报道的大黄蜂(Bombus terrestris)里的编辑位点(Porathet al., 2019, Nature Communications),题目进一步统计发现,在黑腹果蝇、西方蜜蜂、大黄蜂三个物种编码区发生编辑的基因中,同源基因的交集数量显着高于随机抽取相同数量基因所得的同源基因数量(图2B、2C),在两两物种之间或者三个物种之间都是一致的比较结果。因此,可以得出的结论是,在果蝇和蜜蜂两个相距较远的进化枝上,同源基因在编码区中独立演化出了编辑位点(图2D)。虽然编辑位点的位置不保守,但在各自的进化枝上都展现出适应性信号。这种趋同演化现象进一步支持了RNA编辑的适应性假说。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。