Mol Cell:中科院高彩霞课题组开发出具有高特异性和高精度的胞嘧啶碱基编辑器
来源:本站原创 2020-08-03 12:00
2020年8月3日讯/生物谷BIOON/---碱基编辑器可以在不引起双链DNA断裂的情况下,在基因组DNA中产生高效的定向点突变,在人类疾病的基因治疗和作物植物的性状改良方面有很大前景。中国科学院遗传与发育生物学研究所高彩霞(Gao Caixia)教授课题组一直在研发植物碱基编辑技术。他们发现胞嘧啶碱基编辑器(cytosine base editor, CB
2020年8月3日讯/生物谷BIOON/---碱基编辑器可以在不引起双链DNA断裂的情况下,在基因组DNA中产生高效的定向点突变,在人类疾病的基因治疗和作物植物的性状改良方面有很大的应用前景。
中国科学院遗传与发育生物学研究所高彩霞(Gao Caixia)教授课题组一直在研发植物碱基编辑技术。他们发现胞嘧啶碱基编辑器(cytosine base editor, CBE)可诱导水稻出现意想不到的全基因组脱靶突变,这就促进全球基因组编辑界对此进行改进。
在一项新的研究中,高彩霞教授课题组基于截短的人APOBEC3胞嘧啶脱氨酶(A3Bctd)构建出两种新的CBE,并开发出一种高通量检测方法,用于评估植物CBE中不依赖于单向导RNA(sgRNA)的脱氨变化。相关研究结果于2020年7月27日在线发表在Molecular Cell期刊上,论文标题为“Rationally Designed APOBEC3B Cytosine Base Editors with Improved Specificity”。
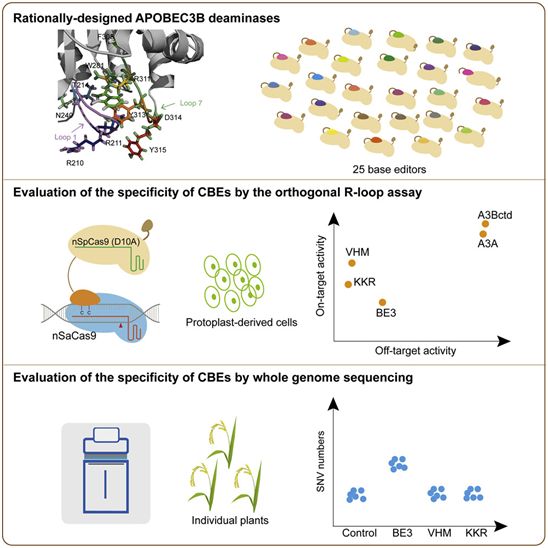
他们首先开发了一种快速、高通量且廉价的方法---nSaCas9介导的正交R环测定法---来评估植物中的CBE。在这种测定法中,正交CRISPR系统nSaCas9被用于在植物细胞中产生单链DNA(ssDNA)区域,作为不依赖于sgRNA的脱氨变化的靶标。为了评估nSaCas9介导的正交R环测定,他们将它与全基因组测序(WGS)测定进行了比较。
一致性的结果表明,nSaCas9介导的正交R-loop测定法为评估CBE的不依赖于sgRNA的脱靶活性提供了一种合理、快速和高通量的方法。
他们随后通过合理设计构建出16个A3Bctd脱氨酶变体,并评估了它们的在靶效率(on-target efficiency)和不依赖于sgRNA的脱靶活性。他们利用nSaCas9介导的正交R环测定法对这些A3Bctd-BE3变体进行了测试,并选择了7个与高效的在靶编辑活性和下降的脱靶活性相关的突变。之后,他们将这些突变进行组合,产生了9个新的具有双氨基酸或三氨基酸替换的A3Bctd-BE3变体。
通过这种方式,他们得到了两个新的CBE变体:A3Bctd-VHM-BE3和A3Bctd-KKR-BE3,这两个变体表现出高效的在靶活性和明显下降的不依赖于sgRNA的脱靶活性。
此外,这两种新的CBE变体在它们的靶位点上的编辑表现得更为精确,主要产生单个和两个胞嘧啶(C)编辑。他们还通过全基因组测序验证了A3Bctd-VHM-BE3和A3Bctd-KKR-BE3的高特异性。(生物谷 Bioon.com)
参考资料:
1.Shuai Jin et al. Rationally Designed APOBEC3B Cytosine Base Editors with Improved Specificity. Molecular Cell, 2020, doi:10.1016/j.molcel.2020.07.005.
2.Researchers develop new cytosine base editors with high specificity and precision
https://phys.org/news/2020-07-cytosine-base-editors-high-specificity.html
中国科学院遗传与发育生物学研究所高彩霞(Gao Caixia)教授课题组一直在研发植物碱基编辑技术。他们发现胞嘧啶碱基编辑器(cytosine base editor, CBE)可诱导水稻出现意想不到的全基因组脱靶突变,这就促进全球基因组编辑界对此进行改进。
在一项新的研究中,高彩霞教授课题组基于截短的人APOBEC3胞嘧啶脱氨酶(A3Bctd)构建出两种新的CBE,并开发出一种高通量检测方法,用于评估植物CBE中不依赖于单向导RNA(sgRNA)的脱氨变化。相关研究结果于2020年7月27日在线发表在Molecular Cell期刊上,论文标题为“Rationally Designed APOBEC3B Cytosine Base Editors with Improved Specificity”。

他们首先开发了一种快速、高通量且廉价的方法---nSaCas9介导的正交R环测定法---来评估植物中的CBE。在这种测定法中,正交CRISPR系统nSaCas9被用于在植物细胞中产生单链DNA(ssDNA)区域,作为不依赖于sgRNA的脱氨变化的靶标。为了评估nSaCas9介导的正交R环测定,他们将它与全基因组测序(WGS)测定进行了比较。
一致性的结果表明,nSaCas9介导的正交R-loop测定法为评估CBE的不依赖于sgRNA的脱靶活性提供了一种合理、快速和高通量的方法。
他们随后通过合理设计构建出16个A3Bctd脱氨酶变体,并评估了它们的在靶效率(on-target efficiency)和不依赖于sgRNA的脱靶活性。他们利用nSaCas9介导的正交R环测定法对这些A3Bctd-BE3变体进行了测试,并选择了7个与高效的在靶编辑活性和下降的脱靶活性相关的突变。之后,他们将这些突变进行组合,产生了9个新的具有双氨基酸或三氨基酸替换的A3Bctd-BE3变体。
通过这种方式,他们得到了两个新的CBE变体:A3Bctd-VHM-BE3和A3Bctd-KKR-BE3,这两个变体表现出高效的在靶活性和明显下降的不依赖于sgRNA的脱靶活性。
此外,这两种新的CBE变体在它们的靶位点上的编辑表现得更为精确,主要产生单个和两个胞嘧啶(C)编辑。他们还通过全基因组测序验证了A3Bctd-VHM-BE3和A3Bctd-KKR-BE3的高特异性。(生物谷 Bioon.com)
参考资料:
1.Shuai Jin et al. Rationally Designed APOBEC3B Cytosine Base Editors with Improved Specificity. Molecular Cell, 2020, doi:10.1016/j.molcel.2020.07.005.
2.Researchers develop new cytosine base editors with high specificity and precision
https://phys.org/news/2020-07-cytosine-base-editors-high-specificity.html
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


