JCIM:计算提升蛋白质-蛋白质相互作用的预测精度
来源:生物谷 2019-10-10 10:12
蛋白质-蛋白质相互作用和识别在生物学过程中有着非常重要的作用。尽管结构生物学已经取得了较大的进展,但直接采用实验方法确定蛋白质-蛋白质复合物结构仍然非常困难。分子对接技术是预测蛋白质-蛋白质复合物结构的有效方法。蛋白质-小分子之间的相互作用一般蛋白质受体有结合口袋,相互作用区域比较明确,而蛋白质-蛋白质相互作用则有可能发生在两个蛋白质表面上任何区域。因此,蛋白质-蛋白质相互作用的预测,需要对蛋白质
蛋白质-蛋白质相互作用和识别在生物学过程中有着非常重要的作用。尽管结构生物学已经取得了较大的进展,但直接采用实验方法确定蛋白质-蛋白质复合物结构仍然非常困难。分子对接技术是预测蛋白质-蛋白质复合物结构的有效方法。蛋白质-小分子之间的相互作用一般蛋白质受体有结合口袋,相互作用区域比较明确,而蛋白质-蛋白质相互作用则有可能发生在两个蛋白质表面上任何区域。因此,蛋白质-蛋白质相互作用的预测,需要对蛋白质表面进行全局且充分的搜索,然而全局搜索计算非常耗时。快速傅里叶变换方法(fast Fourier transform, FFT)是蛋白质-蛋白质对接中应用最广泛的搜索方法,因为该方法可以对蛋白质表面的互补情况进行充分且高效的全局搜索。为了推进蛋白质-蛋白质复合物结构预测的发展,欧洲生物信息学中心从2001年开始举办CAPRI预测竞赛(Critical Assessment of PRedicted Interactions),采用尚未公开发表的蛋白质-蛋白质复合物结构对不同课题组的预测方法进行测试。
尽管CAPRI预测竞赛已经取得了较大进展,但基于FFT全局搜索的蛋白质-蛋白质分子对接方法仍然存在一些明显的局限性。首先,任何原子对打分函数,如原子对接触势能,必须定义成相关函数的形式才可以采用FFT方法计算。因此,更精确的距离相关(distance-dependent)原子对打分函数,则难以表示成相关函数形式,从而也限制了精确打分函数在FFT方法中的应用。其次,指定结合位点的分子对接对于提高蛋白质-蛋白质分子对接的准确性非常重要,但位点约束条件也不容易写成相关函数形式。因此,很少有基于FFT方法的蛋白质-蛋白质分子对接程序能够在全局搜索中加入残基位点约束条件。第三,FFT全局搜索中使用的打分函数,一般基于晶体结构进行训练,这些打分函数对于蛋白质结构的微小变化非常敏感。因此,很多FFT对接方法在结合态(Bound)表现较好,而对于更接近真实情况的自由态(UnBound)对接,则因为蛋白质结构柔性变化而无法预测得到正确复合物构象。因此,有必要改进传统的FFT全局搜索策略,发展能够同时实现全局搜索、精确打分和位点约束的蛋白质-蛋白质对接方法。
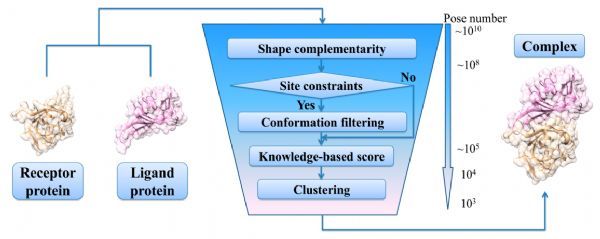
图1. CoDockPP程序流程图
基于FFT的蛋白质-蛋白质对接方法会产生~1010个复合物构象,但传统的方法仅保留了103-104个构象进行后续精确评分。因此,大量正确的复合物构象在初始阶段就已经被排除掉了。常珊课题组建议了一种新的基于FFT的蛋白质-蛋白质对接方法CoDockPP(流程如图1),服务器的网址是http://codockpp.schanglab.org.cn。该方法充分运用了FFT方法全局搜索的优点,选择了几何匹配较好的对接复合物构象。搜索初期保留了任何有表面接触的复合物构象,大约108个构象到下一步。这样可以确保正确的复合物构象进入后续的精确评分。同时,复合物构象被转化到笛卡尔坐标空间。为了加速计算,应用位点约束信息和快速查表的原子对打分函数,在保证一定精度的前提下逐渐减低候选复合物构象数量(~105)。最后,候选复合物构象进一步通过精确的距离相关原子对打分函数进行排序。CoDockPP程序在benchmark 5.0上进行测试,与ZDOCK程序进行全局分子对接的比较,与RosettaDock程序进行指定位点分子对接的比较。测试比较发现,CoDockPP程序均能显着改进了成功率和准确预测的数量。特别是在构象变化比较大的困难(difficult)体系上,CoDockPP程序仍然能够在Top10中准确预测出近天然复合物构象(图2)。论文发表在2019年第8期的Journal of Chemical Information and Modeling杂志上。
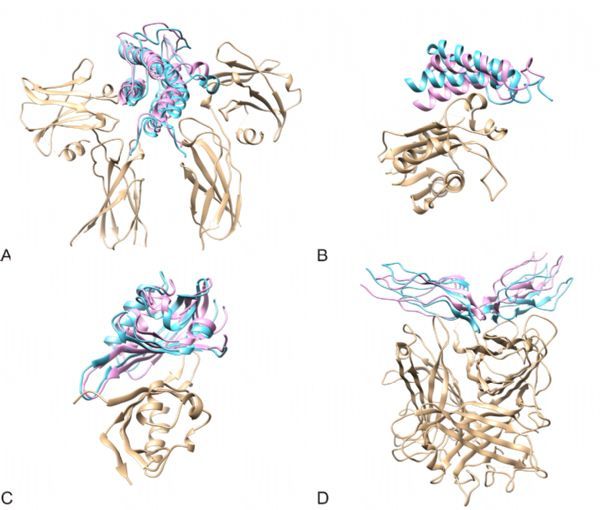
图2. CoDockPP程序在构象变化比较大的困难(difficult)体系上(PDB依次为1EER, 2IDO, 3F1P和3L89)表现较好。黄色和蓝色表示复合物晶体结构中的受体蛋白质和配体蛋白质。粉红色表示CoDockPP预测的Top10中最佳的配体蛋白质结构。
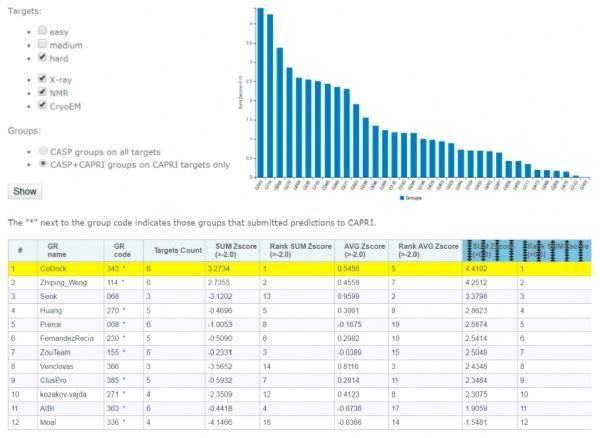
为了推动蛋白质多聚体复合物结构的预测,从2014年开始,CAPRI组委会与著名的蛋白质结构预测CASP(Critical Assessment of protein Structure Prediction)组委会共同组织了联合竞赛。在最新的CASP-CAPRI联合竞赛中,常珊课题组的CoDock团队获得CASP+CAPRI整体排名第8,CAPRI参赛组排名第4的好成绩。特别是在困难(Hard)类型预测中CoDock表现突出,排名第1(图3,排名截图自CASP评估官网http://predictioncenter.org/casp13/zscores_multimer.cgi)。
2019年4月,CAPRI组委会在英国剑桥大学举办了第七届CAPRI评估会,此次评估会上谷歌公司的DeepMind团队介绍了AlphaFold程序在蛋白质单体结构预测中表现。江苏理工学院常珊课题组也受邀参加了此次评估会,是特邀口头报告中唯一的中国团队,课题组主要介绍CoDock团队在CAPRI预测竞赛中的表现,并获得了CAPRI组委会的肯定。
图3. CASP-CAPRI联合竞赛中,困难(Hard)类型预测CoDock团队排名第1
发表论文和链接:
[1] Kong R, Wang F, Zhang J, Xu X J, Chang S. CoDockPP: a multistage approach for global and site-specific protein-protein docking. Journal of Chemical Information and Modeling. 2019, 59(8): 3556-3564.
https://pubs.acs.org/doi/10.1021/acs.jcim.9b00445
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->