2019年3月CRISPR/Cas研究进展
来源:本站原创 2019-04-30 23:51
2019年4月30日讯/生物谷BIOON/---基因组编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一,受到人们的高度重视。CRISPR是规律间隔性成簇短回文重复序列的简称,Cas是CRISPR相关蛋白的简称。CRISPR/Cas最初是在细菌体内发现的,是细菌用来识别和摧毁抗噬菌体和其他病原体入侵的防御系统。 图片来自Thomas Splettstoesser (Wi
2019年4月30日讯/生物谷BIOON/---基因组编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一,受到人们的高度重视。CRISPR是规律间隔性成簇短回文重复序列的简称,Cas是CRISPR相关蛋白的简称。CRISPR/Cas最初是在细菌体内发现的,是细菌用来识别和摧毁抗噬菌体和其他病原体入侵的防御系统。
2018年11月26日,中国科学家贺建奎声称世界上首批经过基因编辑的婴儿---一对双胞胎女性婴儿---在11月出生。他利用一种强大的基因编辑工具CRISPR-Cas9对这对双胞胎的一个基因进行修改,使得她们出生后就能够天然地抵抗HIV感染。这也是世界首例免疫艾滋病基因编辑婴儿。这条消息瞬间在国内外网站上迅速发酵,引发千层浪。有部分科学家支持贺建奎的研究,但是更多的是质疑,甚至是谴责。
即将过去的3月份,有哪些重大的CRISPR/Cas研究或发现呢?小编梳理了一下这个月生物谷报道的CRISPR/Cas研究方面的新闻,供大家阅读。
1.Nat Med:优化的CRISPR-Cas9基因编辑有望治疗镰状细胞病
doi:10.1038/s41591-019-0401-y
在一项新的研究中,来自美国达纳法伯癌症研究所、波士顿儿童医院和马萨诸塞大学医学院等研究机构的研究人员通过将CRISPR-Cas9基因编辑应用于患者自己的造血干细胞中,开发出一种治疗一种最为常见的遗传性血液疾病---镰状细胞病(sickle cell disease)--- 的策略。这种方法克服了之前的技术挑战,而且要比过去更有效地对造血干细胞进行编辑。相关研究结果于2019年3月25日在线发表在Nature Medicine期刊上,论文标题为“Highly efficient therapeutic gene editing of human hematopoietic stem cells”。论文通 讯作者为Daniel Bauer博士,论文第一作者为Yuxuan Wu和Jing Zeng。
这项新的研究使用了CRISPR-Cas9技术,特别是马萨诸塞大学医学院的Scot Wolfe博士领导的一个研究团队进行基因修饰过的Cas9蛋白,来优化基因编辑。在之前对人造血干/祖细胞的基因组进行编辑的尝试中,一旦将这些基因编辑的细胞植入骨髓中,基因编辑的效率、 特异性和长期稳定性就会发生变化。这种新技术提高了基因编辑的靶向性和持久性。
之前在波士顿儿童医院的研究已表明,让一种名为BCL11A的基因失活允许红细胞即便在出生后也会继续产生胎儿形式的血红蛋白。胎儿血红蛋白不会产生镰刀形状,能够代替有缺陷的“成年”血红蛋白。最近,Bauer发现了一个更安全的靶标:BCL11A基因的增强子,仅在 红细胞中有活性。
Bauer说,“通过使用我们开发的这种新型的非常有效的方法,我们能够在我们收集的几乎所有的造血干细胞中对BCL11A的增强子进行编辑,从而克服了对这些细胞进行基因编辑所面临的一些技术挑战。在我们的实验中,95%以上的增强子序列拷贝以我们期望的治疗方式 发生改变。”
这种策略使得携带来自镰状细胞病患者的造血干细胞的小鼠能够产生具有足够的胎儿血红蛋白的红细胞,从而阻止红细胞产生镰刀形状。Bauer团队发现这些经过基因编辑的造血干细胞在移植到骨髓中后产生得到基因校正的红细胞。随后,当从这些小鼠中分离出造血干细 胞并移植到其他小鼠中时,这些造血干细胞再次定植并且仍然携带治疗性的基因变化。
2.bioRxiv:成功利用CRISPR-Cas9对爬行动物进行基因编辑
doi:10.1101/591446
在一项新的研究中,来自美国佐治亚大学的研究人员发现一种可在爬行动物身上使用CRISPR-Cas9基因编辑工具的方法。相关研究结果近期发表在bioRxiv预印版服务器上,论文标题为“CRISPR-Cas9 Gene Editing in Lizards Through Microinjection of Unfertilized Oocytes”。在这篇论文中,他们描述了他们开发的这种技术,以及它在测试的蜥蜴中的效果。
随着科学家们试图更多地了解CRISPR-Cas9本身、它的工作机制及其潜在的应用,它已被用于各种各样的实验。不过,在早期,科学家们认为CRISPR不能与爬行动物一起使用,这是因为爬行动物具有独特的生殖系统---比如,雌性蜥蜴储存精子,仅在最方便的使用加以使 用,这就使得注射CRISPR-Cas9工具较为困难(如果不是不可能的话)。此外,还存在将针插入卵壳而不损坏它和阻止胚胎发育的问题。但是,存在困难并未意味着不可能,正如这些研究人员发现的那样,他们开发出一种成功的解决方法:利用CRISPR-Cas9对几只蜥蜴中 的细胞进行基因编辑。
为了在蜥蜴中使用CRISPR-Cas9,这些研究人员切开了几只测试的雌性蜥蜴并在受精前将这种编辑工具直接注射到卵子中,同时这些卵子仍然存在于母体的卵巢中,然后顺其自然发育。总之,他们将这种编辑工具注射到21只蜥蜴的146个卵子中。在他们的实验中, CRISPR-Cas9经编程后对酪氨酸酶编码基因进行编辑,其中这个基因负责确定蜥蜴的颜色---当它受到失活时,蜥蜴将患上白化病(albino)。他们报道,这种编辑技术成功了四次---4只患有白化病的蜥蜴出生了。他们指出他们的技术应当也适用于其他种类的蜥蜴。
3.Mol Cell:新型DNA剪切技术比传统CRISPR-Cas9更优越
doi:10.1016/j.molcel.2019.03.014
近年来,一种名为CRISPR-Cas9的工具改变了基因研究,就像一把小剪刀一样,允许科学家在精确的位置剪断和编辑DNA链。但有时候,完成这项工作需要的不仅仅是剪刀。现在,一个合作的国际团队推出了一种新的基于CRISPR的工具,它更像是一台粉碎机,能够清除人 体细胞中的长链DNA。
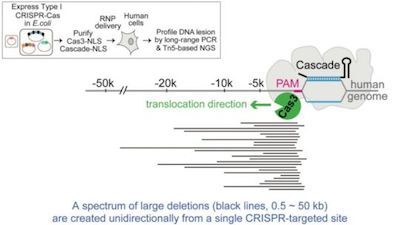
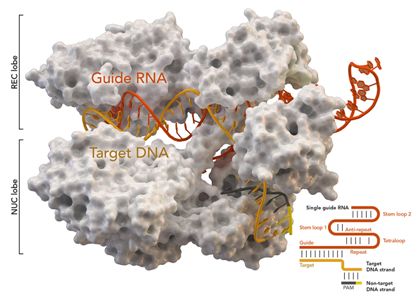
在最近发表在《Molecular Cell》杂志上的研究中,作者描述了他们如何成功地获得了一种名为Type I CRISPR-Cas3的CRISPR-Cas系统,这是第一次在人类细胞中作为远程DNA编辑工具,提供了比当前Cas9更有效的定位和删除长链DNA的方法。这种工具可以用于基因研究 ,以了解疾病的基础,并治疗与长链DNA相关的疾病。
在该研究中,作者他尝试将细菌CRISPR成分作为蛋白质传递到人类胚胎干细胞和另一种称为HAP1的细胞中。通过guideRNA分子,该团队成功地删除了从几百碱基对到100千碱基的靶DNA的部分。
4.Science子刊:重大进展!改变Cas9和gRNA递送比例可提高CRISPR编辑效率
doi:10.1126/sciadv.aav4324
在一项新的研究中,来自美国德克萨斯大学西南医学中心的研究人员开发出一种方法来提高杜兴氏肌肉营养不良症(Duchenne muscular dystrophy, DMD)CRISPR基因编辑的效率,这可能对优化针对其他疾病的基因疗法产生影响。相关研究结果发表在2019年3月6日的 Science Advances期刊上,论文标题为“CRISPR-Cas9 corrects Duchenne muscular dystrophy exon 44 deletion mutations in mice and human cells”。
这些研究人员利用一种单切割CRISPR基因编辑技术校正小鼠和人类细胞中的一种导致DMD的常见突变。DMD是一种由抗肌萎缩蛋白(dystrophin)缺失导致的致命疾病。在对这种技术进行测试时,他们发现调整CRISPR基因编辑组分的剂量能够显著地改善编辑后的基因产生 的抗肌萎缩蛋白的数量。他们进一步发现,这些组分的最佳比例的变化取决于DNA的哪一部分被编辑。
论文通讯作者、德克萨斯大学西南医学中心的Eric Olson博士说,“当我们在抗肌萎缩蛋白编码基因的其他缺陷部分测试CRISPR时,调节我们的组分配方以获得最佳结果可能很重要。这种新的见解可能进一步促进了CRISPR在治疗DMD和许多其他疾病中的应用。”
5.bioRxiv:新研究揭示CRISPR系统的抗病毒工作机制
doi:10.1101/453720
十年前,“没有人认为细菌具有复杂的适应性免疫系统,”蒙大拿州立大学文学与科学学院和农业学院微生物学和免疫学系副教授Blake Wiedenheft说。然而,从那以后,研究人员发现了一种细菌利用机器分子检测和摧毁入侵病毒的机制。这种免疫反应被称为CRISPR, 这是一个缩写,描述了细菌如何将病毒DNA片段整合到自己的基因组中,作为未来识别和对抗病毒的一种方式。
对于Wiedenheft而言,不断增长的CRISPR知识引发了其他问题:病毒是否找到了破坏细菌防御的方法,内在的分子机制如何?
Wiedenheft在3月11日发表于Molecular Cell期刊的一篇科学论文中发表了他的团队最新发现,不仅描述了CRISPR防御的新细节,而且还发现了扩大科学家对资源丰富病毒的理解的发现。
使用强大的电子显微镜和尖端的图像处理技术,Wiedenheft和他的合作者,斯克里普斯研究所的副教授Gabriel Lander,可以看到一个复杂的CRISPR分子通过展开类似于“灯塔”的分子臂来响应病毒DNA。Wiedenheft解释说,灯塔就像“发出危险信号的红色闪光灯”,作 为其他CRISPR分子的生物化学线索来摧毁病毒。
6.Nature子刊:当心!DNA扭曲增加CRISPR-Cas9脱靶编辑风险
doi:10.1038/s41594-019-0188-z
在一项新的研究中,来自英国帝国理工学院和阿斯利康公司的研究人员指出当使用CRISPR-Cas9时,在基因表达和其他细胞过程中经常发生的DNA扭曲可能导致基因组脱靶变化。这些研究结果可能有助于为在临床应用上提高基因编辑准确性铺平道路。相关研究结果发表在 2019年3月的Nature Structural and Molecular Biology期刊上,论文标题为“DNA stretching induces Cas9 off-target activity”。

CRISPR-Cas9是一种允许人们发现和编辑DNA链的基因编辑工具。随着科学家们在医学、药物发现和农业等多个领域使用CRISPR-Cas9,它因它的多种用途获得了全球的认可。在这项新的研究中,CRISPR-Cas9的准确度和精确度是通过一种新的方法进行研究的:使用光学镊 子---一种使用激光束操纵DNA的工具---模拟DNA自然经历的扭曲,就像是细胞的分子机器读取了它。他们随后利用CRISPR-Cas9编辑基因并使用荧光显微镜监测它的准确性。结果表明,当DNA松散和松弛时,CRISPR是准确的,但是当DNA发生扭曲---在这种情况下,DNA遭受 高度拉伸---时,CRISPR编辑准确性降低,并且观察到脱靶编辑。了解这种效应将有助于设计具有更高准确度的CRISPR系统,以及评估这种风险的方法。
论文共同通讯作者、帝国理工学院的David Rueda教授说,“CRISPR-Cas9作为一种有助于预防或抵抗因DNA突变引起的疾病的潜在工具而备受瞩目,我们想要研究它的准确性。当我们操纵DNA结构来模拟DNA经历的自然扭曲时,我们发现在非预期的位点上进行编辑。在这种 情形下,CRISPR-Cas9在靶向系统中显示多达50%的错配,这将是在临床环境中使用这种基因编辑工具时需要考虑的一个问题。”
7.New Phytologist:基于CRISPR技术可以标记多物种的基因组
doi:10.1111/nph.15720
自2012年提出其机制以来,CRISPR / Cas9系统一直在科学界引起涟漪。许多科学家已经发现了Cas9蛋白的剪刀样特性的不同应用。最近,来自莱布尼茨植物遗传和作物植物研究所(IPK Gatersleben)的研究人员现在已经找到了一种以稍微不同的方式利用RNA /蛋白质复 合物的方法。除了传统的原位杂交外,新的RNA引导的核酸内切酶 - 原位标记工具(RGEN-ISL)不再需要DNA的变性。因此,新方法使染色质保持完整,从而可以研究样品的结构。此外,RGEN-ISL可与蛋白质检测方法结合使用,并可实现标记过程的实时可视化。虽然 RGEN-ISL最初是为植物基因组开发的,但它可用于所有生物体,并且在染色体生物学领域显示出一种很有前途的新工具。
在过去的30年中,荧光原位杂交(FISH)已成为用于在染色体水平上可视化原位DNA序列的已建立且常用的方法。然而,该方法需要使所研究的DNA变性,因此经常损害样品的结构。通过将RGEN-ISL方法基于CRISPR-Cas9,IPK研究人员设法绕过FISH的变性步骤,同时整合 了常规FISH方法的所需荧光标记特性。由于新的细胞遗传学工具保留了样本的结构,它开辟了研究基因组时空结构的选择。
进一步的实验表明,RGEN-ISL优于传统方法组合,例如FISH和免疫组织化学,需要较少的制备并且相对更快和更便宜。此外,新方法可在4°C至37°C的宽温度范围内发挥作用,还可与其他蛋白质检测和成像方法结合使用。进一步的优点是RGEN-ISL允许实时可视化CRISPR / Cas9介导的DNA标记,因此揭示了反应的动力学。
8.Nat Biotechnol:新研究能够降低基因编辑的出错几率
doi:10.1038/s41587-019-0050-1
最近,韩国IBS基因组工程中心的研究人员已经确定了基于CRISPR靶向腺嘌呤的基因编辑工具(ABE)的错误率。对这一工具的错误率的评估对于其在临床和生物技术中的应用至关重要。他们的研究结果发表在Nature Biotechnology上。
由Jin-Soo Kim领导的研究小组研究了人类细胞中最近开发的ABE蛋白ABE7.10的错误率。他们确定了受ABE7.10影响的人类基因组位置,并扫描了超出目标的错误。为此,他们使用了改编版的Digenome-seq技术。修改后的Digenome-seq可以检测整个人类基因组中平均60个 脱靶错误。有趣的是,尽管这三种蛋白质被设计成针对同一个位点,但它们识别出不同的脱靶点。
IBS生物学家还展示了一些策略来抑制脱靶修改的数量。在guide RNA末尾添加几个G减少了脱靶错误,以及使用不同类型的Cas9。通过预装配的核糖核蛋白,而不是通过质粒,等等。
9.Science:我国李亦学课题组和杨辉课题组揭示胞嘧啶碱基编辑器诱导大量的单位点脱靶突变
doi:10.1126/science.aav9973
基因组编辑在治疗由致命性突变引起的遗传疾病上有很大的潜力。对基因组编辑的脱靶效应进行全面分析是验证这种编辑实用性所必需的。科学家们已开发出多种方法来检测全基因组范围内的基因编辑脱靶位点。然而,这些方法并不适用于检测体内的单核苷酸变异(SNV)。
在一项新的研究中,中国科学院的李亦学(Yixue Li)课题组、杨辉(Hui Yang)课题组和美国斯坦福大学的Lars M. Steinmetz课题组开发出一种称为GOTI(genome-wide off-target analysis by two-cell embryo injection, 利用双细胞胚胎注射进行全基因组脱靶分析)的方法来评估三种经常使用的基因编辑工具---CRISPR/Cas9、胞嘧啶碱基编辑器3(BE3, rAPOBEC1-nCas9-UGI)、腺嘌呤碱基编辑器7.10(ABE7.10, TadA-TadA*-nCas9)---诱导的脱靶效应。简言之,这些研究人员将CRISPR/Cas9、BE3或ABE7.10与Cre mRNA一起注射到源自Ai9(CAG-LoxP-Stop-LoxP-tdTomato)小鼠的双细胞胚胎的卵裂球中。在胚胎期第14.5天(ED14.5)时,基于tdTomato的表达,利用荧光活化细胞分选方法(FACS)对经过编辑的卵裂球和未经过编辑的卵裂球的后代细胞进行分选。与此同时,在ED14.5时,整个胚胎也很容易经消化后获得足够的单细胞。他们随后对tdTomato阳性细胞和tdTomato阴性细胞单独地进行全基因组测序(WGS)。以来自相同胚胎的tdTomato阴性样本作为对照,利用三种算法对来自相同胚胎的tdTomato阳性样本中的SNV和indel(insertion or deletion, 插入或删除)进行研究。
在这项新的研究中,这些研究人员包括了12个组:一个Cre组(Cre-only, 仅注射Cre)、6个具有或不具有单向导RNA(sgRNA)的Cas9组(Cas9、Cas9-LacZ、Cas9-Pde6b、Cas9-Tyr-A、Cas9-Tyr-B和Cas9-Tyr-C)、三个具有或不具有sgRNA的BE3组(BE3、BE3-Tyr-C和BE3-Tyr-D)和两个具有或不具有sgRNA的ABE组(ABE7.10和ABE7.10-Tyr-E)。首先,他们通过桑格测序(Sanger sequencing)在胚胎8细胞和ED14.5时验证了他们的方法在胚胎中的在靶效率。为了进一步探究在靶效率和潜在的全基因组脱靶效应,他们对来自ED14.5胚胎的46个样本进行全基因组测序,并证实Cas9、BE3和ABE7.10在tdTomato阳性细胞中高效地诱导indel和核苷酸替换。
令人吃惊的是,这些研究人员在经过BE3处理的胚胎中,发现了平均每个胚胎存在283个SNV,这一水平至少要比在经过Cre或Cas9处理的胚胎中观察到的高出20倍。相反之下,在经过ABE7.10处理的胚胎中,平均每个胚胎存在10个SNV,这一频率接近于自发性突变率。他们进一步地将在BE3-only组(即仅注射BE3的组)中鉴定出的脱靶位点与在BE3-Tyr-C组或BE3-Tyr-D组中鉴定出的脱靶位点进行了比较,并发现sgRNA的存在并不诱导显著高的SNV(P=0.21,Kruskal-Wallis test)。此外,这些变异是在tdTomato阳性细胞而不是在tdTomato阴性细胞中特异地鉴定出的。
令人关注的是,在经过BE3编辑的细胞中鉴定出的90%以上的SNV是G>A或C>T,这一突变偏好并没有在经过Cre、Cas9或ABE7.10处理的细胞中观察到。这一突变偏好与APOBEC1本身的突变偏好相同,这表明这些突变并不是自发的而是由BE3编辑诱导的。之前的研究已表明APOBEC家族的几个成员(包括APOBEC1)发挥作用需要单链DNA。与此相一致的是,这些研究人员的分析表明由BE3诱导的SNV在转录区域中显著富集,特别是在高度表达的基因中。有趣的是,这些脱靶位点中的任何一个并不与经过BE3处理的胚胎中观察到的相同,而且也不与预测的脱靶突变发生重叠。此外,也并未在脱靶序列和靶序列之间观察到相似性,然而,预测的排名靠前的脱靶位点与BE3的在靶位点存在着较高的序列相似性。因此,BE3引起的脱靶SNV并不依赖于sgRNA并且可能是由APOBEC1过度表达导致的。
在经过BE3处理的胚胎中观察到的1698个SNV中,26个SNV位于外显子中,其中的14个导致非同义变化。这些研究人员成功地将通过PCR扩增了其中的20个SNV并通过桑格测序证实了它们的存在。他们还发现1个SNV位于一个原癌基因中,13个SNV位于肿瘤抑制基因中,这就让人对BE3编辑的致癌风险感到担忧。这种风险可能通过表达较低数量的BE3加以降低。然而,他们发现当表达较低数量的BE3时,在靶效率逐渐降低。
令人关注的是,这些研究人员发现大量新生突变是由BE3诱导的,这一点在之前的研究中并未报道过。一种可能的解释就是GOTI研究了源自单个经过基因编辑的卵裂球的细胞群体,而之前的研究使用了大量的细胞群体,在那里,编辑是可变的,而且由于细胞群体平均化,这会导致随机的脱靶信号丢失。不同于BE3的是,ABE7.10并不导致SNV数量增加,这很可能是缺乏TadA的DNA结合能力。这些碱基编辑器的脱靶效应可能通过降低APOBEC1的DNA结合能力或者使用不同的胞苷脱氨酶版本来加以降低。总之,GOTI可用于研究多种基因编辑工具的脱靶效应,而且不受在不同个体中存在的单核苷酸多态性(SNP)的干扰。
10.Science:中科院高彩霞课题组发现胞嘧啶碱基编辑器引发意想不到的全基因组脱靶突变
doi:10.1126/science.aaw7166
在一项新的研究中,中国科学院的高彩霞(Caixia Gao)课题组通过对作为一种重要的作物物种的水稻进行全基因组测序对胞嘧啶碱基编辑器(BE3和HF1-BE3)和腺嘌呤碱基编辑器(ABE)产生的脱靶突变进行全面调查。他们发现胞嘧啶碱基编辑器(BE3和HF1-BE3)诱导全基因组脱靶突变。相关研究结果于2019年2月28日在线发表在Science期刊上,论文标题为“Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice”。
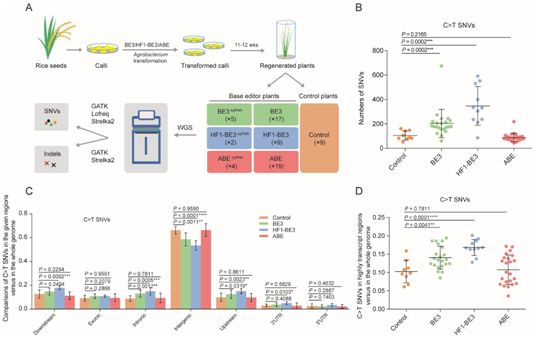
在这项新的研究中,高彩霞课题组选择了三种广泛使用的碱基编辑器:BE3、高保真BE3(HF1-BE3)和ABE,其中BE3和HF1-BE3属于胞嘧啶碱基编辑器(CBE)。将靶向11个基因组位点的总共14个碱基编辑器构造体通过农杆菌转化方法转化到水稻中。他们利用全基因组测序对由BE3、HF1-BE3或ABE编辑的再生T0水稻植物;经过这些碱基编辑器转化但没有经过sgRNA转化的水稻植物以及两个对照组水稻植物(即野生型水稻和转基因水稻的无效分离株)进行分析。
这些碱基编辑器组(即BE3组、HF1-BE3组和ABE组)和对照组在发现的插入或删除(insertion or deletion, indel)数量上没有显著差异。相反之下,BE3组和HF1-BE3组要比ABE组和对照组具有显著更多的单核苷酸变异(SNV)。
在这些碱基编辑器组和对照组中,每株水稻植物的C>T单核苷酸变异(SNV)的平均数量为:203(BE3)、347(HF1-BE3)、88(ABE)和105(对照组)。因此,BE3组和HF1-BE3组水稻植物中的C>T单核苷酸变异数量分别比对照组水稻植物高94.5%和231.9%。
总而言之,由高彩霞课题组产生的数据表明是BE3和HF1-BE3,而并不是ABE,在水稻中诱导全基因组脱靶突变。这些脱靶突变主要是C>T单核苷酸变异,在转录的基因区域中富集,通过当前的计算机方法是无法预测的。含有胞嘧啶脱氨酶的碱基转化单元可能是由BE3和HF1-BE3引发的较高数量的脱靶单核苷酸变异的原因,因而需要加以优化以提高胞嘧啶碱基编辑器(BE3和HF1-BE3)的特异性。(生物谷 Bioon.com)

图片来自Thomas Splettstoesser (Wikipedia, CC BY-SA 4.0)。
即将过去的3月份,有哪些重大的CRISPR/Cas研究或发现呢?小编梳理了一下这个月生物谷报道的CRISPR/Cas研究方面的新闻,供大家阅读。
1.Nat Med:优化的CRISPR-Cas9基因编辑有望治疗镰状细胞病
doi:10.1038/s41591-019-0401-y
在一项新的研究中,来自美国达纳法伯癌症研究所、波士顿儿童医院和马萨诸塞大学医学院等研究机构的研究人员通过将CRISPR-Cas9基因编辑应用于患者自己的造血干细胞中,开发出一种治疗一种最为常见的遗传性血液疾病---镰状细胞病(sickle cell disease)--- 的策略。这种方法克服了之前的技术挑战,而且要比过去更有效地对造血干细胞进行编辑。相关研究结果于2019年3月25日在线发表在Nature Medicine期刊上,论文标题为“Highly efficient therapeutic gene editing of human hematopoietic stem cells”。论文通 讯作者为Daniel Bauer博士,论文第一作者为Yuxuan Wu和Jing Zeng。
这项新的研究使用了CRISPR-Cas9技术,特别是马萨诸塞大学医学院的Scot Wolfe博士领导的一个研究团队进行基因修饰过的Cas9蛋白,来优化基因编辑。在之前对人造血干/祖细胞的基因组进行编辑的尝试中,一旦将这些基因编辑的细胞植入骨髓中,基因编辑的效率、 特异性和长期稳定性就会发生变化。这种新技术提高了基因编辑的靶向性和持久性。
之前在波士顿儿童医院的研究已表明,让一种名为BCL11A的基因失活允许红细胞即便在出生后也会继续产生胎儿形式的血红蛋白。胎儿血红蛋白不会产生镰刀形状,能够代替有缺陷的“成年”血红蛋白。最近,Bauer发现了一个更安全的靶标:BCL11A基因的增强子,仅在 红细胞中有活性。
Bauer说,“通过使用我们开发的这种新型的非常有效的方法,我们能够在我们收集的几乎所有的造血干细胞中对BCL11A的增强子进行编辑,从而克服了对这些细胞进行基因编辑所面临的一些技术挑战。在我们的实验中,95%以上的增强子序列拷贝以我们期望的治疗方式 发生改变。”
这种策略使得携带来自镰状细胞病患者的造血干细胞的小鼠能够产生具有足够的胎儿血红蛋白的红细胞,从而阻止红细胞产生镰刀形状。Bauer团队发现这些经过基因编辑的造血干细胞在移植到骨髓中后产生得到基因校正的红细胞。随后,当从这些小鼠中分离出造血干细 胞并移植到其他小鼠中时,这些造血干细胞再次定植并且仍然携带治疗性的基因变化。
2.bioRxiv:成功利用CRISPR-Cas9对爬行动物进行基因编辑
doi:10.1101/591446
在一项新的研究中,来自美国佐治亚大学的研究人员发现一种可在爬行动物身上使用CRISPR-Cas9基因编辑工具的方法。相关研究结果近期发表在bioRxiv预印版服务器上,论文标题为“CRISPR-Cas9 Gene Editing in Lizards Through Microinjection of Unfertilized Oocytes”。在这篇论文中,他们描述了他们开发的这种技术,以及它在测试的蜥蜴中的效果。
随着科学家们试图更多地了解CRISPR-Cas9本身、它的工作机制及其潜在的应用,它已被用于各种各样的实验。不过,在早期,科学家们认为CRISPR不能与爬行动物一起使用,这是因为爬行动物具有独特的生殖系统---比如,雌性蜥蜴储存精子,仅在最方便的使用加以使 用,这就使得注射CRISPR-Cas9工具较为困难(如果不是不可能的话)。此外,还存在将针插入卵壳而不损坏它和阻止胚胎发育的问题。但是,存在困难并未意味着不可能,正如这些研究人员发现的那样,他们开发出一种成功的解决方法:利用CRISPR-Cas9对几只蜥蜴中 的细胞进行基因编辑。
为了在蜥蜴中使用CRISPR-Cas9,这些研究人员切开了几只测试的雌性蜥蜴并在受精前将这种编辑工具直接注射到卵子中,同时这些卵子仍然存在于母体的卵巢中,然后顺其自然发育。总之,他们将这种编辑工具注射到21只蜥蜴的146个卵子中。在他们的实验中, CRISPR-Cas9经编程后对酪氨酸酶编码基因进行编辑,其中这个基因负责确定蜥蜴的颜色---当它受到失活时,蜥蜴将患上白化病(albino)。他们报道,这种编辑技术成功了四次---4只患有白化病的蜥蜴出生了。他们指出他们的技术应当也适用于其他种类的蜥蜴。
3.Mol Cell:新型DNA剪切技术比传统CRISPR-Cas9更优越
doi:10.1016/j.molcel.2019.03.014
近年来,一种名为CRISPR-Cas9的工具改变了基因研究,就像一把小剪刀一样,允许科学家在精确的位置剪断和编辑DNA链。但有时候,完成这项工作需要的不仅仅是剪刀。现在,一个合作的国际团队推出了一种新的基于CRISPR的工具,它更像是一台粉碎机,能够清除人 体细胞中的长链DNA。

(图片来源:Zhang lab, University of Michigan)
在最近发表在《Molecular Cell》杂志上的研究中,作者描述了他们如何成功地获得了一种名为Type I CRISPR-Cas3的CRISPR-Cas系统,这是第一次在人类细胞中作为远程DNA编辑工具,提供了比当前Cas9更有效的定位和删除长链DNA的方法。这种工具可以用于基因研究 ,以了解疾病的基础,并治疗与长链DNA相关的疾病。
在该研究中,作者他尝试将细菌CRISPR成分作为蛋白质传递到人类胚胎干细胞和另一种称为HAP1的细胞中。通过guideRNA分子,该团队成功地删除了从几百碱基对到100千碱基的靶DNA的部分。
4.Science子刊:重大进展!改变Cas9和gRNA递送比例可提高CRISPR编辑效率
doi:10.1126/sciadv.aav4324
在一项新的研究中,来自美国德克萨斯大学西南医学中心的研究人员开发出一种方法来提高杜兴氏肌肉营养不良症(Duchenne muscular dystrophy, DMD)CRISPR基因编辑的效率,这可能对优化针对其他疾病的基因疗法产生影响。相关研究结果发表在2019年3月6日的 Science Advances期刊上,论文标题为“CRISPR-Cas9 corrects Duchenne muscular dystrophy exon 44 deletion mutations in mice and human cells”。
这些研究人员利用一种单切割CRISPR基因编辑技术校正小鼠和人类细胞中的一种导致DMD的常见突变。DMD是一种由抗肌萎缩蛋白(dystrophin)缺失导致的致命疾病。在对这种技术进行测试时,他们发现调整CRISPR基因编辑组分的剂量能够显著地改善编辑后的基因产生 的抗肌萎缩蛋白的数量。他们进一步发现,这些组分的最佳比例的变化取决于DNA的哪一部分被编辑。
论文通讯作者、德克萨斯大学西南医学中心的Eric Olson博士说,“当我们在抗肌萎缩蛋白编码基因的其他缺陷部分测试CRISPR时,调节我们的组分配方以获得最佳结果可能很重要。这种新的见解可能进一步促进了CRISPR在治疗DMD和许多其他疾病中的应用。”
5.bioRxiv:新研究揭示CRISPR系统的抗病毒工作机制
doi:10.1101/453720
十年前,“没有人认为细菌具有复杂的适应性免疫系统,”蒙大拿州立大学文学与科学学院和农业学院微生物学和免疫学系副教授Blake Wiedenheft说。然而,从那以后,研究人员发现了一种细菌利用机器分子检测和摧毁入侵病毒的机制。这种免疫反应被称为CRISPR, 这是一个缩写,描述了细菌如何将病毒DNA片段整合到自己的基因组中,作为未来识别和对抗病毒的一种方式。
对于Wiedenheft而言,不断增长的CRISPR知识引发了其他问题:病毒是否找到了破坏细菌防御的方法,内在的分子机制如何?
Wiedenheft在3月11日发表于Molecular Cell期刊的一篇科学论文中发表了他的团队最新发现,不仅描述了CRISPR防御的新细节,而且还发现了扩大科学家对资源丰富病毒的理解的发现。
使用强大的电子显微镜和尖端的图像处理技术,Wiedenheft和他的合作者,斯克里普斯研究所的副教授Gabriel Lander,可以看到一个复杂的CRISPR分子通过展开类似于“灯塔”的分子臂来响应病毒DNA。Wiedenheft解释说,灯塔就像“发出危险信号的红色闪光灯”,作 为其他CRISPR分子的生物化学线索来摧毁病毒。
6.Nature子刊:当心!DNA扭曲增加CRISPR-Cas9脱靶编辑风险
doi:10.1038/s41594-019-0188-z
在一项新的研究中,来自英国帝国理工学院和阿斯利康公司的研究人员指出当使用CRISPR-Cas9时,在基因表达和其他细胞过程中经常发生的DNA扭曲可能导致基因组脱靶变化。这些研究结果可能有助于为在临床应用上提高基因编辑准确性铺平道路。相关研究结果发表在 2019年3月的Nature Structural and Molecular Biology期刊上,论文标题为“DNA stretching induces Cas9 off-target activity”。

图片来自David Rueda。
CRISPR-Cas9是一种允许人们发现和编辑DNA链的基因编辑工具。随着科学家们在医学、药物发现和农业等多个领域使用CRISPR-Cas9,它因它的多种用途获得了全球的认可。在这项新的研究中,CRISPR-Cas9的准确度和精确度是通过一种新的方法进行研究的:使用光学镊 子---一种使用激光束操纵DNA的工具---模拟DNA自然经历的扭曲,就像是细胞的分子机器读取了它。他们随后利用CRISPR-Cas9编辑基因并使用荧光显微镜监测它的准确性。结果表明,当DNA松散和松弛时,CRISPR是准确的,但是当DNA发生扭曲---在这种情况下,DNA遭受 高度拉伸---时,CRISPR编辑准确性降低,并且观察到脱靶编辑。了解这种效应将有助于设计具有更高准确度的CRISPR系统,以及评估这种风险的方法。
论文共同通讯作者、帝国理工学院的David Rueda教授说,“CRISPR-Cas9作为一种有助于预防或抵抗因DNA突变引起的疾病的潜在工具而备受瞩目,我们想要研究它的准确性。当我们操纵DNA结构来模拟DNA经历的自然扭曲时,我们发现在非预期的位点上进行编辑。在这种 情形下,CRISPR-Cas9在靶向系统中显示多达50%的错配,这将是在临床环境中使用这种基因编辑工具时需要考虑的一个问题。”
7.New Phytologist:基于CRISPR技术可以标记多物种的基因组
doi:10.1111/nph.15720
自2012年提出其机制以来,CRISPR / Cas9系统一直在科学界引起涟漪。许多科学家已经发现了Cas9蛋白的剪刀样特性的不同应用。最近,来自莱布尼茨植物遗传和作物植物研究所(IPK Gatersleben)的研究人员现在已经找到了一种以稍微不同的方式利用RNA /蛋白质复 合物的方法。除了传统的原位杂交外,新的RNA引导的核酸内切酶 - 原位标记工具(RGEN-ISL)不再需要DNA的变性。因此,新方法使染色质保持完整,从而可以研究样品的结构。此外,RGEN-ISL可与蛋白质检测方法结合使用,并可实现标记过程的实时可视化。虽然 RGEN-ISL最初是为植物基因组开发的,但它可用于所有生物体,并且在染色体生物学领域显示出一种很有前途的新工具。
在过去的30年中,荧光原位杂交(FISH)已成为用于在染色体水平上可视化原位DNA序列的已建立且常用的方法。然而,该方法需要使所研究的DNA变性,因此经常损害样品的结构。通过将RGEN-ISL方法基于CRISPR-Cas9,IPK研究人员设法绕过FISH的变性步骤,同时整合 了常规FISH方法的所需荧光标记特性。由于新的细胞遗传学工具保留了样本的结构,它开辟了研究基因组时空结构的选择。
进一步的实验表明,RGEN-ISL优于传统方法组合,例如FISH和免疫组织化学,需要较少的制备并且相对更快和更便宜。此外,新方法可在4°C至37°C的宽温度范围内发挥作用,还可与其他蛋白质检测和成像方法结合使用。进一步的优点是RGEN-ISL允许实时可视化CRISPR / Cas9介导的DNA标记,因此揭示了反应的动力学。
8.Nat Biotechnol:新研究能够降低基因编辑的出错几率
doi:10.1038/s41587-019-0050-1
最近,韩国IBS基因组工程中心的研究人员已经确定了基于CRISPR靶向腺嘌呤的基因编辑工具(ABE)的错误率。对这一工具的错误率的评估对于其在临床和生物技术中的应用至关重要。他们的研究结果发表在Nature Biotechnology上。
由Jin-Soo Kim领导的研究小组研究了人类细胞中最近开发的ABE蛋白ABE7.10的错误率。他们确定了受ABE7.10影响的人类基因组位置,并扫描了超出目标的错误。为此,他们使用了改编版的Digenome-seq技术。修改后的Digenome-seq可以检测整个人类基因组中平均60个 脱靶错误。有趣的是,尽管这三种蛋白质被设计成针对同一个位点,但它们识别出不同的脱靶点。
IBS生物学家还展示了一些策略来抑制脱靶修改的数量。在guide RNA末尾添加几个G减少了脱靶错误,以及使用不同类型的Cas9。通过预装配的核糖核蛋白,而不是通过质粒,等等。
9.Science:我国李亦学课题组和杨辉课题组揭示胞嘧啶碱基编辑器诱导大量的单位点脱靶突变
doi:10.1126/science.aav9973
基因组编辑在治疗由致命性突变引起的遗传疾病上有很大的潜力。对基因组编辑的脱靶效应进行全面分析是验证这种编辑实用性所必需的。科学家们已开发出多种方法来检测全基因组范围内的基因编辑脱靶位点。然而,这些方法并不适用于检测体内的单核苷酸变异(SNV)。
在一项新的研究中,中国科学院的李亦学(Yixue Li)课题组、杨辉(Hui Yang)课题组和美国斯坦福大学的Lars M. Steinmetz课题组开发出一种称为GOTI(genome-wide off-target analysis by two-cell embryo injection, 利用双细胞胚胎注射进行全基因组脱靶分析)的方法来评估三种经常使用的基因编辑工具---CRISPR/Cas9、胞嘧啶碱基编辑器3(BE3, rAPOBEC1-nCas9-UGI)、腺嘌呤碱基编辑器7.10(ABE7.10, TadA-TadA*-nCas9)---诱导的脱靶效应。简言之,这些研究人员将CRISPR/Cas9、BE3或ABE7.10与Cre mRNA一起注射到源自Ai9(CAG-LoxP-Stop-LoxP-tdTomato)小鼠的双细胞胚胎的卵裂球中。在胚胎期第14.5天(ED14.5)时,基于tdTomato的表达,利用荧光活化细胞分选方法(FACS)对经过编辑的卵裂球和未经过编辑的卵裂球的后代细胞进行分选。与此同时,在ED14.5时,整个胚胎也很容易经消化后获得足够的单细胞。他们随后对tdTomato阳性细胞和tdTomato阴性细胞单独地进行全基因组测序(WGS)。以来自相同胚胎的tdTomato阴性样本作为对照,利用三种算法对来自相同胚胎的tdTomato阳性样本中的SNV和indel(insertion or deletion, 插入或删除)进行研究。
在这项新的研究中,这些研究人员包括了12个组:一个Cre组(Cre-only, 仅注射Cre)、6个具有或不具有单向导RNA(sgRNA)的Cas9组(Cas9、Cas9-LacZ、Cas9-Pde6b、Cas9-Tyr-A、Cas9-Tyr-B和Cas9-Tyr-C)、三个具有或不具有sgRNA的BE3组(BE3、BE3-Tyr-C和BE3-Tyr-D)和两个具有或不具有sgRNA的ABE组(ABE7.10和ABE7.10-Tyr-E)。首先,他们通过桑格测序(Sanger sequencing)在胚胎8细胞和ED14.5时验证了他们的方法在胚胎中的在靶效率。为了进一步探究在靶效率和潜在的全基因组脱靶效应,他们对来自ED14.5胚胎的46个样本进行全基因组测序,并证实Cas9、BE3和ABE7.10在tdTomato阳性细胞中高效地诱导indel和核苷酸替换。
令人吃惊的是,这些研究人员在经过BE3处理的胚胎中,发现了平均每个胚胎存在283个SNV,这一水平至少要比在经过Cre或Cas9处理的胚胎中观察到的高出20倍。相反之下,在经过ABE7.10处理的胚胎中,平均每个胚胎存在10个SNV,这一频率接近于自发性突变率。他们进一步地将在BE3-only组(即仅注射BE3的组)中鉴定出的脱靶位点与在BE3-Tyr-C组或BE3-Tyr-D组中鉴定出的脱靶位点进行了比较,并发现sgRNA的存在并不诱导显著高的SNV(P=0.21,Kruskal-Wallis test)。此外,这些变异是在tdTomato阳性细胞而不是在tdTomato阴性细胞中特异地鉴定出的。
令人关注的是,在经过BE3编辑的细胞中鉴定出的90%以上的SNV是G>A或C>T,这一突变偏好并没有在经过Cre、Cas9或ABE7.10处理的细胞中观察到。这一突变偏好与APOBEC1本身的突变偏好相同,这表明这些突变并不是自发的而是由BE3编辑诱导的。之前的研究已表明APOBEC家族的几个成员(包括APOBEC1)发挥作用需要单链DNA。与此相一致的是,这些研究人员的分析表明由BE3诱导的SNV在转录区域中显著富集,特别是在高度表达的基因中。有趣的是,这些脱靶位点中的任何一个并不与经过BE3处理的胚胎中观察到的相同,而且也不与预测的脱靶突变发生重叠。此外,也并未在脱靶序列和靶序列之间观察到相似性,然而,预测的排名靠前的脱靶位点与BE3的在靶位点存在着较高的序列相似性。因此,BE3引起的脱靶SNV并不依赖于sgRNA并且可能是由APOBEC1过度表达导致的。
在经过BE3处理的胚胎中观察到的1698个SNV中,26个SNV位于外显子中,其中的14个导致非同义变化。这些研究人员成功地将通过PCR扩增了其中的20个SNV并通过桑格测序证实了它们的存在。他们还发现1个SNV位于一个原癌基因中,13个SNV位于肿瘤抑制基因中,这就让人对BE3编辑的致癌风险感到担忧。这种风险可能通过表达较低数量的BE3加以降低。然而,他们发现当表达较低数量的BE3时,在靶效率逐渐降低。
令人关注的是,这些研究人员发现大量新生突变是由BE3诱导的,这一点在之前的研究中并未报道过。一种可能的解释就是GOTI研究了源自单个经过基因编辑的卵裂球的细胞群体,而之前的研究使用了大量的细胞群体,在那里,编辑是可变的,而且由于细胞群体平均化,这会导致随机的脱靶信号丢失。不同于BE3的是,ABE7.10并不导致SNV数量增加,这很可能是缺乏TadA的DNA结合能力。这些碱基编辑器的脱靶效应可能通过降低APOBEC1的DNA结合能力或者使用不同的胞苷脱氨酶版本来加以降低。总之,GOTI可用于研究多种基因编辑工具的脱靶效应,而且不受在不同个体中存在的单核苷酸多态性(SNP)的干扰。
10.Science:中科院高彩霞课题组发现胞嘧啶碱基编辑器引发意想不到的全基因组脱靶突变
doi:10.1126/science.aaw7166
在一项新的研究中,中国科学院的高彩霞(Caixia Gao)课题组通过对作为一种重要的作物物种的水稻进行全基因组测序对胞嘧啶碱基编辑器(BE3和HF1-BE3)和腺嘌呤碱基编辑器(ABE)产生的脱靶突变进行全面调查。他们发现胞嘧啶碱基编辑器(BE3和HF1-BE3)诱导全基因组脱靶突变。相关研究结果于2019年2月28日在线发表在Science期刊上,论文标题为“Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice”。

图片来自Institute of Genetics and Developmental Biology, Chinese Academy of Sciences。
在这项新的研究中,高彩霞课题组选择了三种广泛使用的碱基编辑器:BE3、高保真BE3(HF1-BE3)和ABE,其中BE3和HF1-BE3属于胞嘧啶碱基编辑器(CBE)。将靶向11个基因组位点的总共14个碱基编辑器构造体通过农杆菌转化方法转化到水稻中。他们利用全基因组测序对由BE3、HF1-BE3或ABE编辑的再生T0水稻植物;经过这些碱基编辑器转化但没有经过sgRNA转化的水稻植物以及两个对照组水稻植物(即野生型水稻和转基因水稻的无效分离株)进行分析。
这些碱基编辑器组(即BE3组、HF1-BE3组和ABE组)和对照组在发现的插入或删除(insertion or deletion, indel)数量上没有显著差异。相反之下,BE3组和HF1-BE3组要比ABE组和对照组具有显著更多的单核苷酸变异(SNV)。
在这些碱基编辑器组和对照组中,每株水稻植物的C>T单核苷酸变异(SNV)的平均数量为:203(BE3)、347(HF1-BE3)、88(ABE)和105(对照组)。因此,BE3组和HF1-BE3组水稻植物中的C>T单核苷酸变异数量分别比对照组水稻植物高94.5%和231.9%。
总而言之,由高彩霞课题组产生的数据表明是BE3和HF1-BE3,而并不是ABE,在水稻中诱导全基因组脱靶突变。这些脱靶突变主要是C>T单核苷酸变异,在转录的基因区域中富集,通过当前的计算机方法是无法预测的。含有胞嘧啶脱氨酶的碱基转化单元可能是由BE3和HF1-BE3引发的较高数量的脱靶单核苷酸变异的原因,因而需要加以优化以提高胞嘧啶碱基编辑器(BE3和HF1-BE3)的特异性。(生物谷 Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


