Nat Struct Mol Biol | 西湖大学申恩志/宋春青/吴建平等合作揭示哺乳动物piRNA复合体的基因沉默新机制
来源:生物探索 2024-04-28 12:14
研究发现,与无脊椎动物的EfPiwi相比,MILI和HILI piwi表现出增强的靶标结合、更宽的靶标容量和强大的靶标切割。
西湖大学申恩志、宋春青及吴建平共同通讯在Nature Structural & Molecular Biology 在线发表题为“Mammalian PIWI-piRNA-target complexes reveal features for broad and efficient target silencing”的研究论文,该研究发现小鼠MILI和人类HILI piRNA诱导的沉默复合物(piRISCs)比来自sponge Ephydatia fluviatilis的无脊椎动物的对应物更有效地结合和切割靶标。在没有靶标的情况下,MILI和HILI piRISC采用更宽的核酸结合通道,与EfPiwi piRISC相比,它们显示出更长的预先排列的piRNA seed,这与它们比EfPiwi piRISC更有效地捕获靶标的能力是一致的。
在靶标存在的情况下,在哺乳动物piRISC中,加强microRNA RISC中seed-靶标保真度的seed门采用放松状态,揭示了MILI和HILI如何容忍seed-靶标错配以扩大靶标谱。脊椎动物特有的赖氨酸会扭曲piRNA seed,将piRNA靶双链的轨迹从中央间隙转移到PAZ叶。功能分析表明,这种赖氨酸促进了靶蛋白的结合和裂解。因此,该研究为小鼠和人类的piRNA靶向机制提供了分子基础,并表明哺乳动物的piRNA机制可以使用有限的piRNA物种来实现广泛的靶标沉默。

转座子,被称为基因组的“病原体”,能够在基因组内动员和复制自己。它们进化迅速,并通过促进新蛋白质和新活动的发展而成为基因组进化的强大力量。尽管有这些有益的作用,不受控制的转座子动员会导致遗传不稳定、不育和疾病。在动物中,PIWI蛋白及其小RNA向导(piRNAs)维持基因组完整性和生殖细胞发育,以确保生育能力和遗传信息的忠实传递。转座子—piRNA定向沉默的主要目标—在大多数哺乳动物基因组中占据很大一部分,而piRNA的源位点(称为piRNA簇)只占一小部分。例如,逆转录转座子至少占人类基因组的45%,但piRNA簇仅占人类基因组的~0.37% 。
相比之下,转座子到piRNA簇的含量在果蝇基因组中明显较低(转座子占14%,piRNA簇占3.2%)。piRNA沉默机制作为一种应对基因组内不断变化的转座子动力学的快速进化途径,可能与不同物种的宿主基因组寄生虫共同进化。因此,哺乳动物PIWI蛋白可能具有与无脊椎动物不同的靶向特征,以满足其保护哺乳动物种系基因组的作用。
对来自无脊椎动物(包括蚕、蝇)的PIWI蛋白的结构研究揭示了一种双叶结构,其中N-PAZ和MID-PIWI裂片形成了核酸结合通道的壁。这些无脊椎动物PIWI预先安排了一个短的seed片段,其结合亲和力低于靶向miRNA。此外,一个小α-螺旋(~1.5转)延伸了“种子”结构,拓宽了piRNA 种子区附近的通道,从而加强了种子匹配靶向,促进了piRNA -靶标碱基配对的后续传播,从而激活催化作用。虽然它们被认为是相似的,但脊椎动物的PIWI复合物,尤其是人类的PIWI复合物的结构尚未被报道。
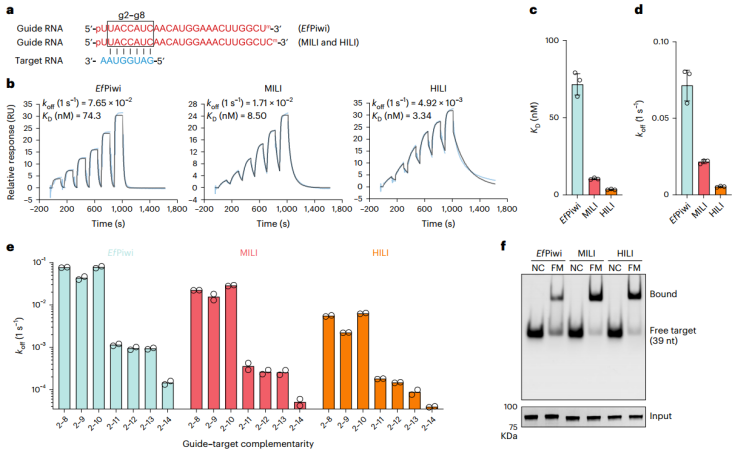
piRNAs靶结合在哺乳动物中比在无脊椎动物中更有效(Credit: Nature Structural & Molecular Biology)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


