国产纳米孔平台CycloneSEQ性能获独立研究验证:与ONT相比表现如何?
来源:华大智造 2026-01-22 17:35
近期,香港城市大学李润生团队在bioRxiv预印本平台发表了一项系统比较研究,题为《When CycloneSEQ meets Oxford Nanopore Technologies: a performance comparison of contemporary nanopore DNA sequencing platforms》(Liu et al.)。该研究通过六种细菌样本的全基因组测序数据,从读长准确率、组装完整性与表观基因组学应用等维度,客观对比了华大序风CycloneSEQ CS2(对应CycloneSEQ平台的S0 体系)与Oxford Nanopore Technologies(ONT)R10.4.1平台的性能差异。
核心结论显示:
CycloneSEQ 的测序数据Modal值达97.7%(Q16.4),经短读长校正后组装完整性接近100%,并能识别12种细菌甲基化基序。
研究指出,在关键性能指标上,CycloneSEQ已展现出与ONT相当的能力。
DOI:10.64898/2026.01.14.699587
纳米孔测序技术发展现状
纳米孔测序凭借其长读长、实时测序、直接检测表观遗传修饰等独特优势,在病原体快速鉴定、基因组完成图构建、耐药基因追踪等场景中展现出重要价值。长期以来,该领域由ONT主导。CycloneSEQ平台的出现,被视为中国在纳米孔测序领域实现自主知识产权的重要进展。
本研究是首个系统性、多维度比较CycloneSEQ与ONT平台性能的独立评估工作,既验证了CycloneSEQ平台的技术可行性,也为其在实际应用中的定位与发展提供了客观依据。
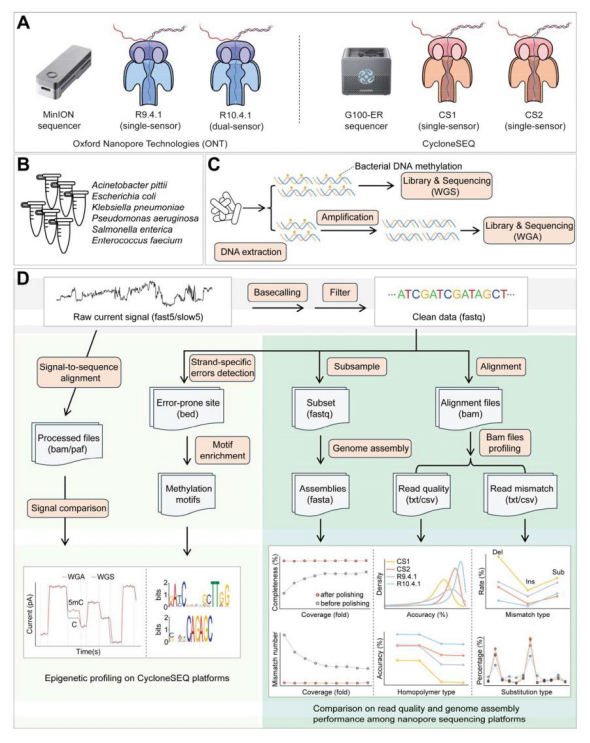
图:项目设计概览。
核心性能分析
reads准确率:CycloneSEQ CS2实现显著提升
数据表现:
· CycloneSEQ CS2:平均观测准确率96.0%(Q14.0),Modal 值 97.7%(Q16.4)
· ONT R10.4.1:平均观测准确率96.8%(Q14.9),Modal 值99.0%(Q20)
· 差距变化:与CS1版本相比,CS2将平均准确率差距缩小至0.8%。
技术解读:
这一进步得益于CycloneSEQ平台的文库制备和碱基识别算法的协同优化。研究指出,CycloneSEQ CS2的估计准确率与观测准确率之间的差距已收窄至0.39%,表明其碱基质量值(Q-score)校准更为可靠。这有助于在数据分析中更准确地依据原始Q值进行过滤。
同聚物区域性能:超越R9.4.1,逼近R10.4.1
关键发现:
同聚物(Homopolymer)区域的准确测序是纳米孔技术的一大挑战。研究表明CycloneSEQ CS2在A/T和G/C同聚物的识别准确性上表现优于ONT R9.4.1,接近R10.4.1水平。这对于精准解析细菌的相变基因、毒力岛等富含重复序列的区域具有重要的实际意义。
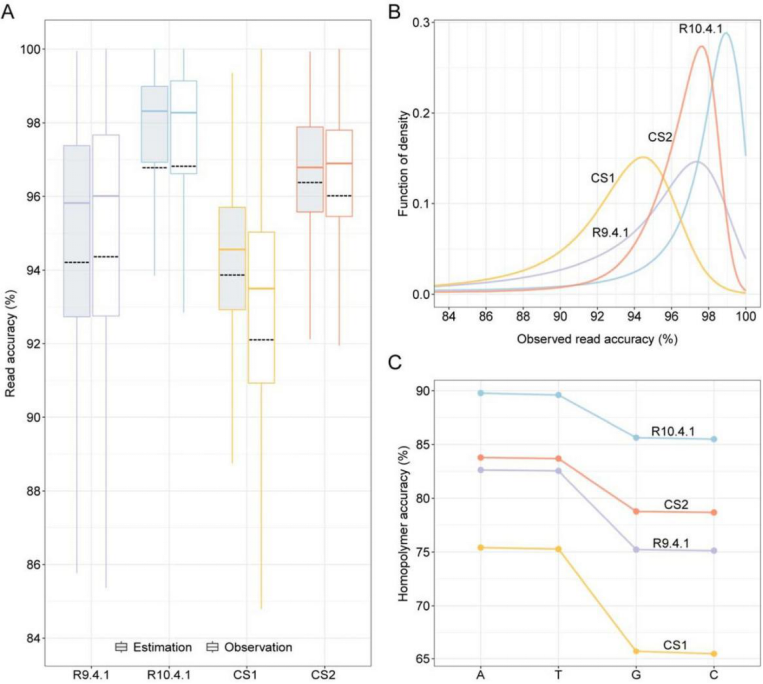
图:纳米孔测序平台之间的读段质量比较。
基因组组装性能:从"原始数据"到"完成图"的完整路径
原始长读长组装的挑战与解决方案
原始读长组装挑战:
CycloneSEQ原始读长的插入/缺失(InDel)错误率高于ONT,导致未校正前每100 kbp存在超过4个InDel。不过,通过结合约50倍覆盖度的长读长与短读长校正,可显著改善组装质量,获得近乎完整的细菌基因组。
校正后表现优异:
采用50×覆盖度结合短读长校正后:
· 组装完整性:提升至近100%(细菌染色体回收率>99.9%)
· BUSCO评估:稳定在97%完整性分数
· InDel密度:降至每100 kbp约1个,与ONT校正后水平相当
操作建议:
· 标准流程:推荐采用"CycloneSEQ长读长+ 短读长混合组装"策略。
· 成本控制:相较于纯ONT R10.4.1测序,CycloneSEQ在试剂成本上具有潜在优势,可将节省的资金投入短读长校正。
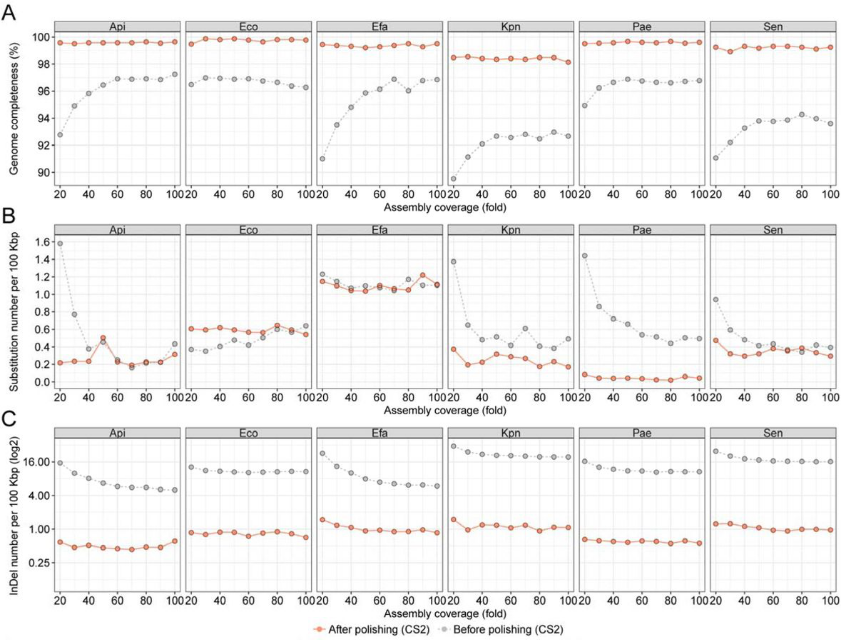
图:纳米孔测序平台的组装质量表现。
覆盖度优化策略
研究发现:
组装完整性在50×覆盖度后呈现边际效益递减,80×覆盖度下各平台(R9.4.1, R10.4.1, CS1, CS2)的BUSCO分数无显著差异。这为制定经济高效的测序方案提供了直接依据。
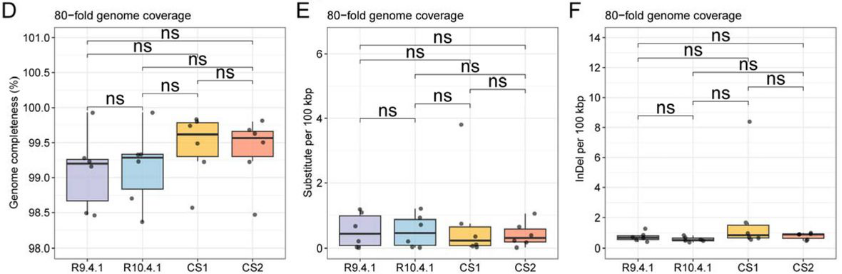
图:纳米孔测序平台的组装质量表现。
推荐方案:
· 常规菌株鉴定:30-50×覆盖度
· 高质量完成图:50-80×覆盖度 + 短读长polishing
· 复杂重复区域解析:可适当提高至100×
表观基因组学应用:甲基化检测的独特优势
检测能力与灵敏度
核心发现:
研究团队成功利用CycloneSEQ reads中存在的链特异性错误,从头发现了12种细菌DNA甲基化 motifs(methylation motifs),包括:
· 常见的GATC(Dam甲基化)、CCWGG(Dcm甲基化)
· 罕见的CTGCAG、GCGC等 motifs
· 潜在的新甲基化 motifs 发现能力
技术机制:
与ONT类似,甲基化碱基在纳米孔中产生的电流信号偏移导致碱基识别错误,通过比较正负链的测序错误模式,可高灵敏度定位甲基化位点。这证明了CycloneSEQ平台在无需额外实验处理的情况下,直接检测6mA、4mC等细菌常见甲基化修饰的能力。研究团队还为此开发了适配CycloneSEQ信号的分析流程,为表观基因组学研究打开了大门。
与ONT的性能对比
CycloneSEQ表现亮点:
· motifs覆盖广度:在部分菌株中识别出ONT未检测到的甲基化模式。
· 信号特征:独特的错误谱可能为新型表观遗传标记的发现提供线索。
待改进:
· CCWGG过拟合:CycloneSEQ碱基识别模型在该 motifs 上存在系统性偏差,导致假阳性率升高,需设置更严格的过滤阈值。
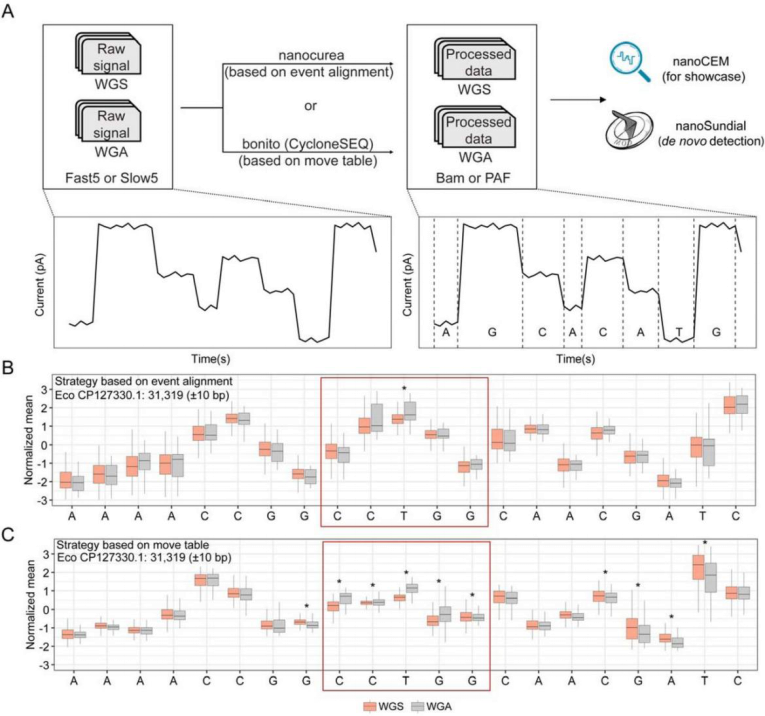
图:CycloneSEQ 平台检测到显著电流信号差异。
CycloneSEQ技术优势与改进方向
当前优势:
· 快速迭代能力:CycloneSEQ在数月内完成从CS1到CS2的更新,实现关键性能大幅提升。
· 成本效益:“50倍覆盖度+短读长校正”方案兼具质量与经济性。
待改进点方面:
· InDel错误率:研究指出单碱基插入缺失是CycloneSEQ reads中最主要的错误类型,这也是影响无需校正的组装质量的核心因素。
· 特定序列的过拟合:在甲基化 motifs CCWGG上,模型在无甲基化的WGA数据中产生了异常高的错误信号,表明基础模型存在对训练数据的过拟合。
· Q值校准精度:CycloneSEQ CS1版本存在估计准确率高于观测准确率的情况,CS2虽已改善,但校准精度仍有提升空间。
技术演进:CycloneSEQ S1体系展望
据悉,CycloneSEQ即将推出S1体系,涵盖试剂、算法与软件多方面的升级:
1. 试剂与化学优化:“精进”测序试剂盒(S1 体系)
· 准确率提升:通过优化马达蛋白与生化体系的协同作用,旨在将单碱基InDel错误率控制在更低的水平,原始测序准确率可达Q20以上。
· 增强的稳定性:改进的缓冲体系可提供更稳定的测序环境,显著提升有效数据产出率。
2. 算法与软件升级:“精进”碱基识别器
· 抗过拟合模型:采用更大规模、更多样化的训练数据集(包括更多种属的WGS与WGA数据),重点解决如CCWGG等特定 motifs 的过拟合问题,提升模型泛化能力。
· Q值校准:实现更精细的Q值校准,使质量分数能更真实地反映每个碱基的识别置信度。
· 同聚物优化:针对G/C同聚物识别准确率相对较低的问题,进行专项模型优化。
3.分析生态扩展:“拓展”分析套件(CycloneFlow)
通过集成自主开发的专用生信工具集,全面赋能长读长数据分析。该套件核心包括:长读长序列条码拆分工具 Citrus、长读长测序修饰检测数据分析工具 Poremod、长读长序列质控分析工具 Rosa 等。
未来展望
本研究是对CycloneSEQ技术路线的一次系统性验证,也是中国纳米孔测序产业化进程中的重要里程碑。尽管在原始读长准确率与生态系统成熟度方面仍有提升空间,但CycloneSEQ在组装后质量、表观遗传学应用与成本控制方面已展现出差异化竞争力。
从CycloneSEQ CS1到CS2(S0)的快速迭代,体现了华大序风团队的技术执行力;即将到来的S1体系,则进一步彰显了CycloneSEQ平台在性能优化与用户需求响应上的持续投入。CycloneSEQ有望为全球科研工作者,提供一个高性能、高性价比且自主可控的纳米孔测序选择,推动生命科学研究的深入发展。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


