2022年4月1日Science期刊精华
来源:生物谷原创 2022-04-07 10:29
本周又有一期新的Science期刊(2022年4月1日)发布,它有哪些精彩研究呢?让小编一一道来。
本周又有一期新的Science期刊(2022年4月1日)发布,它有哪些精彩研究呢?让小编一一道来。
1.Science:首次发布完整的人类基因组序列
doi:10.1126/science.abj6987
之前为全世界所称颂的人类基因组测序工作并不完整,因为当时的DNA测序技术无法读取人类基因组中的某些部分。即使在更新之后,它仍然缺少大约8%的人类基因组。在一项新的研究中,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员描述了有史以来第一次完整的人类基因组---一套构建和维持人类的指令---的测序。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“The complete sequence of a human genome”。

人类T2T-CHM13全基因组组装概述,图片来自Science, 2022, doi:10.1126/science.abj6987。
论文共同通讯作者、华盛顿大学研究员Evan Eichler说,“一些使我们成为独特的人类的基因实际上是在基因组的暗物质中,它们被完全遗漏了。花了20多年时间,但我们终于完成了。”Eichler参与了当前的研究工作和最初的人类基因组计划(Human Genome Project)。
科学家们指出人类基因组的全貌将使得对人类的进化和生物学特性有更多的了解,同时也为衰老、神经退行性疾病、癌症和心脏病等领域的医学发现打开大门。
2.Science:人类着丝粒的完整基因组和表观遗传图谱
doi:10.1126/science.abl4178
为了在细胞分裂过程中忠实地将遗传物质分配给子细胞,纺锤丝必须通过一种称为“动粒(kinetochore)”的结构与DNA结合,动粒在每条染色体的着丝粒(centromere)上组装。人类着丝粒位于一大串称为α卫星重复序列(alpha satellite, αSat)的串联重复DNA序列内,这些序列通常横跨每条染色体上的数百万个碱基对。一大串αSat序列经常被其他类型的功能不甚明了的串联卫星重复序列所包围,同时还有包括可发生转录的基因在内的非重复序列。以前的基因组测序工作由于其规模和重复性,无法产生富含卫星重复序列的区域的完整组装,从而限制了研究它们的分布、变异和功能的能力。
在过去的20年里,人类参考基因组中几乎完全没有着丝粒周缘区域和着丝粒(pericentromeric and centromeric, peri/centromeric)卫星DNA序列。在一项新的研究中,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员使用完整的、端粒到端粒(T2T)的人类基因组组装,开发并部署了定制的计算方法,以揭示这些卫星重复序列在大的和小的长度尺度上的分化和进化模式。他们还进行了一些实验,以精确绘制哪些αSat重复序列与动粒蛋白相互作用。最后,他们比较了多个个体之间的peri/centromeric区域,以了解这些序列在不同的遗传背景下是如何变化的。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“Complete genomic and epigenetic maps of human centromeres”。
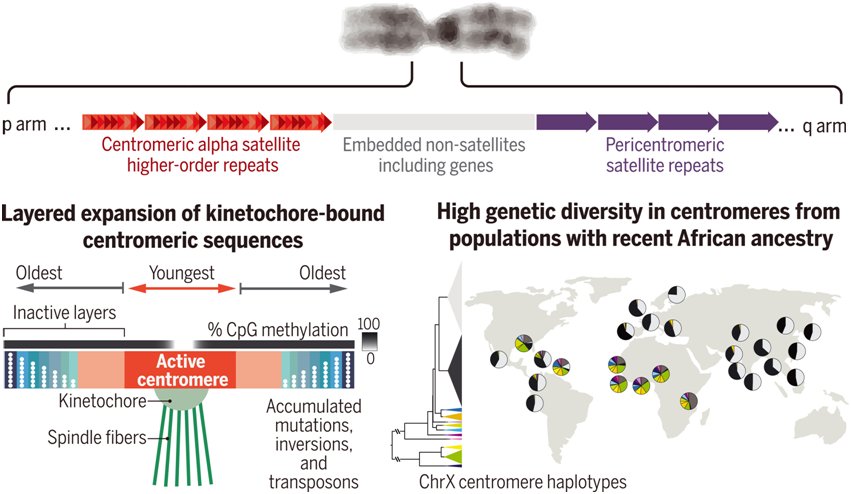
无间隙组装阐明着丝粒进化。图片来自Science, 2022, doi:10.1126/science.abl4178。
卫星重复序列占T2T-CHM13基因组的6.2%,其中αSat是最大的一个组成部分(占该基因组的2.8%)。通过对每个着丝粒的αSat的序列关系的详细研究,这些作者发现全基因组的证据表明人类着丝粒是通过“分层扩张(layered expansions)”进化的。具体来说,不同的重复序列变异出现在每个着丝粒区域内,并通过类似于连续串联重复(tandem duplication)的机制进行扩张,而较老的侧翼序列则随着时间的推移而缩小和分化。
这些作者还发现,每串αSat内最近扩张的重复序列更有可能与内部的动粒蛋白---着丝粒蛋白A(CENP-A)---相互作用,这与CpG甲基化降低的区域相吻合。这表明局部卫星重复序列扩张、动粒定位和DNA低甲基化之间有密切的关系。此外,他们发现了影响多种卫星重复序列类型的大型和意外的结构重排,包括活跃的着丝粒αSat。
3.Science:完整人类参考基因组改进了对人类遗传变异的分析
doi:10.1126/science.abl3533
人类参考基因组的核心应用之一是作为几乎所有人类基因组研究的比较基线。不幸的是,人类参考基因组的许多困难区域几十年来一直没有得到解决,并且受到塌陷区、序列缺失和其他问题的影响。相对于目前的人类参考基因组GRCh38,人类T2T-CHM13(Telomer-to-Telomere CHM13)基因组填补了所有剩余的空白,将这种基因组序列增加了近200 Mbp(Mbp),校正了数以千计的结构错误,并为科学探索释放了人类基因组中最复杂的区域。
在一项新的研究中,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员展示了T2T-CHM13参考基因组是如何在全球不同的队列中普遍改善读取序列映射和变异识别的。这个队列包括来自扩大的1000基因组计划(1KGP)的经过短读测序的所有3202个样本,以及17个经过长读测序的全球不同样本。通过应用最先进的方法来调用单核苷酸变异(SNV)和结构变异(SV),他们记录了T2T-CHM13相对于之前的人类参考基因组序列的优势和局限性,并强调它有望在基因组的技术挑战区域内揭示新的生物学见解。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“A complete reference genome improves analysis of human genetic variation”。
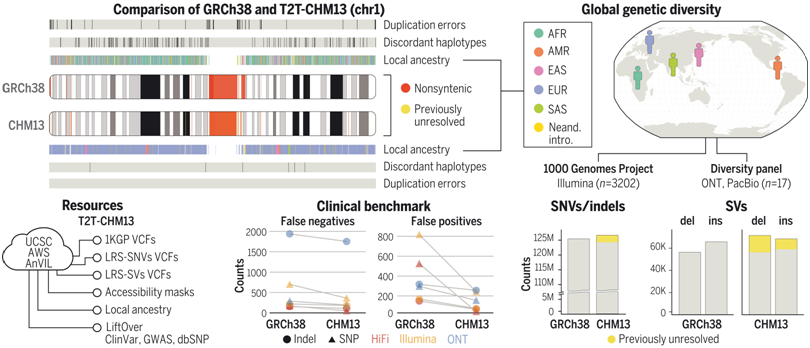
T2T-CHM13的基因组特征和可用资源。图片来自Science, 2022, doi:10.1126/science.abl3533。
在1KGP样本中,与GRCh38相比,这些作者在全基因组范围内利用T2T-CHM13发现了超过100万个额外的高质量变异。在以前未解决的基因组区域,他们在每个样本中发现了几十万个变异---这是进化和生物医学发现的好机会。T2T-CHM13提高了602个三人组之间的孟德尔一致率,并消除了每个样本数以万计的假性SNV,包括将269个具有挑战性的医学相关基因的假阳性率降低了12倍。这些校正在很大程度上是由于在GRCh38中由错误塌陷区域或重复区域引起的长达9 Mbp以上的不准确序列中对70个蛋白编码基因的改进。通过使用T2T-CHM13参考基因组还可以在全基因组范围内更好地了解结构变异,大大改善了序列插入和缺失之间的平衡。最后,通过为T2T-CHM13提供大量的资源(包括1KGP基因型、可访问性掩码和突出的注释数据库),他们的研究工作将促进从目前的人类参考基因组向T2T-CHM13过渡。
4.Science:揭示完整人类基因组中重复序列的转录和表观遗传状态
doi:10.1126/science.abk3112
在一项新的研究中,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员实施了一个全面的重复序列注释工作流程,使用以前已知的人类重复序列和从头重复序列建模,然后进行人工整理,包括利用基因注释评估重叠,分段复制,串联重复序列和经过注释的重复序列。通过使用这种方法,他们开发出一个更新的人类重复序列目录,并完善了以前的重复序列注释。他们在T2T-CHM13中发现了43个以前未知的重复序列和重复序列变异(repeat variant),并表征了19个复杂的经常携带基因的复合重复序列结构。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“From telomere to telomere: The transcriptional and epigenetic state of human repeat elements”。
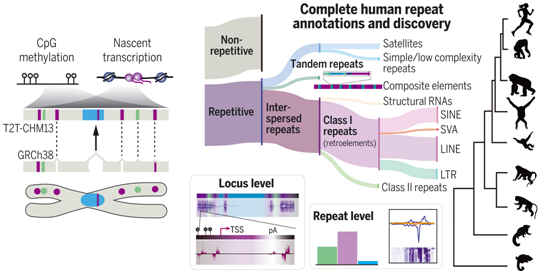
CHM13的端粒到端粒组装支持重复序列注释和发现。图片来自Science, 2022, doi:10.1126/science.abk3112。
通过使用精确的PRO-seq(precision nuclear run-on sequencing)技术和牛津纳米孔技术公司长读测序数据产生的CpG甲基化位点,这些作者在全基因组范围内评估了逆转录因子中的RNA聚合酶结合,揭示了新生转录、序列分歧、CpG密度和甲基化之间的相关性。他们将这些分析扩展到评估所有重复序列的RNA聚合酶占有率,包括位于所有人类染色体的以前无法进入的着丝粒中的高密度卫星重复序列。
此外,在早期发育阶段和完整的细胞周期时间序列中,这些作者使用依赖图谱和不依赖图谱的方法,发现整个卫星重复序列中的RNA聚合酶占有率很低;相反,转座因子转录很丰富,并作为CpG甲基化和着丝粒亚结构变化的一个界限。这些数据共同揭示了转录活跃的逆转录因子和DNA甲基化之间的动态关系,以及新的重复序列家族和复合重复序列的衍生和进化的潜在机制。
通过关注新兴的HG002 X染色体的端粒到端粒组装,这些作者揭示了人类群体可能存在高水平的重复序列变异,包括影响基因拷贝数的复合重复序列拷贝数。此外,他们强调了重复序列对基因组结构多样性的影响,揭示了人类和灵长类动物之间拷贝数差异极大的重复序列扩张,同时也对逆转录因子转导事件进行了高置信度的注释。
5.Science:揭示完整人类基因组中的片段重复及其变异
doi:10.1126/science.abj6965
较大的、高同一性的重复序列---被称为片段重复(segmental duplication, SD)---通常是基因组中最后被测序和组装的区域。虽然人类参考基因组为构建SD景观提供了路线图,但是这种基因组中剩余的50%以上的空白对应于复杂的SD区域。SD是进化基因创新的主要来源,对类人猿物种内部和之间的遗传变异起着不成比例的作用。有了完整的人类基因组:T2T-CHM13,科学家们就有可能识别基因并发现人类的遗传变异模式。
在一项新的研究中,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员在T2T-CHM13中发现了51 Mbp(million base pair,百万个碱基对)的额外人类SD,如今估计人类基因组的7%由SD组成,即在总共31亿个碱基对的人类基因组中,SD共有218 Mbp。SD占近端着丝的短臂的三分之二(68.1Mbp中的45.1Mbp),这些SD是人类基因组中最大的(见图中的A部分)。此外,54%的近端着丝SD的拷贝数是可变的,或者在被研究的六个人中映射到不同的染色体上。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“Segmental duplications and their variation in a complete human genome”。
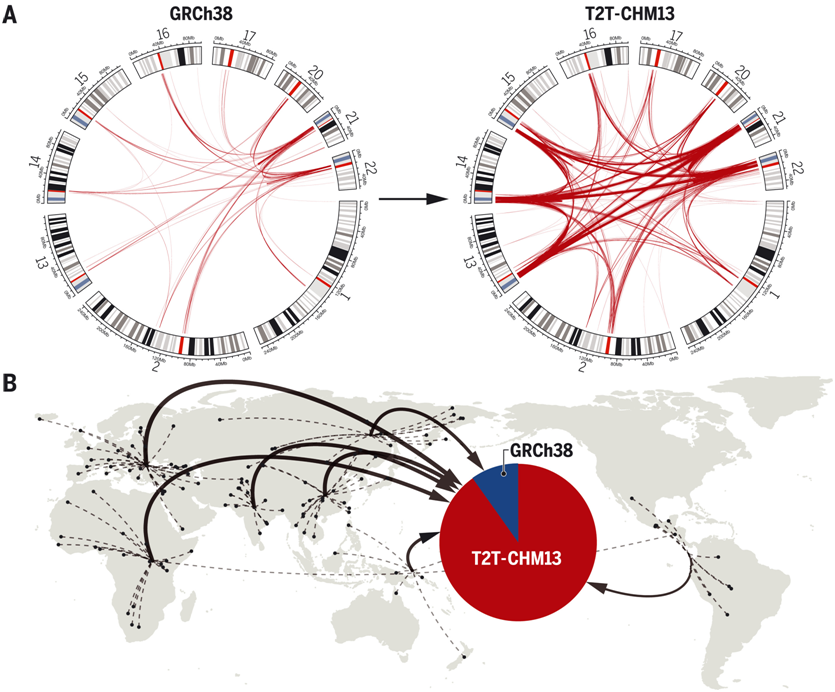
更完整的片段重复改善了基因分型。图片来自Science, 2022, doi:10.1126/science.abj6965。
对目前参考基因组(GRCh38)和T2T-CHM13的SD含量进行详细比较,发现有81Mbp的以前未解决的或结构可变的SD。来自一个由268个人组成的多样性小组的短读全基因组序列数据显示,人类拷贝数与T2T-CHM13相匹配的可能性为与GRCh38匹配的9倍(前者为59.26 Mbp,后者为6.55Mbp),包括119个蛋白编码基因(见图中的B部分)。
利用来自25个人类单倍型的长读测序数据,这些作者调查了人类遗传变异的模式,发现结构和单核苷酸多样性的显著增加。他们确定了基因丰富的区域(比如TBC1D3),这些区域在个体之间的差异达数十万个碱基对和基因拷贝数,显示了一些最高的全基因组结构杂合度(85%至90%)。
6.Science:分析完整人类基因组中的表观遗传模式
doi:10.1126/science.abj5089
人类参考基因组已成为许多大规模计划的基础,包括对表观基因组(epigenome)进行编目的集体努力,其中表观基因组是控制基因活动和细胞功能的一组标记和蛋白相互作用。然而,二十多年来,构建完整表观基因组的努力一直受到不完整人类参考基因组的阻碍。
随着近期技术的进步,来自端粒到端粒(Telomere-to-Telomere, T2T)联盟的研究人员如今可以通过一个完整的人类基因组端粒到端粒组装:T2T-CHM13,全面研究人类基因组的结构和功能。因此,人们如今可以拓宽人类表观基因组,包括2.25亿个碱基对的额外序列。相关研究结果发表在2022年4月1日的Science期刊上,论文标题为“Epigenetic patterns in a complete human genome”。
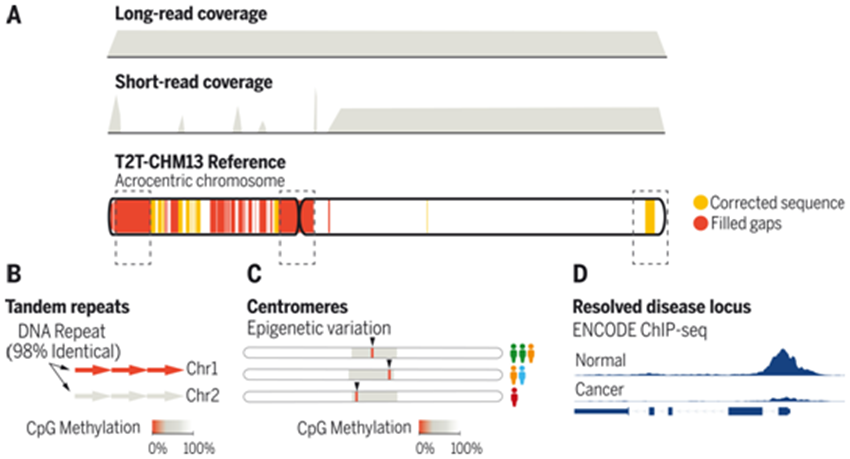
描述整个人类基因组的表观遗传特征。图片来自Science, 2022, doi:10.1126/science.abj5089。
表观基因组是指DNA修饰(如CpG甲基化)、蛋白-DNA相互作用、组蛋白修饰和染色质结构,它们共同影响基因表达、基因组调节和基因组稳定性。这些表观遗传特征在细胞分裂时是可遗传的,但在发育过程中是动态的,产生了不同组织和细胞类型所特有的模式。在这项新的研究中,这些作者提出了对人类基因组的表观遗传学注释,比如他们探索了以前未解决的区域,包括近端着丝的染色体短臂、片段重复的基因(segmentally duplicated genes)以及包括人类着丝粒在内的各种重复序列类别。对以前缺失的8%的人类基因组产生完整的表观遗传学注释,为阐明这些基因组元件的功能作用提供了基础,这对人们理解基因组的调节、功能和进化至关重要。
7.Science:青铜时代和铁器时代的人口流动是新疆人口历史的基础
doi:10.1126/science.abk1534
中国的新疆地区与山脉接壤,是一个重要的历史区域。在一项新的研究中,我们科学家对古代基因组进行了取样,研究了该地区从青铜时代(距今约5000年至3000年)到铁器时代(距今约3000年至2000年),再到进入历史时代(距今约2000年)的人口变化。这一分析表明,较年长的个体代表了来自草原文化的祖先,后来东亚和中亚的祖先在青铜时代末期和铁器时代初期进入该地区。在历史时期,混合继续进行,但保留了核心的草原成分,使人口形成了一个遗传连续体。在一个中心人群中保留这种遗传连续性是令人惊讶的,因为它代表了在孤立人群中更典型的观察模式。此外,这些遗传联系确定了一个以前未知的谱系,有可能解释印欧语系的传播。
8.Science:一个关键基因是实验性食物网持续存在的基础
doi:10.1126/science.abf2232
在过去的几十年里,关键物种的识别,即那些在构建一个群落或生态系统中具有重要作用的物种,在不同的系统中都有增加。Barbour等人将这一概念扩展到了基因上,表明一个特定植物防御基因的单个等位基因促进了整个小型实验营养系统的物种共存。具体来说,具有这个等位基因的植物生长得更快,支持两种食草动物及其捕食者的更大种群。这一发现表明,基因型变异可以在有机体系统的结构和功能中发挥作用。
9.Science:探究哺乳动物的脑化率
doi:10.1126/science.abl5584
在脊椎动物中,哺乳动物的大脑与身体大小的比例最大(脑化)。人们一直认为,这种关系在哺乳动物进化的早期就出现了,扩大的大脑引领着它们进入新的和多样化的形式。然而,Bertrand等人研究了从古新世开始的所有哺乳动物的脑化率,发现身体尺寸首先增加,以便在恐龙灭绝后进行生态位填充。只是到了后来,即始新世,大脑大小才开始增加,这可能是由于在日益复杂的环境中需要有更大的认知能力。这导致了今天的高度脑化的大脑,包括人类的大脑。
10.Science:一个脊髓小胶质细胞群体参与缓解性和复发性神经疼痛
doi:10.1126/science.abf6805
对神经系统的损害从病理上改变了躯体感觉系统,这可能导致神经性疼痛的发生。虽然疼痛产生已经得到了很好的研究,但协调疼痛恢复的机制仍然不清楚。Kohno等人在神经损伤的小鼠中发现了一个CD11c+脊髓小胶质细胞群体在疼痛产生后出现,对神经病理性疼痛的恢复至关重要。这些细胞促进疼痛恢复的能力依赖于它们高水平的胰岛素样生长因子-1(IGF1)的表达。即使在疼痛恢复后,CD11c+小胶质细胞仍然存在,如果它们遭受耗竭,疼痛的超敏性就会恢复。这些发现阐明了缓解性和复发性神经疼痛的机制,并可能有助于治疗策略的开发。
11.Science:在实验人群中,选择性社会学习保留了复杂的认知算法
doi:10.1126/science.abn0915
我们世代积累复杂算法的能力使人类能够适应不同的环境,并解决超出我们个人限制的挑战。然而,创新算法的文化积累是难以解释的。Thompson等人研究了大量的参与者,以探索不同学习条件下的算法演化。涉及不同策略或不同模型的成功水平知识的选择性社会学习比随机社会学习或一次尝试的非社会学习更能保存难以发明的高效算法。有两种有效的算法被很多人使用,但最有效的算法只在选择性的社会学习下传播。(生物谷 Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


