Cell重大突破:AI从超1亿个分子中预测强力抗生素,杀伤超级耐药细菌
来源:本站原创 2020-02-26 10:09
2020年2月26日讯 /生物谷BIOON /--一项开创性的机器学习方法已经从1亿多个分子中识别出了强大的新型抗生素,包括一种可以对付多种细菌的分子--包括肺结核和被认为无法治愈的菌株。研究人员表示,这种名为halicin的抗生素是第一个被人工智能发现的抗生素。尽管人工智能以前曾被用于协助抗生素发现过程的某些部分,但他们表示,这是人工智能首次在不使用任何人
研究人员表示,这种名为halicin的抗生素是第一个被人工智能发现的抗生素。尽管人工智能以前曾被用于协助抗生素发现过程的某些部分,但他们表示,这是人工智能首次在不使用任何人类假设的情况下,从零开始识别出全新种类的抗生素。这项研究由剑桥麻省理工学院的合成生物学家Jim Collins领导,发表在《Cell》杂志上。
宾夕法尼亚匹兹堡大学的计算生物学家Jacob Durrant说,这项研究意义非凡。他说,研究小组不仅确定了候选分子,还在动物实验中验证了有希望的分子。更重要的是,这种方法也可以应用于其他类型的药物,如用于治疗癌症或神经退行性疾病的药物。
图片来源:Cell
细菌对抗生素的耐药性在全球范围内急剧上升,研究人员预测,除非紧急开发新的药物,否则到2050年,耐药感染每年可能导致1000万人死亡。但在过去的几十年里,新抗生素的发现和监管审批都有所放缓。"人们不断地发现同样的分子," Collins说。"我们需要新的化学反应和新的作用机制。"
忘记你的假设
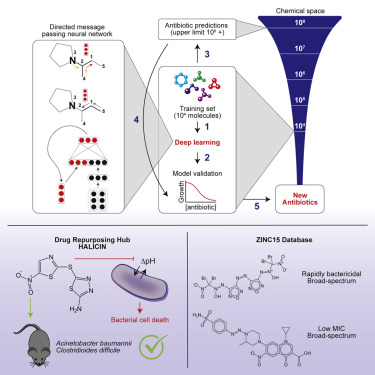
Collins和他的团队开发了一个神经网络--一种受大脑结构启发的人工智能算法--一个原子一个原子地学习分子的特性。
研究人员利用已知的2335个抗菌分子,训练其神经网络识别抑制大肠杆菌生长的分子。这包括一个包含约300种经批准的抗生素的文库,以及800种来自植物、动物和微生物来源的天然产品。
麻省理工学院(MIT)的人工智能研究员、该研究合作者Regina Barzilay表示,该算法学会了在不考虑药物作用方式和化学基团标签的情况下预测分子功能。"因此,该模型可以学习人类专家不知道的新模式。"
一旦这个模型被训练好,研究人员就用它来筛选一个叫做"药物再利用中心"的库,这个库包含了大约6000个正在被研究的用于人类疾病的分子。他们利用算法预测哪种抗生素对大肠杆菌有效,并只向他们展示与传统抗生素不同的分子。
从这些结果中,研究人员选择了大约100个候选分子进行动物实验。结果发现其中一种被用作糖尿病治疗的分子被证明是一种有效的抗生素,他们用电影《2001: A Space Odyssey》中的智能电脑HAL来命名这种抗生素,并将其命名为halicin。在小鼠实验中,这种分子对多种病原体都有活性,包括一株艰难梭状芽胞杆菌和一株鲍曼不动杆菌。鲍曼不动杆菌具有"泛耐药性",急需开发新的抗生素。
质子流
抗生素通过一系列的机制起作用,如阻断参与细胞壁生物合成、DNA修复或蛋白质合成的酶。但halicin的机制是非常规的:它破坏了细胞膜上的质子流动。在最初的动物试验中,它似乎也具有低毒和抗抗性。在实验中,对其他抗生素化合物的耐药性通常在一两天内出现,Collins说。"但即使经过30天的测试,我们也没有发现对halicin的任何耐药性。"
该团队随后在一个名为ZINC15的数据库中筛选了超过1.07亿个分子结构。在23项测试中,有8项被确定具有抗菌活性。其中两种对多种病原体都有很强的活性,甚至可以战胜对抗生素有抗药性的大肠杆菌。
图片来源:Stephanie Schuller SPL
匹兹堡卡耐基梅隆大学的计算生物学家Bob Murphy说,这项研究是"利用计算方法发现和预测潜在药物特性的大量工作的一个很好的例子"。他指出,人工智能方法之前已经被开发出来,挖掘巨大的基因和代谢物数据库,以识别可能包括新型抗生素在内的分子类型。
但是Collins和他的团队说,他们的方法是不同的--他们不是搜索特定的结构或分子类别,而是训练他们的网络寻找具有特定活动的分子。该团队现在希望与外部团体或公司合作,让halicin进入临床试验。他们还想拓宽方法,发现更多新的抗生素,从零开始设计分子。Barzilay说他们这个最新的工作是一个概念证明的工作。"这项研究将所有这些结合起来,并证明了它的作用。"(生物谷Bioon.com)
参考资料:
【1】Jo Marchant. Powerful antibiotics discovered using AI. Natrue.2020. doi: 10.1038/d41586-020-00018-3
【2】Jonathan M. Stokes et al., (2020), A Deep Learning approach to Antibiotic Discovery, Cell, DOI: https://doi.org/10.1016/j.cell.2020.01.021
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->