Science:开发出一种检测CRISPR脱靶效应的新方法---DISCOVER-Seq
来源:本站原创 2019-04-20 23:54
2019年4月20日讯/生物谷BIOON/---自从CRISPR基因组编辑技术于2012年发明以来,它已经显示出治疗许多难治性疾病的巨大希望。然而,科学家们一直在努力在治疗相关的细胞类型中鉴定潜在的脱靶效应,这仍然是将治疗方法转移到临床应用的主要障碍。如今,在一项新的研究中,来自美国加州大学伯克利分校、加州大学旧金山分校、格拉德斯通研究所和瑞典阿斯利康公司的研究人员开发出一种可靠的方法来实现这一目
2019年4月20日讯/生物谷BIOON/---自从CRISPR基因组编辑技术于2012年发明以来,它已经显示出治疗许多难治性疾病的巨大希望。然而,科学家们一直在努力在治疗相关的细胞类型中鉴定潜在的脱靶效应,这仍然是将治疗方法转移到临床应用的主要障碍。如今,在一项新的研究中,来自美国加州大学伯克利分校、加州大学旧金山分校、格拉德斯通研究所和瑞典阿斯利康公司的研究人员开发出一种可靠的方法来实现这一目标。相关研究结果发表在2019年4月19日的Science期刊上,论文标题为“Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq”。论文通讯作者为加州大学伯克利分校的Jacob E. Corn。论文第一作者为加州大学伯克利分校的Beeke Wienert和Stacia Wyman。
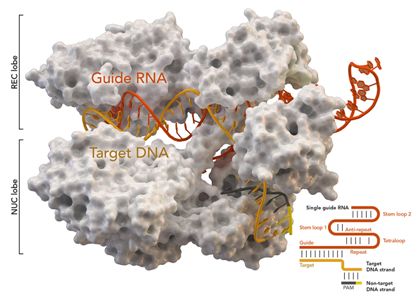
CRISPR通过在特定位置切割DNA来编辑人的基因组。所面临的挑战是确保这种工具不会在其他地方进行切割,即一种被称为“脱靶效应”的DNA损伤,这可能会带来无法预料的后果。
Wienert博士说,“当CRISPR进行切割时,DNA会被破坏。因此,为了生存,细胞将许多不同的DNA修复因子募集到基因组中的特定位点上以修复断裂并将切割末端连接在一起。我们认为如果我们能够找到这些DNA修复因子的位置,我们就可以鉴定出被CRISPR切割的位点。”
为了测试这种想法,这些研究人员研究了一组不同的DNA修复因子。他们发现其中的一种称为MRE11的DNA修复因子是DNA切割位点的第一批响应者之一。他们利用MRE11开发了一种名为DISCOVER-Seq的新技术,它可以识别出CRISPR切割基因组的确切位点。
论文共同作者、格拉德斯通研究所高级研究员Bruce R. Conklin博士解释道,“人类基因组非常庞大---如果你打印完整的人DNA序列,那么你最终会得到一本高达16层的小说。当我们想用CRISPR切割DNA时,这就像我们试图删除这本小说中特定页面上的一个特定单词一样。”
Conklin补充道,“你可以将DNA修复因子视为给这本书添加的不同类型的书签。虽然一些DNA修复因子可能会将整个章节作为书签,但是MRE11却是一个能够精确到这本书中已发生变化的字母。”
目前存在检测CRISPR脱靶效应的不同方法。然而,它们具有一些不足之处,比如产生假阳性结果和杀死它们正在检查的细胞。此外,迄今为止最常用的方法仅限于在实验室中用于体外培养的细胞,但不包括它在患者来源的干细胞或动物组织中的使用。
Corn说,“鉴于我们的方法依赖于细胞的自然修复过程来识别切割位点,它经证实是一种侵入性更小、更可靠的方法。我们能够在诱导性多能干细胞、患者细胞和小鼠中测试我们新开发的DISCOVER-Seq方法,而且我们的研究结果表明这种方法可潜在地用于任何系统,而不仅仅是在实验室中。”
这种正在用于新的细胞类型和系统中的DISCOVER-Seq方法也揭示出对CRISPR编辑基因组机制的新认识,这将导致更好地理解这种工具如何发挥作用的生物学特性。
Conklin说,“这种新方法极大地简化了识别脱靶效应的过程,同时也提高了结果的准确性。这可能让我们更好地预测基因组编辑如何在临床环境中发挥作用。因此,它代表了改善临床前研究和让基于CRISPR的疗法更接近有需要的患者的一个重要步骤。”(生物谷 Bioon.com)
参考资料:
Beeke Wienert et al. Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq. Science, 2019, doi:10.1126/science.aav9023.
Hannah R. Kempton et al. When genome editing goes off-target. Science, 2019, doi:10.1126/science.aax1827.

图片来自Thomas Splettstoesser (Wikipedia, CC BY-SA 4.0)。
Wienert博士说,“当CRISPR进行切割时,DNA会被破坏。因此,为了生存,细胞将许多不同的DNA修复因子募集到基因组中的特定位点上以修复断裂并将切割末端连接在一起。我们认为如果我们能够找到这些DNA修复因子的位置,我们就可以鉴定出被CRISPR切割的位点。”
为了测试这种想法,这些研究人员研究了一组不同的DNA修复因子。他们发现其中的一种称为MRE11的DNA修复因子是DNA切割位点的第一批响应者之一。他们利用MRE11开发了一种名为DISCOVER-Seq的新技术,它可以识别出CRISPR切割基因组的确切位点。
论文共同作者、格拉德斯通研究所高级研究员Bruce R. Conklin博士解释道,“人类基因组非常庞大---如果你打印完整的人DNA序列,那么你最终会得到一本高达16层的小说。当我们想用CRISPR切割DNA时,这就像我们试图删除这本小说中特定页面上的一个特定单词一样。”
Conklin补充道,“你可以将DNA修复因子视为给这本书添加的不同类型的书签。虽然一些DNA修复因子可能会将整个章节作为书签,但是MRE11却是一个能够精确到这本书中已发生变化的字母。”
目前存在检测CRISPR脱靶效应的不同方法。然而,它们具有一些不足之处,比如产生假阳性结果和杀死它们正在检查的细胞。此外,迄今为止最常用的方法仅限于在实验室中用于体外培养的细胞,但不包括它在患者来源的干细胞或动物组织中的使用。
Corn说,“鉴于我们的方法依赖于细胞的自然修复过程来识别切割位点,它经证实是一种侵入性更小、更可靠的方法。我们能够在诱导性多能干细胞、患者细胞和小鼠中测试我们新开发的DISCOVER-Seq方法,而且我们的研究结果表明这种方法可潜在地用于任何系统,而不仅仅是在实验室中。”
这种正在用于新的细胞类型和系统中的DISCOVER-Seq方法也揭示出对CRISPR编辑基因组机制的新认识,这将导致更好地理解这种工具如何发挥作用的生物学特性。
Conklin说,“这种新方法极大地简化了识别脱靶效应的过程,同时也提高了结果的准确性。这可能让我们更好地预测基因组编辑如何在临床环境中发挥作用。因此,它代表了改善临床前研究和让基于CRISPR的疗法更接近有需要的患者的一个重要步骤。”(生物谷 Bioon.com)
参考资料:
Beeke Wienert et al. Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq. Science, 2019, doi:10.1126/science.aav9023.
Hannah R. Kempton et al. When genome editing goes off-target. Science, 2019, doi:10.1126/science.aax1827.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


