PNAS:开发出超灵敏的SHERLOCK疟疾测试方法
来源:本站原创 2020-10-07 10:25
2020年10月7日讯/生物谷BIOON/---为了实现世界卫生组织(WHO)全球疟疾控制方案确定的消除疟疾的目标,必须在确定的地理区域内消除疟原虫的所有局部传播。实现这一目标的一个重要基石是开发快速、灵敏和物种特异性的诊断能力,这些能力在许多疟疾流行地区的低资源环境中是有用的。目前,四种主要的致疟原虫物种---恶性疟原虫、间日疟原虫、卵形疟原虫和三日疟原虫
2020年10月7日讯/生物谷BIOON/---为了实现世界卫生组织(WHO)全球疟疾控制方案确定的消除疟疾的目标,必须在确定的地理区域内消除疟原虫的所有局部传播。实现这一目标的一个重要基石是开发快速、灵敏和物种特异性的诊断能力,这些能力在许多疟疾流行地区的低资源环境中是有用的。
目前,四种主要的疟原虫物种---恶性疟原虫、间日疟原虫、卵形疟原虫和三日疟原虫---的存在是通过对血液样本的显微镜分析来确定的,在血液样本的红细胞中可以检测到疟原虫,或者通过所谓的快速诊断测试来确定特定的疟原虫蛋白(抗原)。
美国波士顿儿童医院传染病诊断实验室副医学主任、哈佛医学院病理学与医学副教授Nira Pollock博士说,“不幸的是,现有的快速诊断方法不能将所有四种疟原虫区分开来,这对启动针对性的治疗过程很重要,最重要的是,它们无法有效地检测无症状个体中的低数量疟原虫。” 哈佛医学院儿科副教授、波士顿儿童医院传染病高级副医师Jeffrey Dvorin医学博士补充道,“这些‘无症状携带者(asymptomatic carrier)’是传播疟疾的蚊子持续传播这种疾病的潜伏库,对于正在进行的全球消除疟疾的努力极为重要。”
如今,在一项新的研究中,Pollock及其同事们开发出一种可现场应用的超灵敏诊断测试方法,可特异性地检测有症状和无症状疟疾患者体内所有疟原虫物种的DNA序列。这种新的疟疾诊断方法将优化的10分钟快速样品制备方案与基于CRISPR的SHERLOCK系统相结合,在简单的报告装置中,再过60分钟就能实现高度特异性和灵敏度的疟原虫检测。相关研究结果近期发表在PNAS期刊上,论文标题为“Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria”。
论文通讯作者、哈佛医学院怀斯生物启发工程研究所创始核心成员James Collins博士说,“这种可用于现场的SHERLOCK疟疾检测方法超越了世界卫生组织设定的理想测试方法的灵敏度和特异性要求,可用于检测所有主要疟原虫物种的无症状携带者中的低密度疟原虫。它的高度精简的设计可以为目前消除疟疾道路上的诊断瓶颈提供一种可行的解决方案,以便更广泛地实现低资源环境下的疟疾监测。”
这些研究人员证实他们的经过改造的SHERLOCK(Specific High-sensitivity Enzymatic Reporter unLOCKing)测试方法能够检测到每微升血液中少于2个疟原虫,这是世界卫生组织建议的在流行地区具有广泛实用性的测试方法的 “检测极限(limit of detection, LOD)”。通过分析含有恶性疟原虫和间日疟原虫物种的临床样本,他们能够以100%的灵敏度、100%的特异性识别出真正的阳性样本,并在真正的阴性样本中正确识别出缺少某种疟原虫物种的样本,从而显示出这种测试方法的临床潜力。接近100%的灵敏度和特异性是诊断测试方法在实际测试中使用的关键特性。此外,所设计的这种测试方法还可以确定经常发生突变的恶性疟原虫虫株的存在,这些虫株因发生突变而失去了HRP2抗原,因而逃避了常规快速诊断测试方法的检测。
Collins在怀斯生物启发工程研究所和麻省理工学院的研究团队与布罗德研究所的Feng Zhang团队共同开发了SHERLOCK技术。它被授权给了初创公司Sherlock Biosciences公司,该公司利用它为其他疾病的应用开发了一种快速分子诊断方法。近期,该公司开发的基于SHERLOCK的COVID-19诊断方法获得了FDA的紧急使用授权。
其他的方法已经被开发出来,比如这种新的SHERLOCK测试方法,可扩增和检测疟原虫的DNA(或RNA)核酸分子。然而,迄今为止,这些方法仍然受限于它们需要昂贵的实验设备(就像基于聚合酶链反应的方法那样)、复杂的样品制备技术和训练有素的人员,或者在在简单的情况下,“等温”扩增方法在同一个温度下执行,但是它们没有实现在现场测试中所需的灵敏度。
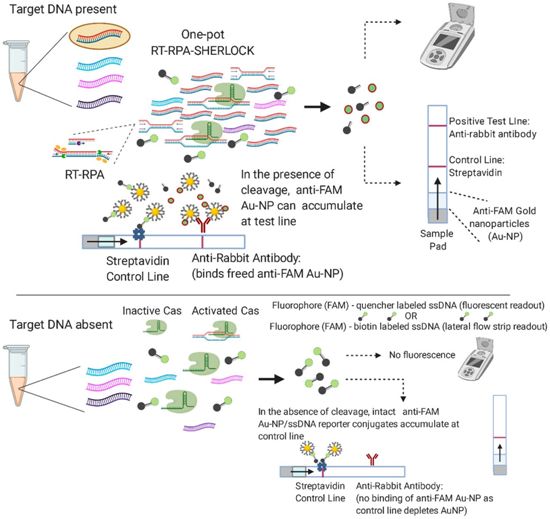
这种SHERLOCK疟疾测试方法利用了CRISPR-Cas12a酶,该酶经编程后可被结合到靶核酸序列上的向导RNA(gRNA)活化,就这项研究而言,靶核酸序列来自这四种疟原虫之一。活化的Cas12a然后非特异性地切割它附近的任何单链DNA链,而且是每秒发生大约1250次切割反应。这些研究人员在他们的测试方法中利用了这种扩增活性,将它与优化的样品制备相结合,不需要像其他一些核酸扩增测试方法那样进行特定的核酸提取步骤,并在前端对特定的疟原虫DNA和RNA序列进行等温扩增。识别扩增的疟原虫序列中物种特异性基因的gRNA随后激活Cas12a的活性,这样这种酶就会附带地攻击单链DNA报告序列,这种报告序列的切割产物有助于发出指示病原体特异性核酸存在的信号。在这种测试方法的后端,这种信号可被设计为手持设备中的荧光变化,或者在及时测试(POCT)设备中常用的侧向流测试条上的特定色带。
论文第一作者、Collins团队成员Rose Lee说,“重要的是,这种测试方法可兼容不同的样本类型,如全血、血浆、血清和干血,而且用于扩增、Cas12a活化和信号产生所需的所有成分都可以在单一试管中冻干,在它们与患者样本溶解和混合后,可发生一锅化反应(one-pot-reaction)。这避免了必须依赖功能性的冷链,并允许在低资源环境下以最少的专业知识进行测试。”(生物谷 Bioon.com)
参考资料:
1.Rose A. Lee el al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. PNAS, 2020, doi:10.1073/pnas.2010196117.
2.CRISPR-based malaria testing on-the-fly
https://medicalxpress.com/news/2020-09-crispr-based-malaria-on-the-fly.html
目前,四种主要的疟原虫物种---恶性疟原虫、间日疟原虫、卵形疟原虫和三日疟原虫---的存在是通过对血液样本的显微镜分析来确定的,在血液样本的红细胞中可以检测到疟原虫,或者通过所谓的快速诊断测试来确定特定的疟原虫蛋白(抗原)。
美国波士顿儿童医院传染病诊断实验室副医学主任、哈佛医学院病理学与医学副教授Nira Pollock博士说,“不幸的是,现有的快速诊断方法不能将所有四种疟原虫区分开来,这对启动针对性的治疗过程很重要,最重要的是,它们无法有效地检测无症状个体中的低数量疟原虫。” 哈佛医学院儿科副教授、波士顿儿童医院传染病高级副医师Jeffrey Dvorin医学博士补充道,“这些‘无症状携带者(asymptomatic carrier)’是传播疟疾的蚊子持续传播这种疾病的潜伏库,对于正在进行的全球消除疟疾的努力极为重要。”
如今,在一项新的研究中,Pollock及其同事们开发出一种可现场应用的超灵敏诊断测试方法,可特异性地检测有症状和无症状疟疾患者体内所有疟原虫物种的DNA序列。这种新的疟疾诊断方法将优化的10分钟快速样品制备方案与基于CRISPR的SHERLOCK系统相结合,在简单的报告装置中,再过60分钟就能实现高度特异性和灵敏度的疟原虫检测。相关研究结果近期发表在PNAS期刊上,论文标题为“Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria”。

一锅化SHERLOCK测试方法的示意图。RT-RPA扩增疟原虫的靶序列,这种扩增过程与经过编程的Cas12a检测并行发生,从而导致靶序列切割以及荧光团标记的单链DNA报告分子的附带切割。荧光团标记的单链DNA报告分子可通过荧光策略进行检测,或者通过使用金纳米颗粒的测试条进行检测。图片来自PNAS, 2020, doi:10.1073/pnas.2010196117。
论文通讯作者、哈佛医学院怀斯生物启发工程研究所创始核心成员James Collins博士说,“这种可用于现场的SHERLOCK疟疾检测方法超越了世界卫生组织设定的理想测试方法的灵敏度和特异性要求,可用于检测所有主要疟原虫物种的无症状携带者中的低密度疟原虫。它的高度精简的设计可以为目前消除疟疾道路上的诊断瓶颈提供一种可行的解决方案,以便更广泛地实现低资源环境下的疟疾监测。”
这些研究人员证实他们的经过改造的SHERLOCK(Specific High-sensitivity Enzymatic Reporter unLOCKing)测试方法能够检测到每微升血液中少于2个疟原虫,这是世界卫生组织建议的在流行地区具有广泛实用性的测试方法的 “检测极限(limit of detection, LOD)”。通过分析含有恶性疟原虫和间日疟原虫物种的临床样本,他们能够以100%的灵敏度、100%的特异性识别出真正的阳性样本,并在真正的阴性样本中正确识别出缺少某种疟原虫物种的样本,从而显示出这种测试方法的临床潜力。接近100%的灵敏度和特异性是诊断测试方法在实际测试中使用的关键特性。此外,所设计的这种测试方法还可以确定经常发生突变的恶性疟原虫虫株的存在,这些虫株因发生突变而失去了HRP2抗原,因而逃避了常规快速诊断测试方法的检测。
Collins在怀斯生物启发工程研究所和麻省理工学院的研究团队与布罗德研究所的Feng Zhang团队共同开发了SHERLOCK技术。它被授权给了初创公司Sherlock Biosciences公司,该公司利用它为其他疾病的应用开发了一种快速分子诊断方法。近期,该公司开发的基于SHERLOCK的COVID-19诊断方法获得了FDA的紧急使用授权。
其他的方法已经被开发出来,比如这种新的SHERLOCK测试方法,可扩增和检测疟原虫的DNA(或RNA)核酸分子。然而,迄今为止,这些方法仍然受限于它们需要昂贵的实验设备(就像基于聚合酶链反应的方法那样)、复杂的样品制备技术和训练有素的人员,或者在在简单的情况下,“等温”扩增方法在同一个温度下执行,但是它们没有实现在现场测试中所需的灵敏度。
这种SHERLOCK疟疾测试方法利用了CRISPR-Cas12a酶,该酶经编程后可被结合到靶核酸序列上的向导RNA(gRNA)活化,就这项研究而言,靶核酸序列来自这四种疟原虫之一。活化的Cas12a然后非特异性地切割它附近的任何单链DNA链,而且是每秒发生大约1250次切割反应。这些研究人员在他们的测试方法中利用了这种扩增活性,将它与优化的样品制备相结合,不需要像其他一些核酸扩增测试方法那样进行特定的核酸提取步骤,并在前端对特定的疟原虫DNA和RNA序列进行等温扩增。识别扩增的疟原虫序列中物种特异性基因的gRNA随后激活Cas12a的活性,这样这种酶就会附带地攻击单链DNA报告序列,这种报告序列的切割产物有助于发出指示病原体特异性核酸存在的信号。在这种测试方法的后端,这种信号可被设计为手持设备中的荧光变化,或者在及时测试(POCT)设备中常用的侧向流测试条上的特定色带。
论文第一作者、Collins团队成员Rose Lee说,“重要的是,这种测试方法可兼容不同的样本类型,如全血、血浆、血清和干血,而且用于扩增、Cas12a活化和信号产生所需的所有成分都可以在单一试管中冻干,在它们与患者样本溶解和混合后,可发生一锅化反应(one-pot-reaction)。这避免了必须依赖功能性的冷链,并允许在低资源环境下以最少的专业知识进行测试。”(生物谷 Bioon.com)
参考资料:
1.Rose A. Lee el al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. PNAS, 2020, doi:10.1073/pnas.2010196117.
2.CRISPR-based malaria testing on-the-fly
https://medicalxpress.com/news/2020-09-crispr-based-malaria-on-the-fly.html
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


