研究开发出一种人工基因组的高效简化策略
来源:深圳先进院 2021-01-11 12:59
近日,中国科学院深圳先进技术研究院研究员戴俊彪团队开发出一种被称为SGC(SCRaMbLE-based genome compaction)的人工基因组的高效简化策略,并以此方法删除了第十二号染色体左臂中超过一半的非必需基因,为第一个最小真核基因组的构建、理解真核生命的核心组成奠定了理论和技术基础。相关研究成果以Compacting
近日,中国科学院深圳先进技术研究院研究员戴俊彪团队开发出一种被称为SGC(SCRaMbLE-based genome compaction)的人工基因组的高效简化策略,并以此方法删除了第十二号染色体左臂中超过一半的非必需基因,为第一个最小真核基因组的构建、理解真核生命的核心组成奠定了理论和技术基础。相关研究成果以Compacting a synthetic yeast chromosome arm为题,发表在Genome Biology上。戴俊彪、深圳先进院副研究员罗周卿、英国曼彻斯特大学教授蔡毅之为论文的共同通讯作者,罗周卿和深圳先进院研究助理余康为论文的共同第一作者。
最小真核基因组的构建是基因组学中的重要议题,被称为该领域的“圣杯”。通过精简基因组、去除冗余基因,可为认识生命的起源和进化提供重要线索,有助于深化对基因组功能组成和运转方式的认识。2016年,最小原核基因组由J. Craig Venter团队构建出来。面对更复杂的真核生物基因组,如何构建其最小基因组是合成基因组学领域具有挑战性的问题。
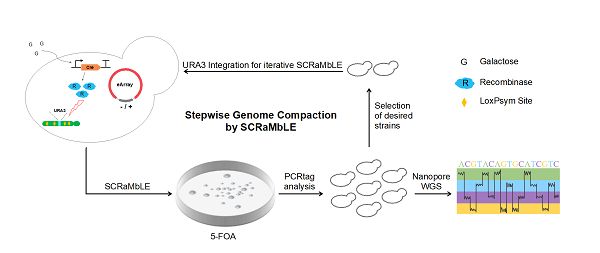
Sc2.0项目是世界上第一个真核基因组合成项目,已成功构建出六条染色体。与野生型基因组相比,合成酵母基因组的一个特征是在酵母基因组中系统性地插入了大量的序列特异性重组位点loxPsym,这使得在Cre重组酶表达时,合成基因组可以发生倒置、缺失、重复和易位等各种重排(SCRaMbLE系统),为最小酵母基因组的构建提供了可能,研究团队对此进行了深入研究,最终实现了真核合成染色体的高效精简。
SCRaMbLE产生的基因组重排是随机的,删除是其中的一种形式,为确保SCRaMbLE之后的基因组均发生序列删除,研究人员通过选取合适的插入位点,将选择性标记--URA3基因插入到合成的染色体中;通过药物对该基因的反筛作用,富集了该基因所在片段被删除的菌株。此外,SCRaMbLE介导的删除以两个loxPsym位点之间的序列为基本单位,因此,若两个loxPsym位点之间包含必需基因,则该片段内的其他非必需基因也无法被删除,如何覆盖到这些非必需基因,实现无偏删除?
对此,研究人员利用酿酒酵母同源重组技术,以eArray的形式为酵母细胞提供了必需基因的额外拷贝。在eArray存在的情况下,单次SCRaMbLE的删除能力提升了3倍左右,甚至可以一次删除合成序列上超过1/3的基因。为实现基因组的逐步简化,直至最小化,研究人员建立了合成基因组的连续删减流程,通过3次的连续删减,在保证菌株存活的前提下,最终删除了合成序列上65个非必需基因中的39个,将左臂的长度缩短了近100 kbp(原总长度为170 kbp)。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


