Nat Med : 黄荷凤院士/张静澜/徐晨明团队发表综合性无创产前筛查临床研究成果
来源:生物探索 2024-01-24 09:49
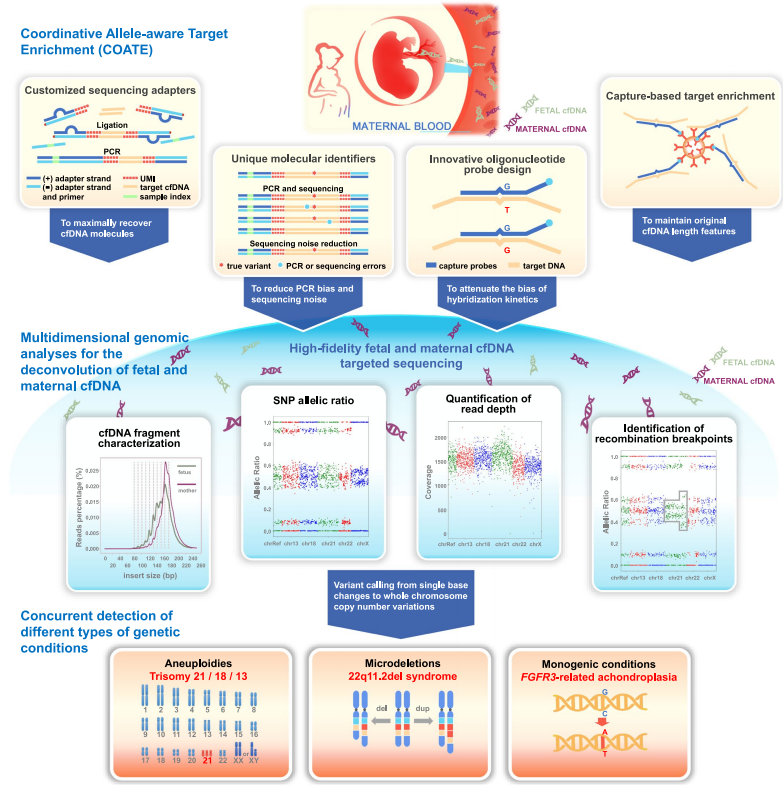
筛查技术同时覆盖了染色体非整倍体、染色体微缺失和单基因变异这三种最主要的人类遗传变异,扩展了无创产前筛查的检测范围,同时提高了检测准确性。
复旦大学生殖与发育研究院黄荷凤院士、张静澜研究员、徐晨明研究员联合浙江大学张丹教授、国家卫健委出生缺陷研究与预防重点实验室/湖南省儿童医院王华教授共同在Nature Medicine 发表题为Prospective prenatal cell-free DNA screening for genetic conditions of heterogenous etiologies(基于游离核酸的前瞻性多种类遗传病产前筛查)的研究成果,该研究是一项前瞻性、多中心的观察性研究,采用基于COATE-seq技术的综合性无创产前筛查技术,通过检测孕妇血浆中的游离DNA全面筛查胎儿不同类型的遗传变异(cfDNA综合筛查)。该筛查技术同时覆盖了染色体非整倍体、染色体微缺失和单基因变异这三种最主要的人类遗传变异,扩展了无创产前筛查的检测范围,同时提高了检测准确性。该研究展示在真实临床情境下对高风险孕妇进行全面筛查的临床研究结果,在产前筛查领域具有重要意义。

该研究将1090名参与者的cfDNA综合筛查结果与产前或产后诊断结果进行了比较,在135名孕妇中检测出了基因变异,灵敏度为98.5%,特异性为99.3%。在876个超声怀疑有结构异常的胎儿中,cfDNA综合筛查发现了55个(56.1%)非整倍体、6个(6.1%)微缺失和37个(37.8%)单基因致病变异。研究结果证实,将目标单基因病与染色体异常同时纳入筛查,使检出率提高了60.7%(从61例提高到98例)。研究还表明,cfDNA综合筛查在孕早期发现无症状胎儿(如软骨发育不全胎儿)和影像学方法不易发现的神经缺陷方面具有重要意义。例如,37个单基因病胎儿中,有13个(35.1%)检出与产后神经系统损伤(如学习障碍、发育迟缓和智力障碍)相关的致病变异(根据美国医学遗传学和基因组学学会变异解读指南定级),但未在产前表现出大脑或中枢神经系统异常。
全面的产前无细胞DNA筛选试验(Source: Nature Medicine )
为了将cfDNA综合筛查扩展到普通孕妇群体中,需要更具体的疾病纳入标准,该研究提出了一个临床优先级体系 “SEPH”,重点关注表型严重(severe outcome)、早期发病(early onset)、发病率高(prevalent incidence)和检测方法学性能高(high performance)的疾病。该论文研究结果显示cfDNA综合筛查可以准确识别高危妊娠中染色体和单基因水平的胎儿致病性变异,有助于实现更加精准的产前和围产期管理,将对出生缺陷防治工作产生深远的影响。

胎儿结构异常妊娠中诊断性遗传变异的检出率(Source: Nature Medicine )
由于隐性遗传病多数为父母来源,孕前携带者筛查可提前明确生育风险,通过生育指导来防控出生缺陷;但显性单基因疾病以新发突变为主,无法通过检测父母排除风险,胎儿发育过程中没有严重结构异常的单基因病可能会在超声筛查中被漏检,是当前出生缺陷防控领域的薄弱环节。该研究将高发、严重、新发突变为主的显性单基因病纳入检测范围,具有显著的临床价值,是未来无创产前筛查技术的发展方向。随着研究的深入和技术的完善,未来有望为孕妇提供更安全、更准确的产前筛查服务,一次采集孕妇外周血就可完成对所有常见变异的同步检测,真正实现孕妇的全面无创产前筛查。
复旦大学附属妇产科医院张静澜/吴琰婷/陈松长,浙江大学医学院附属妇产科医院罗琼,湖南省妇幼保健院席惠为本文的共同第一作者。
专家点评:
专家1(乔杰院士, 北京大学常务副校长,医学部主任):
在这项研究中,黄荷凤院士团队运用了他们自主研发的协同等位基因靶向富集测序(COATE-seq)技术,用于检测染色体非整倍体和染色体微缺失。COATE-seq技术的核心创新在于其对不同等位基因的无偏好靶向富集探针设计,突破传统探针设计的限制,展现了新颖巧妙的思路,为cfDNA检测的临床应用展示了新的道路。COATE-seq技术与国内常用的低深度全基因组测序方法不同,通过有效降低等位基因杂交的偏好可更精准地检测胎儿cfDNA单核苷酸多态性(SNP)的等位基因频率。这种高精度的检测方法使得COATE-seq能够更有效地区分母胎来源的cfDNA。此外,该研究在生物信息学分析方面也进行了重要发展,通过同时分析等位基因频率和测序深度,提升了染色体异常的检测精度。COATE-seq技术在检测单基因病的点突变方面同样展现了显著优势。因为胎儿游离DNA在母体血液中的比例相对较低,在母体背景下检测胎儿的单基因变异通常较为困难,COATE-seq通过高深度测序获取等位基因频率,并利用cfDNA片段的大小分布的信息,为单基因变异的检测提升了特异性。黄荷凤院士团队的该项临床研究最大的创新之处在于将单基因病纳入无创产前筛查范围,这一进步不仅显著提高了胎儿遗传病的检出率,而且在推广至临床应用后,将为孕妇及医生在妊娠的决策和管理过程中提供更加安全、及时、准确的信息,标志着在遗传病和出生缺陷防治领域的一个重要突破。
该研究的主要目的是评估新型无创产前检测技术COATE-seq的检测效能,因此参与者限定为高风险孕妇。要将COATE-seq技术推广到更广泛的人群中,如文中讨论部分所言,需要在普通孕妇中进行进一步的临床研究。此外,黄荷凤院士团队特别提出了将单基因病纳入针对普通妊娠的无创产前筛查的规则,包括更细致的疾病选择标准,确定纳入疾病的发病率阈值、表型严重程度量化指标等。这些标准将有助于优化筛查流程,确保筛查的临床实用性。这些考量体现了黄院士团队在进行临床研究时的严谨和科学态度,他们不仅关注技术开发本身,还重视实际临床应用中的有效性和适用性,在推动科学技术进步的同时,确保新技术能更安全、有效地服务于广大孕妇,在临床上提高妊娠管理的质量和效果。
专家2 (杨正林院士, 四川省人民医院院长,中国生物物理学会临床分子诊断会长):
这项观察性临床研究的核心研究目的是通过比较cfDNA检测和遗传学诊断结果,评估产前cfDNA综合筛查在多种染色体异常和单基因病中的临床有效性。该筛查涵盖了7种常见非整倍体和9种微缺失综合征,以及与显性单基因病相关的75个基因。临床研究设计严谨,入组病人进行侵入性产前诊断基于临床指征,且所有临床妊娠管理决策均依据诊断结果而非仅凭cfDNA筛查结果。研究结果表明,与传统NIPT相比,该cfDNA综合筛查在胎儿结构畸形遗传变异的检出率提升了60.7%,突显了单基因病在出生缺陷中的高占比。该研究还观察到染色体病和单基因病的并行产前筛查能够揭示与表型相关的重要数据。例如,32.6%的淋巴系统异常胎儿检出染色体异常,而仅4.3%由单基因变异引起。对于骨骼畸形胎儿,23.5%检出单基因变异,而仅1.2%归因于染色体异常。特别值得关注的是,研究中有13个胎儿未表现出大脑或中枢神经系统异常,但检出与产后神经系统损伤相关的致病变异。产前超声波检查的局限性导致一些单基因病(如Rett综合征)在产前不表现任何症状,或一些病症(如软骨发育不全)直到孕中期或晚期才能通过超声发现。cfDNA综合筛查能在孕早期发现尚未表现症状的患病胎儿,这对临床决策极为重要,但同时也带来遗传咨询和伦理上的考量,需要建立完善的遗传咨询体系来详细解读报告并辅助受检者作出决策。
新发突变所致的显性单基因病是当前产前筛查的薄弱环节,将其纳入胎儿cfDNA综合筛查是NIPT发展的必然趋势。黄荷凤院士团队的研究不仅提出了新颖且高效的技术路径,还展示了相关技术在临床上的有效性,为进一步提升产前诊断、遗传性出生缺陷的防治提供了重要理论和实践依据。
专家3 (漆洪波, 重庆医科大学附属妇女儿童医院、重庆市妇幼保健院院长,中华医学会围产医学分会候任主任委员):
这项多中心前瞻性队列研究通过对比新一代cfDNA筛查与产前或产后遗传学诊断的结果,显示了胎儿cfDNA筛查在包括非整倍体、微缺失和单基因变异在内的多种遗传变异进行同步检测中的临床有效性。这种三合一的全面的无创产前筛查显著提高了造成胎儿结构畸形的遗传变异的检出率,体现了该项新技术在临床应用方面的巨大潜力,能有效填补目前产前筛查在显性单基因病方面的缺失,对完善我国的产前筛查体系和出生缺陷防控工作具有重要的实际意义。对于孕妇而言,这种一站式筛查方式使得一次采血就能完成对染色体病和单基因病的全面筛查,避免了分步骤进行染色体和单基因病检测所累积的时间,为临床决策提供了更多的宝贵时间。对于NIPT筛查的假阳性结果,本研究显示非整倍体筛查仍面临胎盘限制性嵌合和胎儿与胎盘基因组不同等问题,但在显性单基因病筛查方面,其准确性接近100%。这种差异的原因应在更大规模的队列研究中进行深入探索。本论文设计的胎儿cfDNA筛查主要针对遗传病风险较高的孕妇,筛查的75个单基因病基因也是为这些高危人群特别选择的,如果要将其应用于更广泛的人群,需要根据检测内容或不同的地区进行相应的调整。此外,这项研究属于观察性研究,未来可以考虑开展干预性研究,以评估该筛查技术在临床管理上的积极效用。
参考文献:
1. Chiu, R.W., et al. Noninvasive prenatal diagnosis of fetal chromosomal aneuploidy by massively parallel genomic sequencing of DNA in maternal plasma. Proc Natl Acad Sci U S A 105, 20458-20463 (2008).
2. Dungan, J.S., et al. Noninvasive prenatal screening (NIPS) for fetal chromosome abnormalities in a general-risk population: An evidence-based clinical guideline of the American College of Medical Genetics and Genomics (ACMG). Genet Med 25, 100336 (2023).
3. Zhang, J., et al. Non-invasive prenatal sequencing for multiple Mendelian monogenic disorders using circulating cell-free fetal DNA. Nat Med 25, 439-447 (2019).
4. Hanson, B., Scotchman, E., Chitty, L.S. & Chandler, N.J. Non-invasive prenatal diagnosis (NIPD): how analysis of cell-free DNA in maternal plasma has changed prenatal diagnosis for monogenic disorders. Clin Sci (Lond) 136, 1615-1629 (2022).
5. Xu, C., et al. Genetic deconvolution of fetal and maternal cell-free DNA in maternal plasma enables next-generation non-invasive prenatal screening. Cell Discov 8, 109 (2022).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


