2019年12月Science期刊不得不看的亮点研究
来源:本站原创 2019-12-31 23:36
2019年12月31日讯/生物谷BIOON/---2019年12月份即将结束了,12月份Science期刊又有哪些亮点研究值得学习呢?小编对此进行了整理,与各位分享。1.Science:挑战常规!一部分γδT细胞结合到MHC样分子的下面来识别有害细胞doi:10.1126/science.aav3900在一项新的研究中,来自澳大利亚莫纳什大学、澳大利亚研究理
2019年12月31日讯/生物谷BIOON/---2019年12月份即将结束了,12月份Science期刊又有哪些亮点研究值得学习呢?小编对此进行了整理,与各位分享。
1.Science:挑战常规!一部分γδT细胞结合到MHC样分子的下面来识别有害细胞
doi:10.1126/science.aav3900
在一项新的研究中,来自澳大利亚莫纳什大学、澳大利亚研究理事会先进分子成像卓越中心和墨尔本大学多赫蒂研究所等研究机构的研究人员重新定义了过去20年来我们对T细胞识别的认识。 相关研究结果近期发表在Science期刊上,论文标题为“A class of γδ T cell receptors recognize the underside of the antigen-presenting molecule MR1”。

为了与体内其他细胞相互作用,T细胞依赖于称为T细胞受体的专门受体,该受体识别与称为主要组织相容性复合体(MHC)或MHC样分子的特殊分子结合的病毒或细菌片段。在过去的20年中, 普遍的看法是T细胞受体结合在MHC和MHC样分子的上面以进行识别。
在这项新的研究中,这些研究人员描述了一类鲜为人知的称为γδT细胞的T细胞中的一个细胞群体,该群体能够识别一种称为MR1的MHC样分子。通过在澳大利亚同步加速器上使用高强度X射线 束,他们获得了γδT细胞受体与MR1之间相互作用的详细三维图像,并获得令人感兴趣的结果:γδT细胞受体结合在MHC样分子的下面以进行识别。这种高度不同寻常的识别机制重塑了我们 对T细胞受体如何与它们的靶分子相互作用的理解,并代表着T细胞生物学领域取得的重大发展。
论文共同第一作者、莫纳什大学生物医学发现研究所的Jér?me Le Nours博士说,“我们把它想象成附着在细胞上的标志。我们一直以为T细胞会过来,从上面读取这种标志。我们已确定有些T 细胞可以从下方接近并与它互动。这些都是可以改变我们未来研究T细胞生物学方法的重要细节。这很重要,这是因为T细胞是我们免疫系统的关键武器,而且了解T细胞如何靶向并作用于其他 细胞对于利用它们的力量进行开发治疗感染和癌症等疾病的免疫疗法至关重要。”
2.Science:对人类血细胞中的所有蛋白编码基因进行全基因组范围内的转录组分析
doi:10.1126/science.aax9198
在临床和研究环境中,血液都是对人类进行分子分析的主要来源,并且是许多治疗策略的目标,这突显了需要对构成人类血液的细胞进行全面的分子图谱构建。人类蛋白质图谱计划(www.proteinatlas.org)是一个开放式数据库,旨在通过整合各种组学技术(包括基于抗体的成像)来绘制所有人类蛋白的图谱。在此之前,人类蛋白质图谱包括来自外周血单核细胞的基因表达信息,但不包括来自许多其他血细胞亚群的基因表达信息。为了提高分辨率,人们需要对血液中的细胞进行了深入表征,以提供人血细胞中基因表达的详细细节,并将它们与体内的其他组织相关联在一起。
为此,在一项新的研究中,来自瑞典卡罗林斯卡研究所等研究机构的研究人员对通过流式细胞仪分选技术分离出的18种经典免疫细胞群体进行了基于转录组学的表达分析。他们将血细胞表达谱与组织表达谱相结合在一起,包括来自外部来源的转录组学数据以扩大这个开放式数据库中包含的组织类型和大脑区域的数量。他们依据血细胞和组织中的表达特异性和分布对蛋白编码基因进行了全基因组分类。相关研究结果近期发表在Science期刊上,论文标题为“A genome-wide transcriptomic analysis of protein-coding genes in human blood cells”。
这些研究人员提供了人体血细胞中所有蛋白编码基因的表达图谱,并根据人体所有主要组织和器官中所有蛋白编码基因的特异性和分布进行分类。对血细胞RNA表达谱进行全基因组分析可以鉴定出在各种免疫细胞中的表达水平上调的基因,从而确认了之前已知的蛋白标志物,而且还鉴定出新靶标以进行深入分析。有1448个蛋白编码基因在单个免疫细胞类型中大量地表达。进一步研究相应的蛋白以探索与各个细胞表型相关的生物学功能将是有趣的。
3.Science:宿主细胞利用芳烃受体侦查细菌群体感应信号
doi:10.1126/science.aaw1629
菌感染不会自动导致疾病;许多细菌只有在大量出现时才变得危险。在一项新的研究中,来自德国马克斯普朗克感染生物学研究所等研究机构的研究人员发现宿主细胞具有一种受体,它不能 识别细菌本身,但可以侦察细菌之间的通讯。当有大量细菌存在时,宿主就会使用这种受体来记录它们分泌的称为毒力因子的致病性物质。相关研究结果近期发表在Science期刊上,论文标题 为“Host monitoring of quorum sensing during Pseudomonas aeruginosa infection”。

论文通讯作者、马克斯普朗克感染生物学研究所的Stefan Kaufmann及其研究团队发现宿主细胞能够借助于一种称为芳烃受体(aryl hydrocarbon receptor)的受体观察到铜绿假单胞菌之间 的交流。这种受体检测群体感应分子,使得宿主细胞能够检测到这种细菌何时准备发起攻击。论文第一作者Pedro Moura-Alves解释道,“通过这种刺探行为,宿主细胞能够在需要抵御这种细 菌攻击的时候激活免疫系统。”
实际上,在铜绿假单胞菌达到它们的群体感应水平之前,这种受体窃听这种细菌之间的交谈;对群体感应的早期阶段进行检测,抑制芳烃受体,从而阻断宿主免疫防御的过早启动。Kaufmann 说,“这对宿主是有效的,这是因为它节省了能量,让少量的细菌单独存在,前提是它们不会造成任何损害。只有当它们达到临界数量时,才有足够的能量进行防御。”这也有助于阻止由免 疫系统反应引起的附带损害。
4.Science:挑战常规!揭示microRNA抑制mRNA表达新机制
doi:10.1126/science.aav1741
如今,在一项新的研究中,在美国怀特黑德生物医学研究所生物学教授David Bartel、研究生Sean McGeary和前研究生Kathy Lin的领导下,研究人员收集了关于6种miRNA的大量数据,并在此 基础上开发出一种针对所有单个miRNA的改进型预测模型。他们的发现为miRNA靶标预测提供了前所未有的准确性和粒度。相关研究结果近期发表在Science期刊上,论文标题为“The biochemical basis of microRNA targeting efficacy”。
为了了解miRNA的靶向作用,人们需要在miRNA序列中鉴定出可与miRNA结合的特定位点,并且他们还需要了解每个位点上的相互作用强度—结合亲和力(binding affinity)。通常,当miRNA 的前八个核苷酸中的至少六个与mRNA某位置上的互补核苷酸序列匹配时,miRNA将与mRNA结合。这两个序列就像成排的被推到一起的拼图块:如果每个拼图块都插入到相应的拼图块中,那么这 些成排的拼图块就组合成一个锁定的拼图---miRNA就可结合它的靶标。如果这些拼图块不能拼在一起,这些成排的拼图块就不能连接在一起。这些结合位点与miRNA的前8个核苷酸完全匹配, 被称为典型位点(canonical site),人们过去认为它们之间存在明显的层次关系,无论miRNA的身份如何,每种位点都会产生相似的抑制量。然而,McGeary并未观察到这种情形。
McGeary研究了六种miRNA,并开发了一种方法来测量每种miRNA与大量RNA序列的相对结合亲和力。这些测量以及McGeary从中进行的进一步计算形成了一个新的丰富的数据库,可用来改善 miRNA靶向预测。通过这些实验,这些研究人员发现经典位点的预期靶向层次结构并不适用于所有miRNA。实际上,单个miRNA对这种预期层次结构中较低的一个经典位点有更强的亲和力。此外 ,他们发现每个miRNA都有独特的非经典结合位点,其中的一些位点含有至少一个碱基错配但仍能够结合miRNA。他们发现,在许多情况下,尽管非经典位点的配对不完美或异常,但是miRNA与 一个非经典位点的结合要比与某些经典位点的结合更牢固。 Lin说:“作为人类,我们喜欢按照不同的特征将事物进行分类。但是要建立一种定量模型,你必须认识到每种miRNA与靶标之间的 相互作用是不同的。”
5.长达十年的科学争端终解决!两篇Science论文揭示人黏连蛋白通过挤压DNA环进行基因组组装
doi:10.1126/science.aaz3418; doi:10.1126/science.aaz4475
黏连蛋白(cohesin)是一种结合染色体的多亚基腺苷三磷酸酶复合物。在加载到染色体上后,它会产生DNA环来调节染色体功能。有人提出黏连蛋白通过环挤压来实现基因组组装,然而缺乏直接的证据来支持这一点。
在第一项新的研究中,奥地利维也纳生物中心分子病理学研究所(IMP)主任Jan-Michael Peters及其团队首次证实一种分子机器通过“环挤压(loop extrusion)”主动地和有目的地折叠DNA,从而在间期细胞中实现了多种重要功能。这种针对DNA成环(DNA looping,即形成DNA环)过程提出的新见解改变了关于基因组如何在细胞内组装的旧观点。这一发现阐明了生命的基本机制,并解决了长达十年的科学争端。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“DNA loop extrusion by human cohesin”。
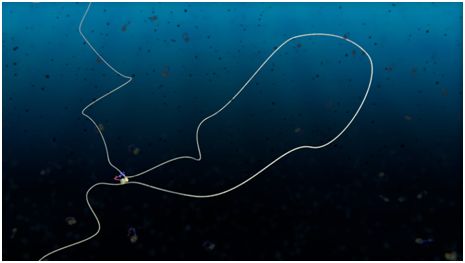
Peters团队(包括Peters实验室资深博士后研究员Iain Davidson)能够在体外的一种简化系统中重建黏连蛋白的功能。因此,Davidson能够观察到单个黏连蛋白分子如何将DNA的单个片段快速地挤压成DNA环。他的发现影响深远,并以多种方式改变了对基因组的整体认识:(1)基因组不是静态的,而是高度动态的结构;(2)基因组DNA的折叠是一种受到主动调节的过程,它涉及通过挤压让DNA分子成环,并且许多DNA环在不断运动;(3)这种DNA成环是由黏连蛋白介导的,因此黏连蛋白必须是一种分子马达,类似于诸如肌球蛋白之类的其他马达蛋白;(4)黏连蛋白分子在DNA周围形成钩环状的环状结构,而且还必须通过多个结合位点动态连接到DNA上,这样才能够折叠DNA;正如去年所发现的那样,凝缩蛋白(condensin)也必须如此。
在第二项新的研究中,来自美国德克萨斯大学西南医学中心和德克萨斯州大学奥斯汀分校的研究人员利用单分子成像揭示重组的人黏连蛋白-NIPBL复合物通过挤压DNA环来压缩裸露的和核小体结合的DNA。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“Human cohesin compacts DNA by loop extrusion”。论文通讯作者为德克萨斯大学西南医学中心的Hongtao Yu博士和德克萨斯州大学奥斯汀分校的Ilya J. Finkelstein博士。
黏连蛋白开展的这种DNA压缩需要三磷酸腺苷(ATP)水解,并且对压力敏感。这种压缩过程以每秒0.5kb的平均速率处理数万个碱基。黏连蛋白对双链DNA的压缩表明黏连蛋白二聚体双向挤压DNA环。这些结果表明黏连蛋白-NIPBL复合物是一种由ATP驱动的能够进行DNA环挤压的分子马达。
总之,这两项新的研究表明与黏连蛋白调节姐妹染色单体黏连在一起的方式不同,这种蛋白在环挤压过程中似乎不会在拓扑结构上捕获DNA。这些结果为染色质组装的环挤压模型提供了直接证据,并表明基因组结构是高度动态变化的。
6.Science:新方法鉴定肠道微生物组产生的代谢物分子
doi:10.1126/science.aav1282; doi:10.1126/science.aaz4164
肠道菌群产生数百种以高水平存在于血液循环中的分子,它们的水平在不同人之间差异很大。这些分子是研究肠道微生物组与宿主相互作用的一个有前景的起点;少数经过详细描述的分子具有强效的免疫或代谢调节活性,并且是G蛋白偶联受体或核激素受体的配体。但是,在大多数情况下,这些分子的产生尚未与特定的细菌菌株或代谢途径相关联在一起,并且难以弄清每种分子对宿主生物学的贡献。
在一项新的研究中,来自美国斯坦福大学等研究机构的研究人员描述了一种基于CRISPR-Cas9的方法,用于在模式共生梭菌---生孢梭菌(Clostridium sporogenes)---中可靠地构建无需进行标记的不产生特定代谢物分子的细菌突变体。这种方法能够构建出多种发生突变的生孢梭菌菌株。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“Depletion of microbiome-derived molecules in the host using Clostridium genetics”。
通过使用这种方法敲除10种由产孢梭菌产生的分子---三甲胺、5-氨基戊酸酯、色胺、吲哚丙酸、异戊酸、2-甲基丁酸、异丁酸、异己酸、丙酸和丁酸,他们展示了它的实用性;我们通过液相色谱-质谱或气相色谱-质谱法验证体外培养的生孢梭菌的提取物中相应代谢物的缺乏来验证了每种敲除的成功性。
接着,他们将野生型生孢梭菌或者这5种缺乏相应代谢物的生孢梭菌突变体之一定植到无菌小鼠中,结果发现在定植野生型生孢梭菌的小鼠中,这些代谢途径的产物在宿主组织中积累,但是每种代谢物可通过定植相应的代谢途径突变体来加以剔除。通过比较接种野生型生孢梭菌定植的小鼠和接种缺乏产生分支短链脂肪酸---异丁酸、2-甲基丁酸或异戊酸---的生孢梭菌突变体的小鼠,他们发现这些丰富的肠道微生物组衍生性分子具有一种以前未知的调节免疫球蛋白A(IgA)浆细胞的活性。
7.Science:成功诱导免疫系统产生关键抗体来抵御HIV的感染
doi:10.1126/science.aay7199; doi:10.1126/science.aaz8647
近日,一项刊登在国际杂志Science上的研究报告中,来自杜克大学医学中心的科学家们通过研究清除了HIV疫苗开发过程中遇到的主要障碍,同时在动物模型中也证实有效,即诱导短时效抗体增殖来成为有效抵御HIV的中坚力量。
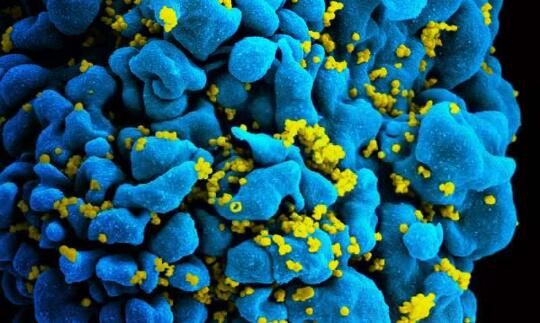
医学博士Barton F. Haynes表示,如今我们并没有HIV疫苗的原因在于机体免疫系统无法制造出能够中和HIV的特殊抗体类型,这项研究中,我们通过对免疫系统进行工程化操作来使其产生一种特殊环境从而制造出合适的抗体。基于此前多年的研究结果,如今研究人员阐明了广谱中和性抗体(bnAbs)如何及何时在HIV感染者机体中出现,以及何时能够有效阻断抗体增殖来抑制病毒。
其中一个问题在于免疫系统,其会将bnAbs视为一种危险并关闭bnAbs的产生过程;另外一个问题就是中和性抗体需要在其遗传组成上发生罕见改变,这在重要的B细胞多样化过程中并不会产生。这项研究中,研究人员追踪了相关突变,随后他们对一种HIV蛋白进行工程化修饰,靶向作用病毒包膜上V3聚糖区域位点,从而使其能够优先与携带必要突变的抗体相结合。利用表达人类中和性抗体前体的小鼠模型进行研究,研究人员发现,免疫原能够诱导B细胞谱系来经历不可能的突变从产生广谱性抗体。
研究者Alt表示,我们能够制造出表达人类广谱性中和抗体的小鼠模型,这就能为我们提供强大的新型模型系统,在该系统中,我们就能够反复检测实验性的HIV疫苗;第二种谱系的bnAbs则会经历一种不可能的突变,其会结合病毒外膜上名为CD4结合位点的不同区域,当研究人员重建了抗体历史后,他们开发出了第二种免疫原,在非人类灵长类动物中进行测试后,研究者发现,这种免疫原同样会选择必要的突变,这就能够帮助开发潜在的CD4结合位点中和性抗体。
8.Science:科学家成功利用九种酶类的链式反应来制造HIV药物islatravir
doi:10.1126/science.aay8484; doi:10.1126/science.aaz7376
近日,一项刊登在国际杂志Science上的研究报告中,来自默克和克迪科思公司的研究人员通过研究成功利用一种九种酶的链式反应方法来制造HIV药物—islatravir;文章中,研究人员描述了他们工作的灵感以及最终产品的效果。
研究者指出,多种多样的天然产物是很多基本元件进行生物合成的结果,很多有机体会利用酶类的选择性来完成每一步过程,而这通常会涉及一种多步的连锁反应,其中第一个反应就会被用作下一步反应的基础。基于此,研究人员就开始寻找能够制造HIV实验性药物islatravir的方法,首先他们将目光锁定到细菌的核苷补救途径(NSP,nucleoside salvage pathway)上,该过程主要参与从RNA降解到DNA的碱基和核苷的回收,在这一过程中有三种主要的酶类,即脱氧核糖-5-磷酸醛缩酶(DERA,deoxyribose-5-phosphate aldolase)、戊磷酸变位酶(PPM,phosphopentomutase)和嘌呤核苷磷酸化酶(PNP,purine nucleoside phosphorylase)。
为了利用相同的过程来开发药物islatravir,研究者就需要利用一种酶类的特殊特性,即可逆特性,酶类的这种特性能够使其有别于传统的催化剂。研究人员必须找到一种方法来让上述三种酶类接受非天然的底物,解决的方法就是从大肠杆菌的RNP和PPM中借用反向的NSP,从S. halifaxensi菌中借用DERA,为了使其能够发挥作用,研究人员还必须对酶类进行工程化修饰来使其变得更具活性,这项工作还涉及到优化另外两种酶,并在自然状态下使用其它四种酶类。最后的结果就是涉及九种酶类的级联反应,最终就能够制造出药物islatravir。
9.Science:重磅!科学家在眼睛中鉴别出了能帮助大脑区分昼夜的特殊细胞!
doi:10.1126/science.aaz0898
夜间的强光会打乱机体正常的昼夜节律(circadian rhythms),从而引发失眠;实际上,昼夜几率在机体健康上扮演着重要的角色,干扰昼夜节律常常会增加多种疾病的发病风险,比如癌症、心脏病、肥胖、抑郁症等,因此,理解人眼感知光线的机制或能帮助有效预防抑郁、促进夜间睡眠并保持机体健康的昼夜节律。
近日,一项刊登在国际杂志Science上的研究报告中,来自索尔克研究所的科学家们通过研究在眼睛中发现了三种细胞类型,其能帮助检测光线,并使大脑的昼夜节律与周围的光线保持一致,这项研究中,研究者首次评估了这种名为本质感光性视网膜神经节细胞(ipRGCs,intrinsically photosensitive retinal ganglion cells)的细胞对光线的反应,以及其对机体健康的重要应用。
文章中,研究者揭示了三种类型的ipRGCs细胞,第一种细胞对光的响应速度较快,但关闭时间较长,而第二种细胞开启和关闭时间均较长,第三类细胞在灯光非常明亮时才会产生响应,但其开启非常迅速且能在灯光消失后立马关闭。理解每一种ipRGCs细胞的功能或能帮助研究人员设计出更好的光照条件或疗法来开启/关闭这些细胞活性。
研究者指出,ipRGCs细胞负责将光信号传输到大脑中,甚至是在缺乏视杆细胞和视锥细胞的人群中也需要向大脑中传递图像。在携带有正常视杆细胞和视锥细胞的人群中,ipRGCs细胞实际上能与这些细胞密切合作,本文研究表明,ipRGCs细胞能结合视杆和视锥细胞所检测到的光信号,从而为我们所看到的信息增加亮度和对比度信息。
10.Science:重大进展!新研究发现最常见的儿童肾癌的遗传根源
doi:10.1126/science.aax1323
儿童肾癌很少见。维尔姆斯肿瘤(Wilms' tumour)是儿童肾癌的最常见类型,主要影响五岁以下的儿童。它在英国每年诊断出大约80例。十分之九的病例可以通过外科手术去除受影响的肾脏,同时进行化疗,有时还进行放疗,就可被治愈。通常而言,这种癌症仅影响一个肾脏,但是在大约10%的病例中,两个肾脏都受到影响。据推测,维尔姆斯肿瘤的病因是在生命最初几周内的肾脏发育过程中,一个细胞出现了“差错”。
在一项新的研究中,英国研究人员发现了儿童肾癌维尔姆斯肿瘤的最早遗传根源。通过比较来自正常肾脏组织和肿瘤的基因组序列之后,他们鉴定出看上去正常的肾脏组织块实际上携带导致维尔姆斯肿瘤的DNA变化。相关研究结果发表在2019年12月5日的Science期刊上,论文标题为“Embryonal precursors of Wilms tumor”。

这项研究揭示了一种癌症可以在生命的最初几周内形成的新途径,在这种途径中,早期的细胞获得与癌症相关的修饰,并且在肾脏发育时增殖。了解这种癌症的根源有望改善治疗,并有助阻止维尔姆斯肿瘤复发。这一发现还增加了有朝一日有可能在肿瘤产生之前筛查这种癌症的可能性。(生物谷 Bioon.com)
1.Science:挑战常规!一部分γδT细胞结合到MHC样分子的下面来识别有害细胞
doi:10.1126/science.aav3900
在一项新的研究中,来自澳大利亚莫纳什大学、澳大利亚研究理事会先进分子成像卓越中心和墨尔本大学多赫蒂研究所等研究机构的研究人员重新定义了过去20年来我们对T细胞识别的认识。 相关研究结果近期发表在Science期刊上,论文标题为“A class of γδ T cell receptors recognize the underside of the antigen-presenting molecule MR1”。

来自健康供者免疫系统的人T细胞的扫描电镜图,图片来自NIAID。
为了与体内其他细胞相互作用,T细胞依赖于称为T细胞受体的专门受体,该受体识别与称为主要组织相容性复合体(MHC)或MHC样分子的特殊分子结合的病毒或细菌片段。在过去的20年中, 普遍的看法是T细胞受体结合在MHC和MHC样分子的上面以进行识别。
在这项新的研究中,这些研究人员描述了一类鲜为人知的称为γδT细胞的T细胞中的一个细胞群体,该群体能够识别一种称为MR1的MHC样分子。通过在澳大利亚同步加速器上使用高强度X射线 束,他们获得了γδT细胞受体与MR1之间相互作用的详细三维图像,并获得令人感兴趣的结果:γδT细胞受体结合在MHC样分子的下面以进行识别。这种高度不同寻常的识别机制重塑了我们 对T细胞受体如何与它们的靶分子相互作用的理解,并代表着T细胞生物学领域取得的重大发展。
论文共同第一作者、莫纳什大学生物医学发现研究所的Jér?me Le Nours博士说,“我们把它想象成附着在细胞上的标志。我们一直以为T细胞会过来,从上面读取这种标志。我们已确定有些T 细胞可以从下方接近并与它互动。这些都是可以改变我们未来研究T细胞生物学方法的重要细节。这很重要,这是因为T细胞是我们免疫系统的关键武器,而且了解T细胞如何靶向并作用于其他 细胞对于利用它们的力量进行开发治疗感染和癌症等疾病的免疫疗法至关重要。”
2.Science:对人类血细胞中的所有蛋白编码基因进行全基因组范围内的转录组分析
doi:10.1126/science.aax9198
在临床和研究环境中,血液都是对人类进行分子分析的主要来源,并且是许多治疗策略的目标,这突显了需要对构成人类血液的细胞进行全面的分子图谱构建。人类蛋白质图谱计划(www.proteinatlas.org)是一个开放式数据库,旨在通过整合各种组学技术(包括基于抗体的成像)来绘制所有人类蛋白的图谱。在此之前,人类蛋白质图谱包括来自外周血单核细胞的基因表达信息,但不包括来自许多其他血细胞亚群的基因表达信息。为了提高分辨率,人们需要对血液中的细胞进行了深入表征,以提供人血细胞中基因表达的详细细节,并将它们与体内的其他组织相关联在一起。
为此,在一项新的研究中,来自瑞典卡罗林斯卡研究所等研究机构的研究人员对通过流式细胞仪分选技术分离出的18种经典免疫细胞群体进行了基于转录组学的表达分析。他们将血细胞表达谱与组织表达谱相结合在一起,包括来自外部来源的转录组学数据以扩大这个开放式数据库中包含的组织类型和大脑区域的数量。他们依据血细胞和组织中的表达特异性和分布对蛋白编码基因进行了全基因组分类。相关研究结果近期发表在Science期刊上,论文标题为“A genome-wide transcriptomic analysis of protein-coding genes in human blood cells”。
这些研究人员提供了人体血细胞中所有蛋白编码基因的表达图谱,并根据人体所有主要组织和器官中所有蛋白编码基因的特异性和分布进行分类。对血细胞RNA表达谱进行全基因组分析可以鉴定出在各种免疫细胞中的表达水平上调的基因,从而确认了之前已知的蛋白标志物,而且还鉴定出新靶标以进行深入分析。有1448个蛋白编码基因在单个免疫细胞类型中大量地表达。进一步研究相应的蛋白以探索与各个细胞表型相关的生物学功能将是有趣的。
3.Science:宿主细胞利用芳烃受体侦查细菌群体感应信号
doi:10.1126/science.aaw1629
菌感染不会自动导致疾病;许多细菌只有在大量出现时才变得危险。在一项新的研究中,来自德国马克斯普朗克感染生物学研究所等研究机构的研究人员发现宿主细胞具有一种受体,它不能 识别细菌本身,但可以侦察细菌之间的通讯。当有大量细菌存在时,宿主就会使用这种受体来记录它们分泌的称为毒力因子的致病性物质。相关研究结果近期发表在Science期刊上,论文标题 为“Host monitoring of quorum sensing during Pseudomonas aeruginosa infection”。

图片来自Science, 2019, doi:10.1126/science.aaw1629。
论文通讯作者、马克斯普朗克感染生物学研究所的Stefan Kaufmann及其研究团队发现宿主细胞能够借助于一种称为芳烃受体(aryl hydrocarbon receptor)的受体观察到铜绿假单胞菌之间 的交流。这种受体检测群体感应分子,使得宿主细胞能够检测到这种细菌何时准备发起攻击。论文第一作者Pedro Moura-Alves解释道,“通过这种刺探行为,宿主细胞能够在需要抵御这种细 菌攻击的时候激活免疫系统。”
实际上,在铜绿假单胞菌达到它们的群体感应水平之前,这种受体窃听这种细菌之间的交谈;对群体感应的早期阶段进行检测,抑制芳烃受体,从而阻断宿主免疫防御的过早启动。Kaufmann 说,“这对宿主是有效的,这是因为它节省了能量,让少量的细菌单独存在,前提是它们不会造成任何损害。只有当它们达到临界数量时,才有足够的能量进行防御。”这也有助于阻止由免 疫系统反应引起的附带损害。
4.Science:挑战常规!揭示microRNA抑制mRNA表达新机制
doi:10.1126/science.aav1741
如今,在一项新的研究中,在美国怀特黑德生物医学研究所生物学教授David Bartel、研究生Sean McGeary和前研究生Kathy Lin的领导下,研究人员收集了关于6种miRNA的大量数据,并在此 基础上开发出一种针对所有单个miRNA的改进型预测模型。他们的发现为miRNA靶标预测提供了前所未有的准确性和粒度。相关研究结果近期发表在Science期刊上,论文标题为“The biochemical basis of microRNA targeting efficacy”。
为了了解miRNA的靶向作用,人们需要在miRNA序列中鉴定出可与miRNA结合的特定位点,并且他们还需要了解每个位点上的相互作用强度—结合亲和力(binding affinity)。通常,当miRNA 的前八个核苷酸中的至少六个与mRNA某位置上的互补核苷酸序列匹配时,miRNA将与mRNA结合。这两个序列就像成排的被推到一起的拼图块:如果每个拼图块都插入到相应的拼图块中,那么这 些成排的拼图块就组合成一个锁定的拼图---miRNA就可结合它的靶标。如果这些拼图块不能拼在一起,这些成排的拼图块就不能连接在一起。这些结合位点与miRNA的前8个核苷酸完全匹配, 被称为典型位点(canonical site),人们过去认为它们之间存在明显的层次关系,无论miRNA的身份如何,每种位点都会产生相似的抑制量。然而,McGeary并未观察到这种情形。
McGeary研究了六种miRNA,并开发了一种方法来测量每种miRNA与大量RNA序列的相对结合亲和力。这些测量以及McGeary从中进行的进一步计算形成了一个新的丰富的数据库,可用来改善 miRNA靶向预测。通过这些实验,这些研究人员发现经典位点的预期靶向层次结构并不适用于所有miRNA。实际上,单个miRNA对这种预期层次结构中较低的一个经典位点有更强的亲和力。此外 ,他们发现每个miRNA都有独特的非经典结合位点,其中的一些位点含有至少一个碱基错配但仍能够结合miRNA。他们发现,在许多情况下,尽管非经典位点的配对不完美或异常,但是miRNA与 一个非经典位点的结合要比与某些经典位点的结合更牢固。 Lin说:“作为人类,我们喜欢按照不同的特征将事物进行分类。但是要建立一种定量模型,你必须认识到每种miRNA与靶标之间的 相互作用是不同的。”
5.长达十年的科学争端终解决!两篇Science论文揭示人黏连蛋白通过挤压DNA环进行基因组组装
doi:10.1126/science.aaz3418; doi:10.1126/science.aaz4475
黏连蛋白(cohesin)是一种结合染色体的多亚基腺苷三磷酸酶复合物。在加载到染色体上后,它会产生DNA环来调节染色体功能。有人提出黏连蛋白通过环挤压来实现基因组组装,然而缺乏直接的证据来支持这一点。
在第一项新的研究中,奥地利维也纳生物中心分子病理学研究所(IMP)主任Jan-Michael Peters及其团队首次证实一种分子机器通过“环挤压(loop extrusion)”主动地和有目的地折叠DNA,从而在间期细胞中实现了多种重要功能。这种针对DNA成环(DNA looping,即形成DNA环)过程提出的新见解改变了关于基因组如何在细胞内组装的旧观点。这一发现阐明了生命的基本机制,并解决了长达十年的科学争端。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“DNA loop extrusion by human cohesin”。

图片来自Cees Dekker Lab TU Delft/Scixel。
Peters团队(包括Peters实验室资深博士后研究员Iain Davidson)能够在体外的一种简化系统中重建黏连蛋白的功能。因此,Davidson能够观察到单个黏连蛋白分子如何将DNA的单个片段快速地挤压成DNA环。他的发现影响深远,并以多种方式改变了对基因组的整体认识:(1)基因组不是静态的,而是高度动态的结构;(2)基因组DNA的折叠是一种受到主动调节的过程,它涉及通过挤压让DNA分子成环,并且许多DNA环在不断运动;(3)这种DNA成环是由黏连蛋白介导的,因此黏连蛋白必须是一种分子马达,类似于诸如肌球蛋白之类的其他马达蛋白;(4)黏连蛋白分子在DNA周围形成钩环状的环状结构,而且还必须通过多个结合位点动态连接到DNA上,这样才能够折叠DNA;正如去年所发现的那样,凝缩蛋白(condensin)也必须如此。
在第二项新的研究中,来自美国德克萨斯大学西南医学中心和德克萨斯州大学奥斯汀分校的研究人员利用单分子成像揭示重组的人黏连蛋白-NIPBL复合物通过挤压DNA环来压缩裸露的和核小体结合的DNA。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“Human cohesin compacts DNA by loop extrusion”。论文通讯作者为德克萨斯大学西南医学中心的Hongtao Yu博士和德克萨斯州大学奥斯汀分校的Ilya J. Finkelstein博士。
黏连蛋白开展的这种DNA压缩需要三磷酸腺苷(ATP)水解,并且对压力敏感。这种压缩过程以每秒0.5kb的平均速率处理数万个碱基。黏连蛋白对双链DNA的压缩表明黏连蛋白二聚体双向挤压DNA环。这些结果表明黏连蛋白-NIPBL复合物是一种由ATP驱动的能够进行DNA环挤压的分子马达。
总之,这两项新的研究表明与黏连蛋白调节姐妹染色单体黏连在一起的方式不同,这种蛋白在环挤压过程中似乎不会在拓扑结构上捕获DNA。这些结果为染色质组装的环挤压模型提供了直接证据,并表明基因组结构是高度动态变化的。
6.Science:新方法鉴定肠道微生物组产生的代谢物分子
doi:10.1126/science.aav1282; doi:10.1126/science.aaz4164
肠道菌群产生数百种以高水平存在于血液循环中的分子,它们的水平在不同人之间差异很大。这些分子是研究肠道微生物组与宿主相互作用的一个有前景的起点;少数经过详细描述的分子具有强效的免疫或代谢调节活性,并且是G蛋白偶联受体或核激素受体的配体。但是,在大多数情况下,这些分子的产生尚未与特定的细菌菌株或代谢途径相关联在一起,并且难以弄清每种分子对宿主生物学的贡献。
在一项新的研究中,来自美国斯坦福大学等研究机构的研究人员描述了一种基于CRISPR-Cas9的方法,用于在模式共生梭菌---生孢梭菌(Clostridium sporogenes)---中可靠地构建无需进行标记的不产生特定代谢物分子的细菌突变体。这种方法能够构建出多种发生突变的生孢梭菌菌株。相关研究结果发表在2019年12月13日的Science期刊上,论文标题为“Depletion of microbiome-derived molecules in the host using Clostridium genetics”。
通过使用这种方法敲除10种由产孢梭菌产生的分子---三甲胺、5-氨基戊酸酯、色胺、吲哚丙酸、异戊酸、2-甲基丁酸、异丁酸、异己酸、丙酸和丁酸,他们展示了它的实用性;我们通过液相色谱-质谱或气相色谱-质谱法验证体外培养的生孢梭菌的提取物中相应代谢物的缺乏来验证了每种敲除的成功性。
接着,他们将野生型生孢梭菌或者这5种缺乏相应代谢物的生孢梭菌突变体之一定植到无菌小鼠中,结果发现在定植野生型生孢梭菌的小鼠中,这些代谢途径的产物在宿主组织中积累,但是每种代谢物可通过定植相应的代谢途径突变体来加以剔除。通过比较接种野生型生孢梭菌定植的小鼠和接种缺乏产生分支短链脂肪酸---异丁酸、2-甲基丁酸或异戊酸---的生孢梭菌突变体的小鼠,他们发现这些丰富的肠道微生物组衍生性分子具有一种以前未知的调节免疫球蛋白A(IgA)浆细胞的活性。
7.Science:成功诱导免疫系统产生关键抗体来抵御HIV的感染
doi:10.1126/science.aay7199; doi:10.1126/science.aaz8647
近日,一项刊登在国际杂志Science上的研究报告中,来自杜克大学医学中心的科学家们通过研究清除了HIV疫苗开发过程中遇到的主要障碍,同时在动物模型中也证实有效,即诱导短时效抗体增殖来成为有效抵御HIV的中坚力量。

图片来源:NIAID。
医学博士Barton F. Haynes表示,如今我们并没有HIV疫苗的原因在于机体免疫系统无法制造出能够中和HIV的特殊抗体类型,这项研究中,我们通过对免疫系统进行工程化操作来使其产生一种特殊环境从而制造出合适的抗体。基于此前多年的研究结果,如今研究人员阐明了广谱中和性抗体(bnAbs)如何及何时在HIV感染者机体中出现,以及何时能够有效阻断抗体增殖来抑制病毒。
其中一个问题在于免疫系统,其会将bnAbs视为一种危险并关闭bnAbs的产生过程;另外一个问题就是中和性抗体需要在其遗传组成上发生罕见改变,这在重要的B细胞多样化过程中并不会产生。这项研究中,研究人员追踪了相关突变,随后他们对一种HIV蛋白进行工程化修饰,靶向作用病毒包膜上V3聚糖区域位点,从而使其能够优先与携带必要突变的抗体相结合。利用表达人类中和性抗体前体的小鼠模型进行研究,研究人员发现,免疫原能够诱导B细胞谱系来经历不可能的突变从产生广谱性抗体。
研究者Alt表示,我们能够制造出表达人类广谱性中和抗体的小鼠模型,这就能为我们提供强大的新型模型系统,在该系统中,我们就能够反复检测实验性的HIV疫苗;第二种谱系的bnAbs则会经历一种不可能的突变,其会结合病毒外膜上名为CD4结合位点的不同区域,当研究人员重建了抗体历史后,他们开发出了第二种免疫原,在非人类灵长类动物中进行测试后,研究者发现,这种免疫原同样会选择必要的突变,这就能够帮助开发潜在的CD4结合位点中和性抗体。
8.Science:科学家成功利用九种酶类的链式反应来制造HIV药物islatravir
doi:10.1126/science.aay8484; doi:10.1126/science.aaz7376
近日,一项刊登在国际杂志Science上的研究报告中,来自默克和克迪科思公司的研究人员通过研究成功利用一种九种酶的链式反应方法来制造HIV药物—islatravir;文章中,研究人员描述了他们工作的灵感以及最终产品的效果。
研究者指出,多种多样的天然产物是很多基本元件进行生物合成的结果,很多有机体会利用酶类的选择性来完成每一步过程,而这通常会涉及一种多步的连锁反应,其中第一个反应就会被用作下一步反应的基础。基于此,研究人员就开始寻找能够制造HIV实验性药物islatravir的方法,首先他们将目光锁定到细菌的核苷补救途径(NSP,nucleoside salvage pathway)上,该过程主要参与从RNA降解到DNA的碱基和核苷的回收,在这一过程中有三种主要的酶类,即脱氧核糖-5-磷酸醛缩酶(DERA,deoxyribose-5-phosphate aldolase)、戊磷酸变位酶(PPM,phosphopentomutase)和嘌呤核苷磷酸化酶(PNP,purine nucleoside phosphorylase)。
为了利用相同的过程来开发药物islatravir,研究者就需要利用一种酶类的特殊特性,即可逆特性,酶类的这种特性能够使其有别于传统的催化剂。研究人员必须找到一种方法来让上述三种酶类接受非天然的底物,解决的方法就是从大肠杆菌的RNP和PPM中借用反向的NSP,从S. halifaxensi菌中借用DERA,为了使其能够发挥作用,研究人员还必须对酶类进行工程化修饰来使其变得更具活性,这项工作还涉及到优化另外两种酶,并在自然状态下使用其它四种酶类。最后的结果就是涉及九种酶类的级联反应,最终就能够制造出药物islatravir。
9.Science:重磅!科学家在眼睛中鉴别出了能帮助大脑区分昼夜的特殊细胞!
doi:10.1126/science.aaz0898
夜间的强光会打乱机体正常的昼夜节律(circadian rhythms),从而引发失眠;实际上,昼夜几率在机体健康上扮演着重要的角色,干扰昼夜节律常常会增加多种疾病的发病风险,比如癌症、心脏病、肥胖、抑郁症等,因此,理解人眼感知光线的机制或能帮助有效预防抑郁、促进夜间睡眠并保持机体健康的昼夜节律。
近日,一项刊登在国际杂志Science上的研究报告中,来自索尔克研究所的科学家们通过研究在眼睛中发现了三种细胞类型,其能帮助检测光线,并使大脑的昼夜节律与周围的光线保持一致,这项研究中,研究者首次评估了这种名为本质感光性视网膜神经节细胞(ipRGCs,intrinsically photosensitive retinal ganglion cells)的细胞对光线的反应,以及其对机体健康的重要应用。
文章中,研究者揭示了三种类型的ipRGCs细胞,第一种细胞对光的响应速度较快,但关闭时间较长,而第二种细胞开启和关闭时间均较长,第三类细胞在灯光非常明亮时才会产生响应,但其开启非常迅速且能在灯光消失后立马关闭。理解每一种ipRGCs细胞的功能或能帮助研究人员设计出更好的光照条件或疗法来开启/关闭这些细胞活性。
研究者指出,ipRGCs细胞负责将光信号传输到大脑中,甚至是在缺乏视杆细胞和视锥细胞的人群中也需要向大脑中传递图像。在携带有正常视杆细胞和视锥细胞的人群中,ipRGCs细胞实际上能与这些细胞密切合作,本文研究表明,ipRGCs细胞能结合视杆和视锥细胞所检测到的光信号,从而为我们所看到的信息增加亮度和对比度信息。
10.Science:重大进展!新研究发现最常见的儿童肾癌的遗传根源
doi:10.1126/science.aax1323
儿童肾癌很少见。维尔姆斯肿瘤(Wilms' tumour)是儿童肾癌的最常见类型,主要影响五岁以下的儿童。它在英国每年诊断出大约80例。十分之九的病例可以通过外科手术去除受影响的肾脏,同时进行化疗,有时还进行放疗,就可被治愈。通常而言,这种癌症仅影响一个肾脏,但是在大约10%的病例中,两个肾脏都受到影响。据推测,维尔姆斯肿瘤的病因是在生命最初几周内的肾脏发育过程中,一个细胞出现了“差错”。
在一项新的研究中,英国研究人员发现了儿童肾癌维尔姆斯肿瘤的最早遗传根源。通过比较来自正常肾脏组织和肿瘤的基因组序列之后,他们鉴定出看上去正常的肾脏组织块实际上携带导致维尔姆斯肿瘤的DNA变化。相关研究结果发表在2019年12月5日的Science期刊上,论文标题为“Embryonal precursors of Wilms tumor”。

图片来自CC0 Public Domain。
这项研究揭示了一种癌症可以在生命的最初几周内形成的新途径,在这种途径中,早期的细胞获得与癌症相关的修饰,并且在肾脏发育时增殖。了解这种癌症的根源有望改善治疗,并有助阻止维尔姆斯肿瘤复发。这一发现还增加了有朝一日有可能在肿瘤产生之前筛查这种癌症的可能性。(生物谷 Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


