Cell:通过分析全球60个城市的近5000份样本,发现大约1.2万种之前从未发现的细菌和病毒
来源:本站原创 2021-05-31 21:19
2021年5月31日讯/生物谷BIOON/---国际MetaSUB联盟(International MetaSUB Consortium)一项由威尔康奈尔医学院的研究人员领导的追踪微生物的全球努力。根据国际MetaSUB联盟的一项新的研究,在2015年至2017年期间从世界各地的公共交通系统和医院的取样中收集的大约12000种细菌和病毒以前从未被鉴定过。相关
2021年5月31日讯/生物谷BIOON/---国际MetaSUB联盟(International MetaSUB Consortium)是一项由威尔康奈尔医学院的研究人员领导的追踪微生物的全球努力。根据国际MetaSUB联盟的一项新的研究,在2015年至2017年期间从世界各地的公共交通系统和医院的取样中收集的大约12000种细菌和病毒以前从未被鉴定过。相关研究结果于2021年5月26日在线发表在Cell期刊上,论文标题为“A global metagenomic map of urban microbiomes and antimicrobial resistance”。

这些作者在三年时间里收集了近5000份样本,这些样本遍布六大洲32个国家的60个城市。他们使用一种称为鸟枪测序的基因组测序技术分析了这些样本,以检测各种微生物的存在,包括细菌、古生菌(与细菌不同的单细胞生物)和使用DNA作为遗传物质的病毒。(其他类型的病毒使用RNA作为其遗传物质,如引起COVID-19的冠状病毒SARS-CoV-2,在这次COVID-19大流行前的研究中使用的DNA分析方法不会被发现)。
这一领域的研究对于检测已知和未知感染的爆发以及研究不同城市环境中抗生素抵抗性微生物的流行具有重要意义。
这项新的研究发现了10928种病毒和748种细菌,这些病毒和细菌在任何现有的参考数据库中都不存在。
通过收集微生物样本并分析它们的基因---统称为微生物组(microbiome),这些作者希望能够更多地了解生活在人类中的细菌、病毒和其他微生物。例如,这项新的研究可能有助于识别抗生素抵抗性菌株的出现。仅仅依据基因序列预测抗生素抵抗性是具有挑战性的,但是这些作者能够绘制一些已知与抵抗性有关的基因,定量确定它们的丰度,并确认这些基因标志物赋予抵抗性的能力。他们发现,一些城市比其他城市有更多的抵抗性基因,而且其中的一些基因可能有城市特有的特征。
抗菌剂抵抗性仍然是一个主要的全球健康挑战。虽然还需要开展进一步的研究,但是这个数据集显示了微生物组图谱绘制和监测的价值和潜力,以及它可以为医生、科学家和公共卫生官员提供的新见解。
此外,对微生物制造的小分子和蛋白质的了解也可能导致发现新的抗生素以及其他有可能被开发为药物的分子。目前正在使用的许多抗生素和药物都来自于微生物。对新的微生物物种的发现也可能导致新的实验室工具和方法,比如使用称为CRISPR的分子编辑工具的新方法。在这项新的研究中,这些作者发现了838532个新的CRISPR阵列(在细菌内发现的病毒DNA片段)以及430万个新的肽。
由于这些取样工作,这些作者仅通过对一个人鞋子上的DNA进行测序,就可以预测此人的居住地,准确率约为90%。他们发现许多因素会影响一个城市的微生物组,包括总人口和人口密度、海拔、靠近海洋和气候。比如,沿海地区的样本可能含有嗜盐的微生物,而来自人口稠密的城市的样本可能显示出惊人的生物多样性。关于这些不同特征的发现可能使未来的法医研究成为可能。
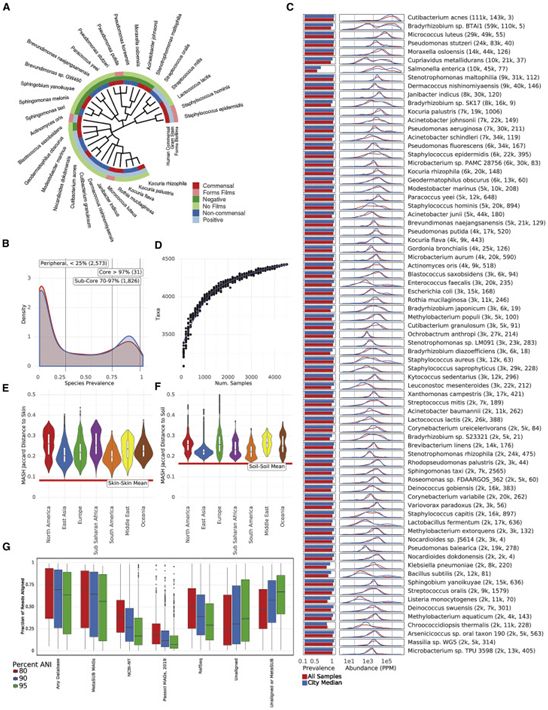
核心微生物组,图片来自Cell, 2021, doi:10.1016/j.cell.2021.05.002。
这些作者于2013年开始收集和分析纽约市地铁系统的微生物样本。在他们发表了称为PathoMap的首批研究结果后,世界各地的科学家们纷纷与他们联系,希望为自己的城市开展类似的研究。国际上的兴趣激发了他们创建国际MetaSUB联盟。如今,MetaSUB联盟继续成长,并已扩大到从空气、水和污水以及硬表面收集的RNA和DNA样本。它获得了一项500万美元的拨款,用于美国三个州(佛罗里达州、纽约州和威斯康星州)的废水测序和病毒追踪,这也是美国疾病控制与预防中心(CDC)新建立的国家废水监测系统(NWSS)的一部分。
这些作者还监督每年6月21日举行的全球城市取样日(Global City Sampling Day, gCSD)等项目,并进行了广泛的研究,包括在2016年夏季奥运会之前、期间和之后对里约热内卢进行的全面微生物分析。这项新研究中分析的许多样本是在2016年和2017年的全球城市采样日收集的。纽约市的采样工作是在威尔康奈尔医学临床与转化科学中心(CTSC)的支持下进行的。这些作者目前正在为今年的研究活动做准备。
这些作者指出,尽管样本是在世界各地收集的,但大部分的分析都是在威尔康奈尔医学院完成的。序列的分析和组合还利用了匹兹堡超级计算中心的极端科学与工程发现环境(XSEDE)超级计算机Bridges和Bridges-2。瑞士的MetaSUB研究人员利用这些组合和原始数据建立了一个可搜索的全球DNA序列门户网站(MetaGraph),该门户网站对所有已知的基因序列(包括MetaSUB数据)进行索引。该门户网站将任何已知的或新发现的遗传成分映射到它们在地球上的位置,可以帮助发现新的微生物相互作用和假定的功能。
未来和正在进行的研究将用长读数和空间成像方法研究RNA和DNA,以及追踪来自全球场所的代谢物,并继续更新行星尺度的遗传图谱。(生物谷 Bioon.com)
参考资料:
David Danko et al. A global metagenomic map of urban microbiomes and antimicrobial resistance. Cell, 2021, doi:10.1016/j.cell.2021.05.002.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


