Molecular Cancer: 单细胞RNA-seq分析发现一种非编码RNA介导肝癌对索拉非尼治疗的耐药性
来源:本站原创 2022-01-22 20:35
索拉非尼是一种多激酶抑制剂,是fda批准的一线药物,用于治疗不能切除的肝细胞癌(Hcc)。不幸的是,这些患者的治疗效果微乎其微,总体生存期仅提高了3个月。在初步应答后,大多数肝癌患者出现抗药性和疾病进展。
索拉非尼是一种多激酶抑制剂,是FDA批准的一线药物,用于治疗不能切除的肝细胞癌(Hcc)。不幸的是,这些患者的治疗效果微乎其微,总体生存期仅提高了3个月。在初步应答后,大多数肝癌患者出现抗药性和疾病进展。虽然已经报道了PI3K/Akt和MAPK/ERK通路的串扰或EMT激活等多种机制,但导致索拉非尼耐药的主要驱动因素仍然不清楚。
最近,快速发展的单细胞转录组分析技术已经被用来综合评估肿瘤微环境的遗传学和肿瘤内异质性的机制,这可能影响抗癌治疗反应。这项技术具有比批量测序技术更高的转录分辨率和灵敏度的潜力。单细胞分析还能够深入询问数百名癌症驱动因素,并识别具有特定基因修饰或表达谱的单个细胞或基因,这可能有助于治疗耐药性的发展。
图片来源: https://doi.org/10.1186/s12943-021-01473-w
在长期接触亲本Hep3B和Huh7(Hep3B-P和Huh7-P)细胞后,作者成功地建立了Hep3B和Huh7索拉非尼耐药细胞(Hep3B-R和Huh7-R)。Hep3B-R和Huh7-R细胞的IC50分别是Hep3B-P和Huh7-P细胞的近2倍和2.3倍。有趣的是,Hep3B-R和Huh7-R细胞比Huh7-P细胞有更强的自我更新能力,形成明显更大和更多的肿瘤球体。
在Hep3B-R和Huh7-R细胞中发现假定的肝癌干细胞群显著增加。为了评估茎类表型的丰富性,采用纳米串分析方法研究了21个茎类相关基因的靶向mRNA表达。正如预期的那样,在Hep3B-R和Huh7-R细胞中观察到这些基因中的大多数显著到适度上调。
在Hep3B-R和Huh7-R细胞中,EMT或干细胞标志如Vimentin、N-cadherin、Notch2、Notch3、AFP和Oct4的蛋白水平均上调,而E-cadherin的蛋白水平下调。这些数据表明,肝癌细胞可能经历了EMT,这与索拉非尼治疗后获得的干性特征有关。
群体水平上stemness/EMT表型的丰富促使我们假设它们的诱导可能是索拉非尼耐药的主要原因。为了验证这一假设,使用10X基因组铬系统对Huh7-P和Huh7-R细胞进行了单细胞RNA测序。经过对低质量细胞的严格过滤,4776个细胞被选择用于随后的单细胞测序数据分析。
尽管显示茎/EMT基因上调,Huh7-R细胞显示出增殖标记MKI67和细胞周期相关基因的下调,这表明索拉非尼处理可能会丰富细胞周期缓慢或处于休眠状态的耐药细胞子集。这与静止的干细胞样细胞保持在休眠或缓慢分裂状态,但随后在癌症治疗后扩张和分裂的现象是一致的。为了进一步证实这些细胞的存在,作者发现一小部分Huh7-P细胞与Huh7-R细胞群聚集在一起。
在与索拉非尼耐药相关的基因中,ZNFX1反义RNA 1(ZFAS1)是Huh7-R细胞中表达最高的基因。ZFAS1作为一种新的调控因子,在HCC(n=10)中过表达,据报道与多种肿瘤的生长和转移有关。因此,作者通过分析公开可用的数据集,确定了ZFAS1在HCC中的表达与临床病理特征之间的相关性。在这项分析中,作者利用GEPIA数据集和加州大学圣克鲁斯分校(UCSC)的XENA项目发现,ZFAS1在HCC和复发肿瘤中的表达显著上调,与匹配的正常肝脏相比。
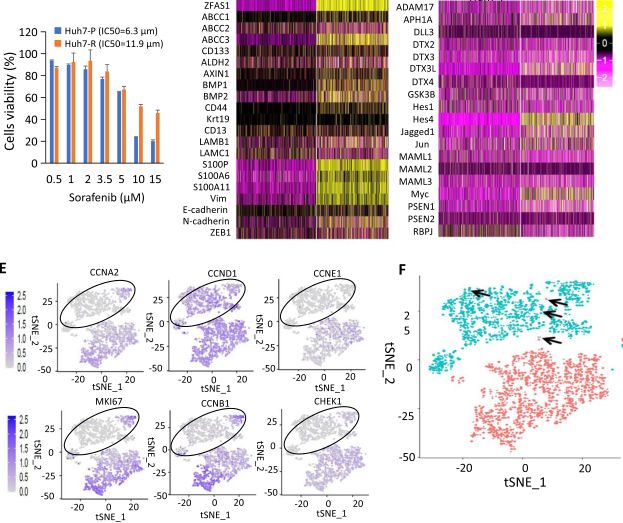
索拉非尼耐药细胞中茎/EMT基因特征的单细胞富集
图片来源: https://doi.org/10.1186/s12943-021-01473-w
综上所述,这项研究证明了茎性和EMT在单细胞水平上驱动索拉非尼耐药性的确切作用。肝癌细胞群体中预先存在一小部分具有内在索拉非尼耐药的静止干细胞样细胞,并在长期接触索拉非尼后变得丰富。这一数据提供了新的见解,表明休眠的干细胞样细胞可能是促进索拉非尼耐药性发展的主要罪魁祸首。
因此,杀死静止的肿瘤干细胞可能是克服索拉非尼耐药性的一种策略。作者还发现了一个新的非编码LncRNA,ZFAS1,它与HCC的增殖和茎性特征的维持密切相关。因此,沉默ZFAS1是克服索拉非尼耐药性的另一种策略。(生物谷 Bioon.com)
参考文献
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。



