科研人员开发临床错义突变致病性预测新工具
来源:网络 2024-10-06 09:24
该工作通过加入相分离特征,开发了对临床错义突变致病性预测的新工具,提高了对IDR区域突变位点致病性预测的准确性。
蛋白质序列中的内在无序区域(Intrinsically disordered regions, IDRs) 一直以来是一个极具挑战性的领域。IDRs存在于超60%的人源蛋白中,而且在人类疾病相关的错义突变 (missense mutations)中,约25%就发生在IDR区域。然而,由于研究IDR中的错义突变非常困难,导致许多IDR突变在ClinVar数据库中被标记为“意义未明突变”(Variants of uncertain significance, VUSs)。这些大量的VUS被判定为“意义未明”的原因在于对于IDR生理功能认知的缺乏。
近年来,IDRs被广泛报道通过相分离(phase separation)形成无膜生物分子凝聚体(condensate)。这些凝聚体(condensates)在许多生物过程中发挥重要作用,例如细胞核内的核仁和转录工厂、细胞质中的应激颗粒以及内耳毛细胞的蛋白质高密度区域等。IDRs中的错义突变可以通过改变相分离行为来扰乱蛋白质的正常生理功能。

上海交通大学Bio-X研究院陆青/贺光/石毅团队在Nature Communications发表题为Decoding Missense Variants by Incorporating Phase Separation via Machine Learning的研究论文。该工作通过加入相分离特征,开发了对临床错义突变致病性预测的新工具,提高了对IDR区域突变位点致病性预测的准确性。
研究者首先利用PhaSepDB以及LLPSDB中通过实验验证的错义突变数据训练了一个名为PSMutPred的计算工具,来预测突变影响蛋白相分离的倾向。现有的基于序列的相分离预测算法仅基于已知的相分离蛋白集合进行训练,PSMutPred比现有算法相比展现出更强的鲁棒性,并能够预测突变是增强还是减弱相分离。研究者还选取了他们感兴趣的耳聋相关基因EPS8进行实验验证分析。随后提出利用相分离特征来改进对IDR疾病变异的预测,将PSMutPred得分等多个相分离相关特征整合到现有的无监督致病性预测方法中(包括EVE和ESM1b)。观察到预测准确性显著提高,尤其是对于低保守性IDR(pLDDT<50)中的变异,精确率-召回率曲线下面积(AUPR)提高了约10%,证明了该方法提高了对IDR区域突变位点致病性预测的准确性。
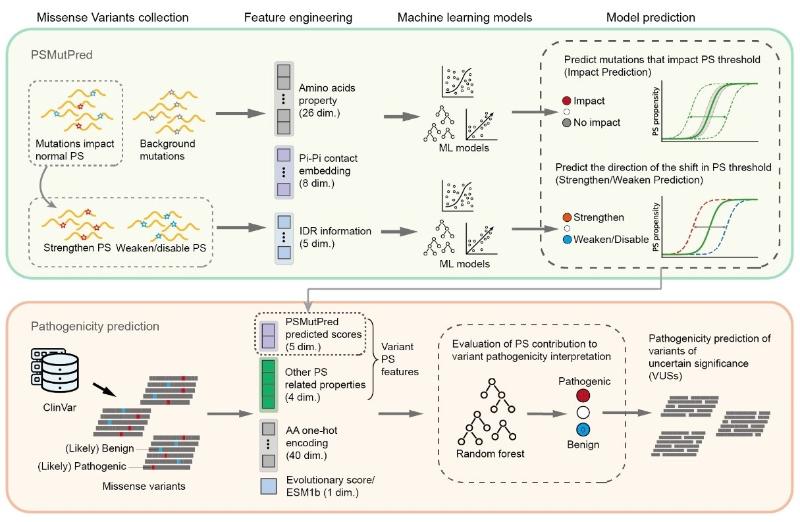
图1:算法概要
该方法已申请了国家专利保护,研究者同时建立了网站(www.psmutpred.online)可以查询并下载人类蛋白质组所有可能突变的预测结果,包含预测突变影响相分离的打分(IP分数)以及突变增强或减弱相分离的预测打分(SP分数)。
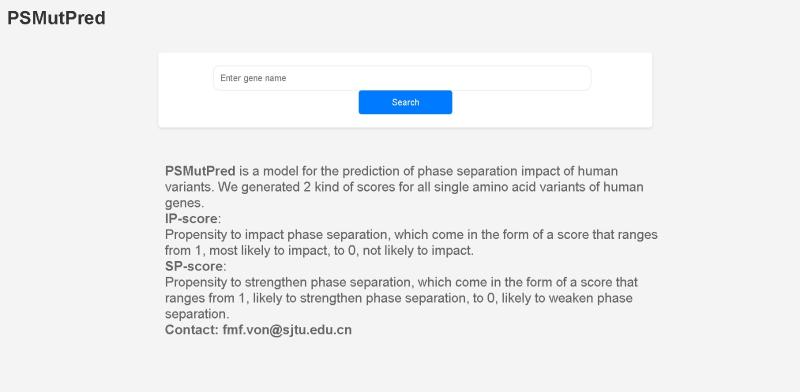
图2:PSMutPred分数查询界面
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


