Nat Cell Biol:单细胞新RNA测序在单次转录爆发层面上揭示了转录原理
来源:生物探索 2024-09-26 13:13
通过改进的单细胞新RNA测序技术(NASC-seq2),研究深入揭示了转录爆发的动力学原理,发现了合成率在决定转录爆发规模中的关键作用。
基因的表达是生命活动的核心过程之一,通过精确的基因调控机制,细胞能够响应外界环境的变化并维持自身的稳态。这种调控不仅影响个体发育,还对疾病的发生发展起着至关重要的作用。转录是基因表达调控的一个关键步骤,而转录的动态过程直接影响着基因产物的数量和功能。在基因表达过程中,RNA聚合酶II负责将DNA模板转录为RNA。然而,越来越多的研究发现,基因转录并非连续进行,而是以短暂的活跃期和静止期交替出现的方式进行,这种现象被称为转录爆发【1】。
尽管转录爆发已被广泛研究,但仍有几个关键问题未得到解答:所有基因是否都以爆发的方式转录,或者是否有一部分基因呈现连续表达?基因之间的转录爆发是否独立进行,还是存在协同爆发的现象?
近日,来自瑞典卡罗林斯卡学院的Rickard Sandberg研究团队在Nature Cell Biology上发表了题为Single-cell new RNA sequencing reveals principles of transcription at the resolution of individual bursts的文章,通过对单细胞RNA测序数据的转录爆发分析,揭示了哺乳动物基因转录主要以独立的爆发形式进行,并且合成率是决定爆发规模的关键因素。

实时成像实验和单细胞RNA测序(scRNA-seq)提供了转录爆发的直接证据【2,3】。然而,现有方法在推断转录动力学参数时存在局限性【4】,尤其是难以区分合成率和转录关停率(synthesis and closing rates)。合成率决定了每次转录爆发期间产生的RNA分子数量,即爆发的规模(burst size);而转录关停率影响的是转录的静止期持续时间,即爆发的频率。这需要进一步优化单细胞层面的新RNA测序技术,从而更精确地解析转录爆发现象。
研究团队开发了NASC-seq2,这是一种改进的新RNA测序方法。该方法通过4-硫代尿苷(4sU)标记新合成的RNA【5】,并结合计算分析来区分新RNA和已有RNA。在实验中,研究者对10,000个原代小鼠成纤维细胞进行了单细胞新RNA测序,以推断转录爆发的动力学参数。NASC-seq2方法显著提高了检测敏感性和细胞处理通量,使得在每个细胞中检测到的基因数量大幅增加。
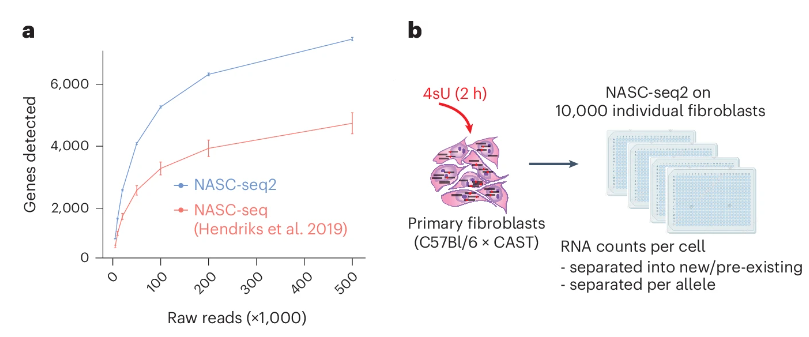
图1:a. 随着测序读取数增加,每个K562细胞检测到的基因数量变化。b. 针对F1原代成纤维细胞的大规模NASC-seq2实验示意图(Credit: Nature Cell Biology)
然后,研究人员对标记的新RNA分子进行定量分析,以推断出每个基因在特定时间内的RNA合成和转录停止的频率。结果表明,合成率(ksyn)是决定每次转录爆发期间产生的RNA分子数量的关键因素,即爆发的规模,而关停率(koff)则相对恒定。这一发现揭示了不同基因在表达过程中如何通过调整合成率来控制转录爆发的大小。
为了进一步探讨基因之间的转录爆发是否存在协同作用,研究者对全基因组范围内的基因进行了分析。他们利用NASC-seq2生成的高分辨率新RNA数据,观察基因对在同一细胞中是否同时发生转录爆发。研究结果显示,大多数基因的转录爆发是独立进行的,只有少数基因对,特别是那些位于基因组近端的同源基因对,表现出轻微的协同爆发现象。
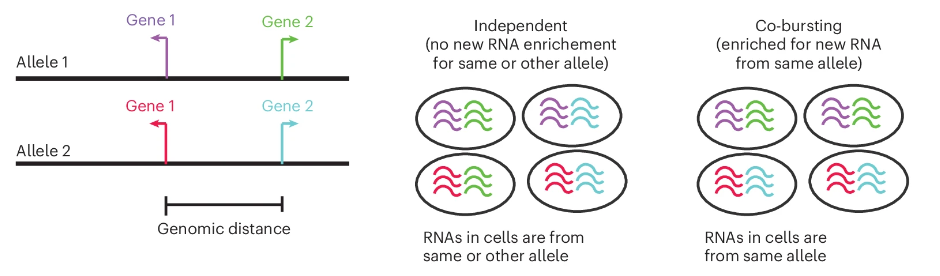
图2:基因组区域中两个相邻基因:如果所有等位基因和基因都是独立转录的,在四个细胞中获得的新RNA的示意图(左),如果相邻基因从同一个等位基因协同爆发,在四个细胞中获得的新RNA的示意图(右)(Credit: Nature Cell Biology)
参考文献:
1. McKnight, S. L. & Miller, O. L. Post-replicative nonribosomal transcription units in D. melanogaster embryos. Cell 17, 551–563 (1979).
2. Chubb, J. R., Trcek, T., Shenoy, S. M. & Singer, R. H. Transcriptional pulsing of a developmental gene. Curr. Biol. 16, 1018–1025 (2006).
3. Dar, R. D. et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl Acad. Sci. USA 109, 17454–17459 (2012).
4. Rodriguez, J. & Larson, D. R. Transcription in living cells: molecular mechanisms of bursting. Annu. Rev. Biochem. 89, 189–212 (2020).
5. Hendriks, G.-J. et al. NASC-seq monitors RNA synthesis in single cells. Nat. Commun. 10, 3138 (2019).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


