科研人员开发相分离关键氨基酸鉴定算法PSPHunter
来源:中山大学 2024-04-24 14:14
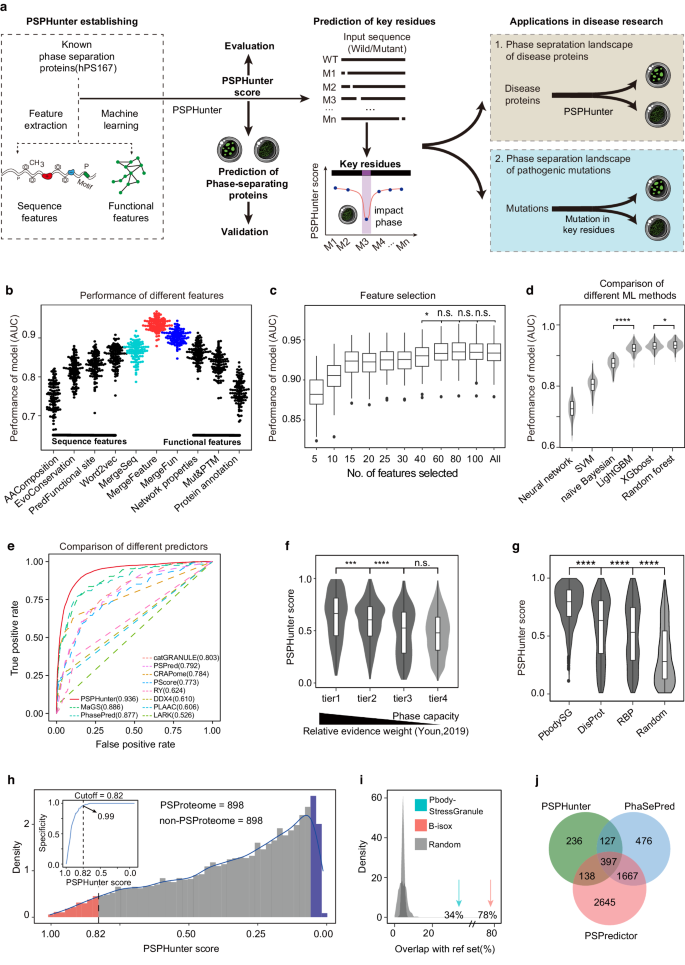
该研究报道了一种基于多信息融合的机器学习模型PSPHunter,可用于鉴定相分离蛋白和相分离关键氨基酸。
中山大学丁俊军课题组在Nature communications发表了题为Precise prediction of phase-separation key residues by machine learning的研究成果。该研究建立了一种机器学习算法PSPHunter(Phase-Separating Protein Hunter),通过整合相分离蛋白的序列和功能特征来预测相分离蛋白并识别关键氨基酸,进一步量化疾病相关突变对相分离的影响,剖析相分离与疾病之间的关系。
在该项研究中,研究人员首先整合现有的相分离蛋白数据以及相分离相关序列和功能特征,建立了机器学习算法PSPHunter,基于算法评分筛选出由898个相分离蛋白构成的相分离蛋白组PSProteome。进而,运用滑动窗口(Sliding-window)策略针对蛋白质序列进行连续截断,定义截断后使相分离能力下降最多的片段为关键氨基酸(Key residues)。通过现有研究与实验两种途径验证了算法可靠性之后,进一步解析了相分离关键氨基酸的内在特征,并评估了突变对于相分离的影响,对于相分离和致病突变之间的关系进行了全面解析。
综上所述,该研究报道了一种基于多信息融合的机器学习模型PSPHunter,可用于鉴定相分离蛋白和相分离关键氨基酸。利用该算法建立相分离蛋白质组、相分离关键氨基酸图谱和相分离功能障碍致病突变图谱等资源,有助于阐明相分离蛋白的功能,探索相分离在转录调控、细胞命运转变和疾病发展中的机制。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


