Nat Cell Biol丨蓝勋和孟安明合作开发高通量、低成本SPATAC-seq技术并绘制斑马鱼早期胚胎发育染色质可及性图谱
来源:生物探索 2024-07-16 09:39
SPATAC-seq是一种基于单细胞组合索引(Single-cell combinatorial indexing)原理设计开发的单细胞染色质可及性技术。
胚胎发育过程中,具有全能性的受精卵迅速分裂和分化为众多细胞谱系并形成各个组织和器官,伴随着细胞内染色质结构的生物化学和结构以及基因表达的高度动态变化【1-2】。解析这一过程中不同细胞类型的发育路径以及决定不同细胞命运的分子调控机制,一直以来是发育生物学中非常重要的研究内容。单细胞表观基因组学和转录组学测序技术通过在单细胞分辨率下全面地描绘胚胎发育过程中细胞谱系的多样性以及基因表达的动态调控【3-5】,极大地改变了人们研究胚胎发育的范式。
斑马鱼(Danio rerio)与人类在基因上有着高度同源性,是一种优良的研究胚胎发育的模式脊椎动物【6】。在过去十年中,许多研究团队绘制了大量的斑马鱼胚胎发育各个阶段的表观基因组和转录组图谱。DANIO-CODE等项目基于这些图谱全面地注释在斑马鱼胚胎发育过程中发挥功能的顺式调控元件(CREs, cis-regulatory elements)【7-12】。近几年,已有多个课题组绘制了斑马鱼早期胚胎发育过程中的单细胞转录组学图谱,并揭示了斑马鱼发育过程中极为复杂的细胞分化谱系【3-5, 12】。scATAC-seq是一种用于分析全基因组染色质可及性区域和研究细胞类型特异性转录调控的工具,可以提供与转录组互补的表观调控信息,有助于我们更为深刻地理解胚胎发育过程中决定不同细胞谱系命运决定的分子调控机制。然而目前在斑马鱼胚胎发生过程中,决定细胞命运和功能的染色质可及性动态变化特征的研究尚不完善。通过在单细胞水平上系统地构建,斑马鱼胚胎发育过程中各个发育阶段的单细胞染色质可及性图谱,可以更好地让我们了解斑马鱼胚胎发育过程中细胞命运决定背后复杂的调控机制。
2024年7月8日,清华大学医学院蓝勋副教授团队与清华大学生命学院孟安明教授团队合作,在Nature Cell Biology上在线发表了题为Mapping the chromatin accessibility landscape of zebrafish embryogenesis at single-cell resolution by SPATAC-seq的文章。此研究开发了一种基于组合索引原理的,低成本、测序文库兼容性高的超高通量单细胞染色质可及性测序技术,SPATAC-seq,并用此技术对斑马鱼胚胎4 hpf至72 hpf内20个连续发育时期的80多万个单细胞进行测序,绘制了斑马鱼胚胎发育染色质可及性图谱ZEPA(Zebrafish Embryogenesis single-cell oPen chromatin Atlas),系统性地重构出发育过程中604种细胞状态的发育路径(developmental trajectory),并识别出不同细胞状态下基因组中总计约96万个CREs。

SPATAC-seq是一种基于单细胞组合索引(Single-cell combinatorial indexing)原理设计开发的单细胞染色质可及性技术(图1)。首先,固定的细胞核在48个不同的PCR孔中通过48种不同的Tn5转座酶进行转座反应,第一轮引入共48种标签;然后收集所有孔中的细胞核并依次随机分配到第二个和第三个48孔板中,通过T4连接酶将第二轮和第三轮48种标签依次连接至转座子i5和i7端的两个磷酸化位点;最后,再次收集所有细胞核并重新分配到96孔板进行PCR反应,引入测序接头、P5/P7以及第四轮标签。理论上,单次SPATAC-seq实验可以处理百万级别的细胞核,并且可以通过增加每一轮标签的数量来进一步提高细胞通量。SPATAC-seq产生的单细胞数据与Bulk ATAC-seq和DNase-seq的数据以及之前发表的单细胞测序数据集都具有很强的正相关性,说明由SPATAC-seq产生的单细胞数据是真实可靠的。
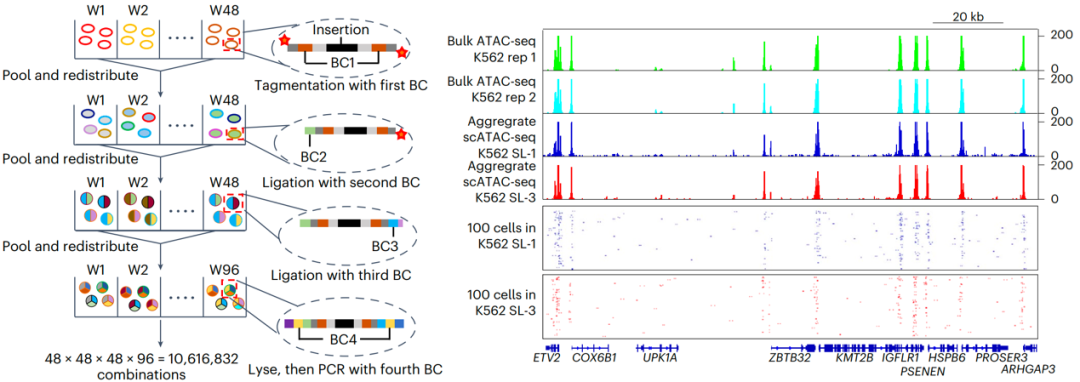
图1 SPATAC-seq实验原理流程及其在K562细胞中的数据质量(Credit: Nature Cell Biology)
为了全面地解析斑马鱼胚胎发育过程中基因组的可及性区域及其动态变化特征,研究者使用SPATAC-seq对4 hpf至72 hpf的20个发育时间点的斑马鱼胚胎进行单细胞染色质可及性测序,覆盖了斑马鱼胚胎发育的囊胚后期、原肠期、体节期、咽囊期和孵化期,一共获得约81万个高质量的单细胞染色质可及性数据(图2)。基于标签转移(Label transfer)等方法,研究者识别出604种细胞状态,并通过k-NN的方法首次在染色质可及性层面上系统性地重构出604种细胞状态的发育路径,相比于之前基于单细胞转录组数据构建的发育路径更为全面【14】,例如研究鉴定出慢肌细胞可能起源于中胚层近轴细胞。
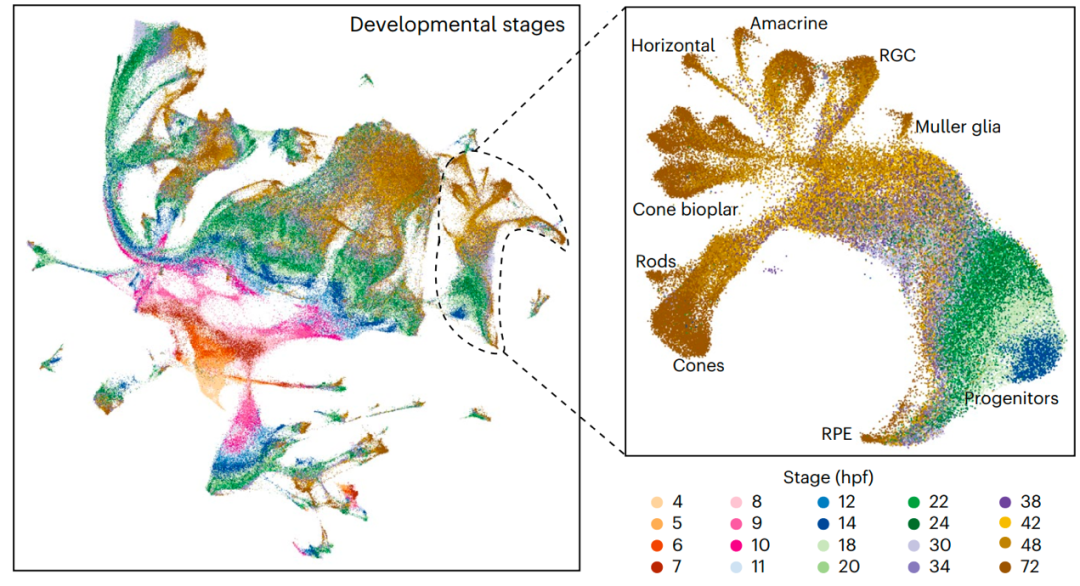
图2 斑马鱼4 hpf至72 hpf的单细胞染色质可及性图谱及retina细胞的分化轨迹 (Credit: Nature Cell Biology)
随后,研究者利用MACS2对从上述20个发育时间点的604种细胞状态进行信号峰的富集分析,绘制出斑马鱼胚胎发育过程中约96万个CREs的综合图谱(覆盖了斑马鱼参考基因组35.7%的区域),同时通过保守性等分析方法表征了这些CREs的真实性和潜在的生物学功能。接下来研究人员利用斑马鱼增强子(enhancer)报告基因实验验证了约65%的细胞类型特异性CREs可以驱动EGFP在斑马鱼体内特定组织或器官中表达,该结果表明部分ZEPA cCREs具有类似于增强子调控对应区域上下游靶基因转录的功能。进一步应用k均值聚类算法,研究者识别出10万多个在胚胎水平上与发育相关的呈现高度动态变化的CREs。为了解析细胞类型特异性的转录调控机制,研究者还利用chromVAR推断并分析了各细胞类型中转录因子的活性,从而识别出如神经嵴细胞中sox10和内皮细胞中etv2等关键调控因子。
最后,研究者以色素细胞和脊索细胞为例,探索了驱动色素前体细胞向黑色素细胞、黄色素细胞和虹色素胞分化,以及脊索细胞向col2a1a阳性及ngs阳性脊索细胞分化的细胞亚型特异性的基因调控程序。
以上这些研究结果,不仅展示了SPATAC-seq技术在单细胞染色质可及性分析中的强大应用潜力,还为研究斑马鱼胚胎发育过程中基因调控网络提供了宝贵的基因组资源。这些发现将有助于未来深入探索不同生物的发育机制和细胞命运决定过程中关键调控因子的作用。
参考文献:
1 Levine, M. & Davidson, E. H. Gene regulatory networks for development. Proceedings of the National Academy of Sciences 102, 4936-4942, doi:doi:10.1073/pnas.0408031102 (2005).
2 Moris, N., Pina, C. & Arias, A. M. Transition states and cell fate decisions in epigenetic landscapes. Nature Reviews Genetics 17, 693-703, doi:10.1038/nrg.2016.98 (2016).
3 Wagner, D. E. et al. Single-cell mapping of gene expression landscapes and lineage in the zebrafish embryo. Science 360, 981-987, doi:10.1126/science.aar4362 (2018).
4 Farrell, J. A. et al. Single-cell reconstruction of developmental trajectories during zebrafish embryogenesis. Science 360, doi:10.1126/science.aar3131 (2018).
5 Farnsworth, D. R., Saunders, L. M. & Miller, A. C. A single-cell transcriptome atlas for zebrafish development. Developmental Biology 459, 100-108, doi:https://doi.org/10.1016/j.ydbio.2019.11.008 (2020).
6 Haffter, P. et al. The identification of genes with unique and essential functions in the development of the zebrafish, Danio rerio. Development 123, 1-36, doi:10.1242/dev.123.1.1 (1996).
7 Baranasic, D. et al. Multiomic atlas with functional stratification and developmental dynamics of zebrafish cis-regulatory elements. Nature Genetics 54, 1037-1050, doi:10.1038/s41588-022-01089-w (2022).
8 Yang, H. et al. A map of cis-regulatory elements and 3D genome structures in zebrafish. Nature 588, 337-343, doi:10.1038/s41586-020-2962-9 (2020).
9 Bogdanović, O. et al. Active DNA demethylation at enhancers during the vertebrate phylotypic period. Nat Genet 48, 417-426, doi:10.1038/ng.3522 (2016).
10 Sur, A. et al. Single-cell analysis of shared signatures and transcriptional diversity during zebrafish development. Dev Cell, doi:10.1016/j.devcel.2023.11.001 (2023).
11 Dorrity, M. W. et al. Proteostasis governs differential temperature sensitivity across embryonic cell types. Cell 186, 5015-5027.e5012, doi:10.1016/j.cell.2023.10.013 (2023).
12 Saunders, L. M. et al. Embryo-scale reverse genetics at single-cell resolution. Nature 623, 782-791, doi:10.1038/s41586-023-06720-2 (2023).
13 Jiang, M. et al. Characterization of the Zebrafish Cell Landscape at Single-Cell Resolution. Front Cell Dev Biol 9, 743421, doi:10.3389/fcell.2021.743421 (2021).
14 Qiu, C. et al. Systematic reconstruction of cellular trajectories across mouse embryogenesis. Nature Genetics 54, 328-341, doi:10.1038/s41588-022-01018-x (2022).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


