DNA甲基化跨代遗传研究取得进展
来源:分子植物卓越中心 2020-06-12 12:22
6月8日,Nature Plants 期刊在线发表了中国科学院分子植物科学卓越创新中心朱健康研究组题为Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation 的研究论文。该研究首次发现DNA从头甲基化的改变也
6月8日,Nature Plants 期刊在线发表了中国科学院分子植物科学卓越创新中心朱健康研究组题为Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation 的研究论文。该研究首次发现DNA从头甲基化的改变也能稳定遗传的现象,并阐明了DNA甲基化和组蛋白修饰等多种表观遗传修饰机制共同决定了从头甲基化通路—RdDM依赖的表观等位基因的形成分子机制。
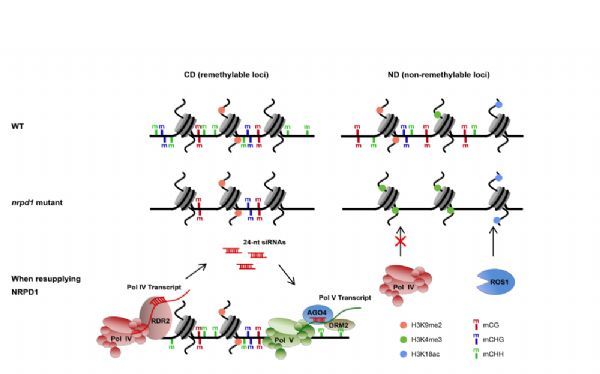
DNA甲基化作为真核生物中最常见的表观遗传修饰之一,其信息能否跨代传递对维持表观基因组稳定性和表观等位基因的形成及传代都是至关重要的。作为植物中的从头甲基化通路,RNA介导的DNA甲基化(RdDM)通路负责所有序列类型(CG, CHG, CHH, H代表A, C, T)的DNA甲基化的从头建立。通常认为该通路靶位点处DNA甲基化的改变是不能稳定遗传的,但全基因组范围内DNA从头甲基化的跨代遗传情况尚不清楚。
RNA聚合酶Pol IV负责产生的24-nt siRNA是RdDM途径的关键一环。该研究通过分析模式植物拟南芥Pol IV最大亚基NRPD1敲除突变体转基因互补株系的DNA甲基化恢复情况,将RdDM靶位点分为两类:甲基化丢失后可以恢复的CD位点和不能恢复的ND位点。生物信息学分析表明CD和ND位点的多种遗传及表观遗传特征是不同的。相较于CD位点,ND位点富含较高水平的活性染色质标记,如组蛋白H3K4me3修饰和H3K18ac修饰;另一方面,不同于ND位点在nrpd1突变体中几乎丢失了所有序列类型的DNA甲基化,CD位点在突变体中依然保留了部分CG以及CHG甲基化。这些特征可能导致了它们在DNA甲基化丢失后恢复过程中的不同表现。
研究人员通过敲除组蛋白H3K4甲基转移酶将部分ND位点转变为CD位点,证明ND位点处较高水平的H3K4me3修饰会阻碍RdDM组分的招募。此外,敲除DNA去甲基化酶ROS1后也会导致部分ND位点转变为CD位点,表明较高水平的H3K18ac修饰可能通过招募DNA去甲基化酶到ND位点进行去甲基化,进而阻碍RdDM通路在此处建立DNA甲基化。研究人员进一步利用CRISPR/dCas9-TET1cd表观基因组定点编辑系统对nrpd1突变体中特定CD位点保留的mCG和mCHG进行定向去甲基化,之后即使恢复NRPD1基因的正常功能,也不能在这些位点从头起始DNA甲基化,证明了CD位点在nrpd1突变体中保留的CG和CHG甲基化可以作为记忆标记招募RdDM组分。
该研究揭示了从头(de novo)DNA甲基化稳定遗传的机制,为深入了解表观等位基因的形成及稳定遗传提供了线索。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。