Nat Mach Intell:识别肿瘤新抗原-物理启发的Sliding-Transformer神经构架
来源:生物探索 2024-10-03 15:39
PISTE算法将物理/生物的先验规则与数据驱动的神经网络框架有机结合,实现了全新的Transformer的注意力机制。
华东理工大学药学院、华东师范大学药学院、人工智能新药创智中心李洪林团队、华东师范大学计算机学院张凯团队,联合复旦大学类脑智能科学与技术研究院张捷团队、上海市第六人民医院胡晓勇团队等多家单位合作在Nature Machine Intelligence发表题为Sliding-attention transformer neural architecture for predicting T cell receptor-antigen-human leucocyte antigen binding 的研究论文。研究人员提出了“物理启发的Sliding-Transformer”算法—PISTE,用于TCR-抗原-HLA结合预测和肿瘤新抗原筛选。该算法在Transformer模型中引入了新颖的物理先验,以残基相互作用的梯度场为指引实现氨基酸残基的逐步定位,从而模拟两个(或更多个)生物序列相互滑动并形成亲和的虚拟过程,为探索TCR/抗原/HLA的复杂相互作用和人类免疫反应的分子基础提供了新的视角和方法。该算法达到了90%以上的精度,提供残基相互作用的清晰图景,并在临床相关的免疫应用方面显示出巨大潜力,为肿瘤新抗原筛选和个性化癌症疫苗研究提供了实用的工具。这项研究不仅体现了人工智能、药学、临床医学与计算科学的交叉融合,也展示了多学科团队合作在推动医学前沿研究中的重要作用。

肿瘤免疫治疗通过刺激和增强机体的抗肿瘤免疫反应来消灭癌细胞。肿瘤新抗原主要是肿瘤细胞发生突变产生的肿瘤特异性抗原,其仅在肿瘤细胞中大量表达,可引发不受中枢和外周耐受性影响的T细胞免疫反应,是免疫系统识别和攻击肿瘤的理想靶点。因此,新抗原的发现和研究对于免疫治疗的发展具有重要意义,为疫苗和免疫疗法的开发提供了重要契机。
肿瘤细胞产生大量的突变肽中,只有一小部分能成功触发抗肿瘤免疫反应。因此准确、快速地识别免疫原性新抗原十分重要。抗原和HLA之间的结合亲和力以及pHLA复合物和相应的TCR之间的“免疫突触”被认为是诱导T细胞反应性的关键决定因素。因此,准确预测TCR-抗原-HLA三元组之间的复杂相互作用是免疫学中的一大计算挑战。由于氨基酸序列的多样性,从大量序列中识别出真正发生亲和的TCR -抗原- HLA三元组非常困难。
其次,即使是最先进的序列模型,如Transformer,其现有的注意力模块仍然无法提供具有明确物理意义的残基相互作用估计,而可解释性是破译免疫反应机制的关键。此外,三元复合物的3D结构数据稀缺,可能产生巨大的实验成本。因此,具有物理可解释性和更高数据效率的预测算法对肿瘤抗原的筛选极为重要。
最后,生物序列呈长尾分布状态,计算模型可能无法准确预测那些具有很少已知TCR配对的肽,或免疫系统中未见过的肽。如何捕获残基相互作用的一般性模式,从而实现有效的迁移学习尚未得到充分的研究。
为了解决上述挑战,作者创新性的提出了“物理启发的的滑动Transformer”算法——Physics Inspired Sliding TransformEr (PISTE),用于TCR -抗原-HLA结合预测问题,解决了当前Transformer注意力机制的局限性,允许在残基水平上有效地探索多个生物序列复杂、高维的相互作用空间。
使用PISTE进行新抗原筛选和排序的流程如图1a所示:首先,获得个体TCR/抗原/HLA三元组序列,并将其输入到PISTE中预测每个三元组的结合评分 (亲和力)。然后使用预测分数来计算每个肽的结合TCR的数量作为免疫原性水平的指标,用于新抗原排序和筛选。
图1b说明了PISTE的网络框架,这是一个具有物理启发的滑动注意机制的Transformer,包括三部分:首先,将抗原/HLA/TCR序列输入序列编码器模块,提取子序列特征;然后使用滑动注意模块估计TCR -抗原- HLA结合。滑动注意力允许残基沿着相互作用的梯度场迭代和协调移动,从而模拟虚拟滑动过程,有效地探索相互作用景观并识别最可能的结合构象。更具体地说,使用滑动注意力来推断抗原如何与HLA结合(第一阶段),并获得HLA-抗原(pHLA)复合物的表示,然后使用滑动注意力来恢复pHLA复合物与TCR之间的相互作用(阶段二),从而建立三元相互作用的全局描述符;最后,将这三个序列反馈到池化模块,得到一个固定维度表示(以HLA伪序列为参考骨架),用于预测全局结合状态。
PISTE在人类免疫过程和生物分子相互作用预测的相关研究中具有几个关键优势:其在预测TCR-抗原- HLA结合方面具有显著的准确率;同时,该方法具有很高的数据效率,即使不使用三维结构训练,亦可准确地重建残基接触图,从而节省了大量的实验成本;此外,提供了不同类型/位置的残基如何相互作用的清晰解释,并能够准确的将预测模型扩展到罕见或新颖的序列,有助于新抗原识别。
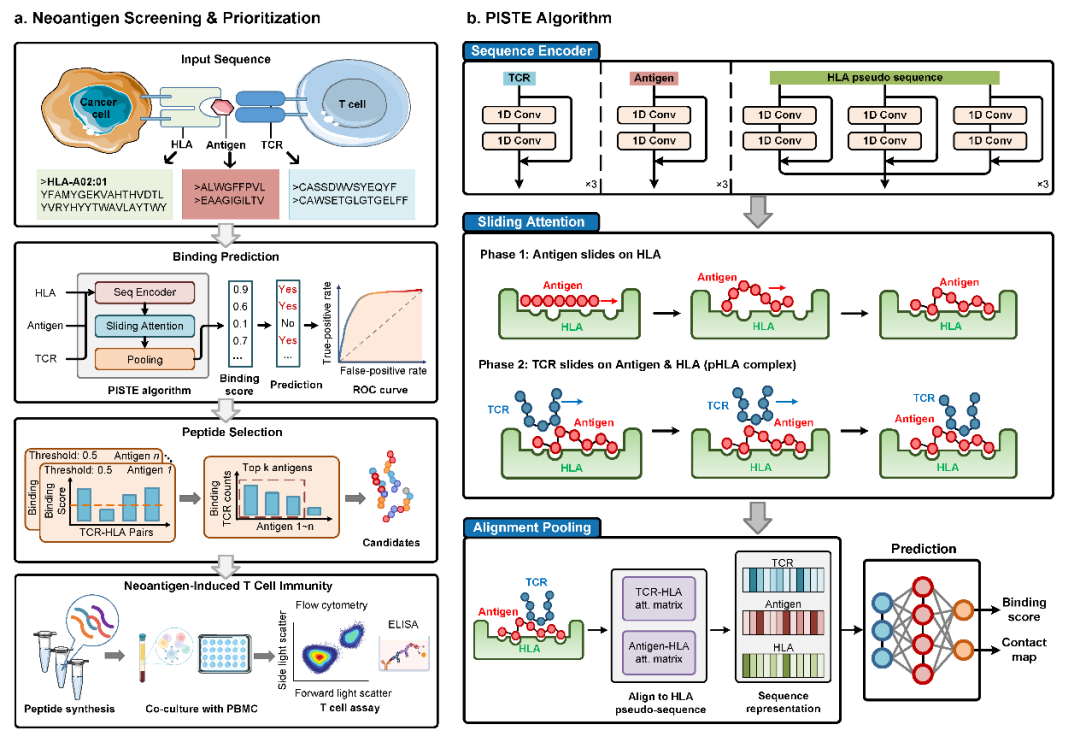
图1. PISTE用于TCR-抗原- HLA结合预测和个性化新抗原筛选。(a)新抗原筛选的流程。(b)PISTE的网络结构,包含序列编码模块,滑动注意力模块和基于比对的池化模块(Credit: Nature Machine Intelligence)
模型预训性能
由于负样本的选择对于评估TCR-抗原-HLA识别模型的泛化性能至关重要,作者分别使用三种采样方法生成负样本,包括:random shuffling、unified peptide和reference TCR负采样。在两个外部测试集上,将PISTE与其他方法进行比较,采用了三种常用的评价指标,包括AUROC,AUPR,PPVn。结果表明,PISTE在三种场景下的均显著高于其他竞争方法。
此外,作者使用PISTE作为工具来恢复和可视化参与不同结合结果的生物序列的分布。通过利用PISTE获得了对于3种特定pHLA复合物的不同TCR的低维表示并采用t-SNE聚类。结果表明PISTE可以捕获结合生物序列的共同属性和亚群的有用表示。
揭示物理上有意义的残基水平相互作用
除了准确预测序列水平的结合状态外,PISTE还可以生成物理上有意义的注意力矩阵,揭示了残基水平的相互作用。作者收集了86个结合的TCR-抗原-HLA三元组及其3D结构,并专门检测了TCR-抗原接触关系和HLA-抗原接触关系。PISTE预测HLA-抗原相互作用矩阵与真实接触矩阵的相关系数为0.75,TCR-抗原相互作用残基关系矩阵的相关系数为0.916,证实了PISTE能够识别TCR、抗原和HLA分子之间复杂的相互作用模式。考虑到PISTE不需要任何3D结构数据进行训练,这是一个强有力的证据,为研究人类免疫反应的分子基础节省了大量的时间和实验成本。
免疫学应用潜力
作者进一步证明了PISTE在几个免疫研究应用中的效用,包括:
检测抗原特异性T细胞克隆。作者使用来自4个健康供体的44个肽- HLA复合物(pHLA)的10X单细胞TCR测序数据,调查了抗原特异性T细胞克隆与结合分数之间的关系。使用PISTE来预测单个T细胞与pHLAs之间的结合亲和力(分数),并记录了最强的分数,结果显示这些结合分数与T细胞克隆率呈正相关,表明对特定抗原表现出优越亲和力的T淋巴细胞对克隆扩增的偏好增加。
TCR-抗原-HLA相互作用影响肿瘤进展和免疫治疗响应。作者通过使用SKCM和GBM队列,探讨了PISTE预测的免疫原性新抗原负荷作为生物标志物的潜力。免疫原性新抗原负荷(INAL)被描述为能够与TCR结合的突变肽的数量,其对应的野生型肽不诱导任何TCR相互作用。结果显示免疫原性新抗原负荷与SKCM和GBM患者的免疫治疗反应和总生存率相关,显著性高于肿瘤突变负荷(TML)和新抗原负荷(NAL)的对照组,表明PISTE预测的免疫原性新抗原负荷具有预后价值。
前列腺癌新抗原的筛选和鉴定。作者结合生物信息学方法分析了8例接受手术治疗的前列腺癌患者的肿瘤全外显子组测序、肿瘤转录组测序和匹配的正常外显子组测序数据,然后利用PISTE预测TCR-新抗原-HLA相互作用,并根据结合结果筛选新抗原。对于每位患者,我们综合个性化排名前3-4位的新抗原,通过细胞水平实验验证了这些新抗原引起的免疫反应。结果显示,75%(6/8)的患者对PISTE预测和筛选的至少一种新抗原引起免疫应答,这表明,PISTE作为预测工具在肿瘤新抗原的识别和筛选中具有显著的有效性和实用性。
总之,PISTE算法将物理/生物的先验规则与数据驱动的神经网络框架有机结合,实现了全新的Transformer的注意力机制:(1)位置编码的动态更新,有效模拟了残基沿其相互作用梯度场迭代移动的虚拟过程;(2)残基相互作用的计算,全面纳入了残基类型、残基位置和相互作用类型,使模型能够以更加接近第一性原理的方式从训练数据中提取信息。PISTE在预测TCR-抗原-HLA结合方面表现出色,是鉴定免疫原性新抗原的重要步骤。同时其为阐明人类免疫反应的分子基础和个性化新抗原筛选/优先排序方面具有极好的通用性,预示着在个性化免疫治疗和癌症疫苗开发领域的巨大潜力。此外,PISTE为分析和预测复杂生物分子相互作用的范例框架。
1. Xie, N., Shen, G., Gao, W. et al. Neoantigens: promising targets for cancer therapy. Sig Transduct Target Ther 8, 9 (2023).
2. Katsikis, P.D., Ishii, K.J. & Schliehe, C. Challenges in developing personalized neoantigen cancer vaccines. Nat Rev Immunol 24, 213–227 (2024).
3. Blass, E., Ott, P.A. Advances in the development of personalized neoantigen-based therapeutic cancer vaccines. Nat Rev Clin Oncol 18, 215–229 (2021).
4. Wells, D. K., van Buuren, M. M., Dang, K. K., Hubbard-Lucey, V. M., Sheehan, K. C., Campbell, K. M., ... & Defranoux, N. A. Key parameters of tumor epitope immunogenicity revealed through a consortium approach improve neoantigen prediction. Cell 183(3), 818-834 (2020).
5. Hudson, D., Fernandes, R.A., Basham, M. et al. Can we predict T cell specificity with digital biology and machine learning?. Nat Rev Immunol 23, 511–521 (2023).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


