Gut Pathog:肠道微生物组或有望作为胰腺癌的“隐形指纹”
来源:生物谷原创 2025-06-20 15:26
本文研究通过分析芬兰和伊朗两个不同人群的粪便样本揭示了胰腺癌患者独特的肠道微生物组特征,这些特征不仅在不同人群中具有一致性,还与胰腺癌的病理生理过程密切相关。
近年来,胰腺癌(PC,Pancreatic Cancer)在全球范围内的发病率和死亡率居高不下,成为医学界亟待攻克的难题。据最新统计,胰腺癌占全球新发癌症病例的1.8%,但其死亡率却高达4.6%,位居癌症相关死亡的第四位。更令人担忧的是,随着生活水平的提高和生活方式的改变,人群胰腺癌的发病率在低收入和中等收入国家呈上升趋势,预计未来40年内其死亡率将会翻倍。目前,胰腺癌的诊断主要依赖于影像学检查和血清标志物检测,但这些方法往往只能在疾病晚期才显示出异常,从而就会导致患者错过最佳治疗时机,因此,开发一种早期、无创的胰腺癌筛查方法已成为当务之急。
由于不同人群的肠道微生物组成存在显著差异,目前对于胰腺癌相关肠道微生物特征的研究结果尚不一致。日前,一篇发表在国际杂志Gut Pathogens上题为“Fecal profiling reveals a common microbial signature for pancreatic cancer in Finnish and Iranian cohorts”的研究报告中,来自芬兰赫尔辛基大学等机构的科学家们通过分析芬兰和伊朗两个不同人群的粪便样本,揭示了胰腺癌患者机体的肠道微生物组特征,并评估其在疾病早期诊断中的潜在应用价值。通过分析不同人群的肠道微生物组,研究人员希望能揭示胰腺癌的共同微生物标志物,并为开发无创诊断工具提供一定的科学依据。
这项研究中,研究人员纳入了33名芬兰胰腺癌患者和50名伊朗胰腺癌患者,以及35名芬兰健康对照组和34名伊朗健康对照组,所有患者均被诊断为胰腺导管腺癌(PDAC,胰腺癌最常见的类型)。研究者采用16S rRNA基因扩增子测序技术对粪便样本进行分析,他们通过自动化DNA提取仪对采集的样本进行DNA提取,并使用Ion Torrent PGM™系统进行单向测序,同时还利用QIAGEN CLC Genomics Workbench软件进行微生物群落组成和多样性的分析。
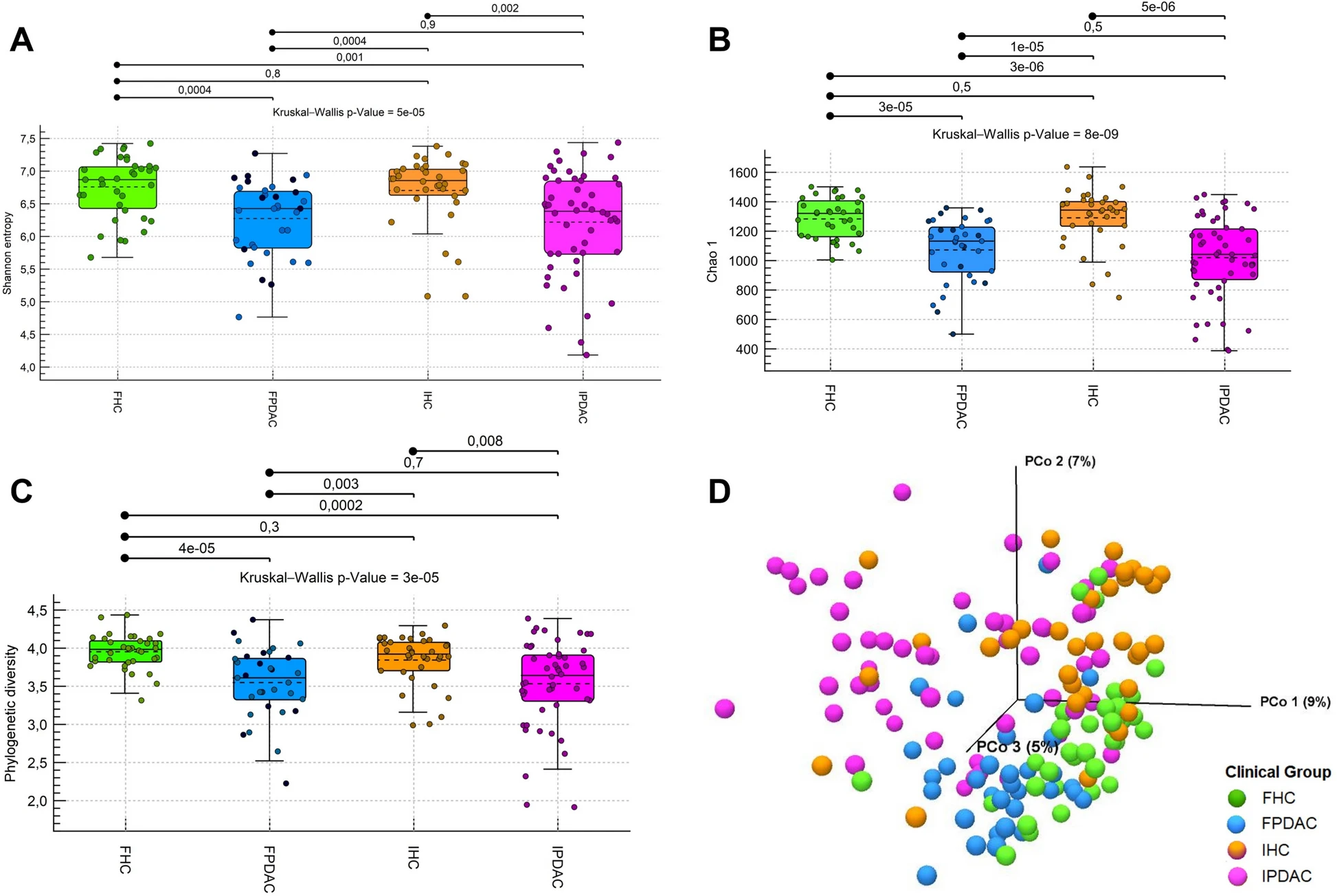
芬兰和伊朗PDAC患者和健康对照者的肠道微生物群多样性
首先,研究人员对样本的微生物群落多样性进行评估,包括α多样性(物种丰富度和均匀度)和β多样性(群落组成差异),随后他们通过广义线性模型(GLM)进行差异丰度分析筛选出与胰腺癌显著相关的细菌分类单元,并利用机器学习算法(如逻辑回归和支持向量机)来构建胰腺癌的预测模型,同时在芬兰队列中进行验证。
研究结果表明,胰腺癌患者的肠道微生物组能表现出显著的多样性降低,且与健康对照组相比,其肠道微生物组成存在显著差异。具体而言,胰腺癌患者的肠道中富集了多种潜在致病菌,如肠杆菌科、肠球菌科和梭杆菌科,而有益菌(如丁酸产生菌)则显著减少,此外,研究人员通过机器学习算法构建的预测模型在伊朗队列中表现出较高的预测性能(AUC值为0.88),并在芬兰队列中得到了良好的验证,这就表明,肠道微生物组具有作为胰腺癌早期诊断标志物的潜力。
综上,本文研究通过分析芬兰和伊朗两个不同人群的粪便样本揭示了胰腺癌患者独特的肠道微生物组特征,这些特征不仅在不同人群中具有一致性,还与胰腺癌的病理生理过程密切相关。研究者认为,肠道微生物组的变化可能会通过影响宿主的免疫反应和炎症状态,从而促进胰腺癌的发生和发展。未来,研究人员还需要在更大规模、更多样化的队列中进一步验证这些发现,并探索肠道微生物组与胰腺癌之间的因果关系。(生物谷Bioon.com)
参考文献:
Sammallahti, H., Rezasoltani, S., Pekkala, S. et al. Fecal profiling reveals a common microbial signature for pancreatic cancer in Finnish and Iranian cohorts. Gut Pathog 17, 24 (2025).doi:10.1186/s13099-025-00698-0
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


