Cell:从人体微生物组中挖掘全新抗菌肽,有望解决抗生素耐药性难题
来源:生物世界 2024-08-21 09:37
该研究利用计算和实验筛选平台,挖掘了一个最近扩展的微生物组微型蛋白数据库,以寻找具有抗菌活性的候选抗菌肽,并鉴定了高效和特异的抗菌肽序列。
宾夕法尼亚大学和斯坦福大学的研究人员合作,在国际顶尖学术期刊 Cell 上发表了题为:Mining human microbiomes reveals an untapped source of peptide antibiotics 的研究论文。
该研究通过计算筛选,从人体微生物组(包括皮肤微生物、口腔微生物、肠道微生物)中挖掘候选抗菌肽,共鉴定出323个由小开放阅读框编码的潜在抗菌肽,其中合成的78个候选抗菌肽中有55个(70.5%)在体外具有抗菌活性,来自肠道细菌普雷沃氏菌Prevotella copri编码的prevotellin-2具有与常用抗生素多粘菌素B相当的抗菌活性。

尽管长期以来注释编码长度≤50个氨基酸残基的蛋白质的基因因计算挑战而被忽视,但最近发现,人体微生物组可编码数十万个小开放阅读框(small open ORF)。其中,只有很小一部分被功能性地表征出来。这些具有潜在抗菌活性的未探索肽序列代表了巨大的未开发来源。
事实上,细菌已经被证明可产生抗菌分子以在复杂的生态系统中竞争。这些分子通过多种不同的机制发挥作用,使细菌能够杀死相关和不相关的菌株,促进对有限的生态位的竞争。
最近有研究利用自然语言处理神经网络模型来探索发现抗菌肽的潜力,这种方法已经成功地从粪便宏蛋白质组数据集中鉴定出了几个有效的抗菌肽,但它可能忽略了许多表达量较少或来自非粪便微生物组来源的肽,例如皮肤和口腔微生物组中已知含有大量抗菌肽基因。
在这项最新研究中,研究团队试图采取一种不受限的方法,考虑来自人体多个部位的人类宏基因组的小肽。研究团队从之前在人类微生物组计划(Human Microbiome Project,HMP)宏基因组中注释的444054个预测小肽的数据集开始。为了将这一广泛的候选小开放阅读框列表缩小到可用于抗菌活性测试的候选小开放阅读框,研究团队使用现有的计算算法预测给定肽序列具有抗菌活性的可能性,最终产生了一个列表,包含323个候选小开放阅读框编码的抗菌肽。
然后,研究团队化学合成了其中的78个抗菌肽,并在体外对高优先级病原体和常见肠道共生菌进行了测试,发现了其中55个具有抗菌活性(70.5%)。其中5个不同来源(皮肤、口腔、肠道)的主要候选抗菌肽对病原体具有高度抗菌活性,而对共生菌的活性有限或无活性。这5个抗菌肽分别是来自Faecalibacterium prausnitzii的faecalibacticin-3、来自Fusobacterium nucleatum的fusobacticin-2、来自Keratinibaculum paraultunense的keratinobacin-1、来自Staphylococcus capitis的staphylococcin-2,以及来自Prevotella copri的prevotellin-2。
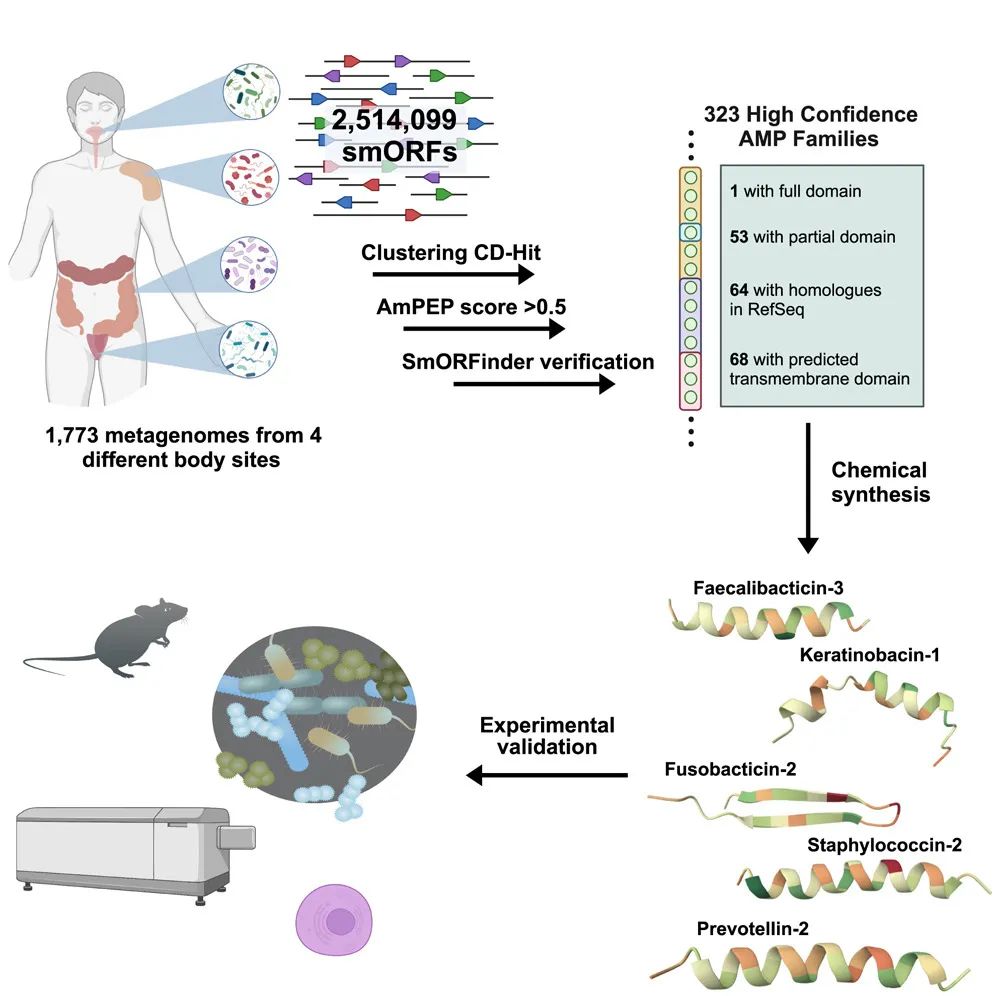
研究团队还进行了体内测试,在临床前感染动物模型中显示出抗菌活性,例如,人类肠道细菌普雷沃氏菌Prevotella copri产生的prevotellin-2具有与常用抗生素多粘菌素B相当的抗菌活性。
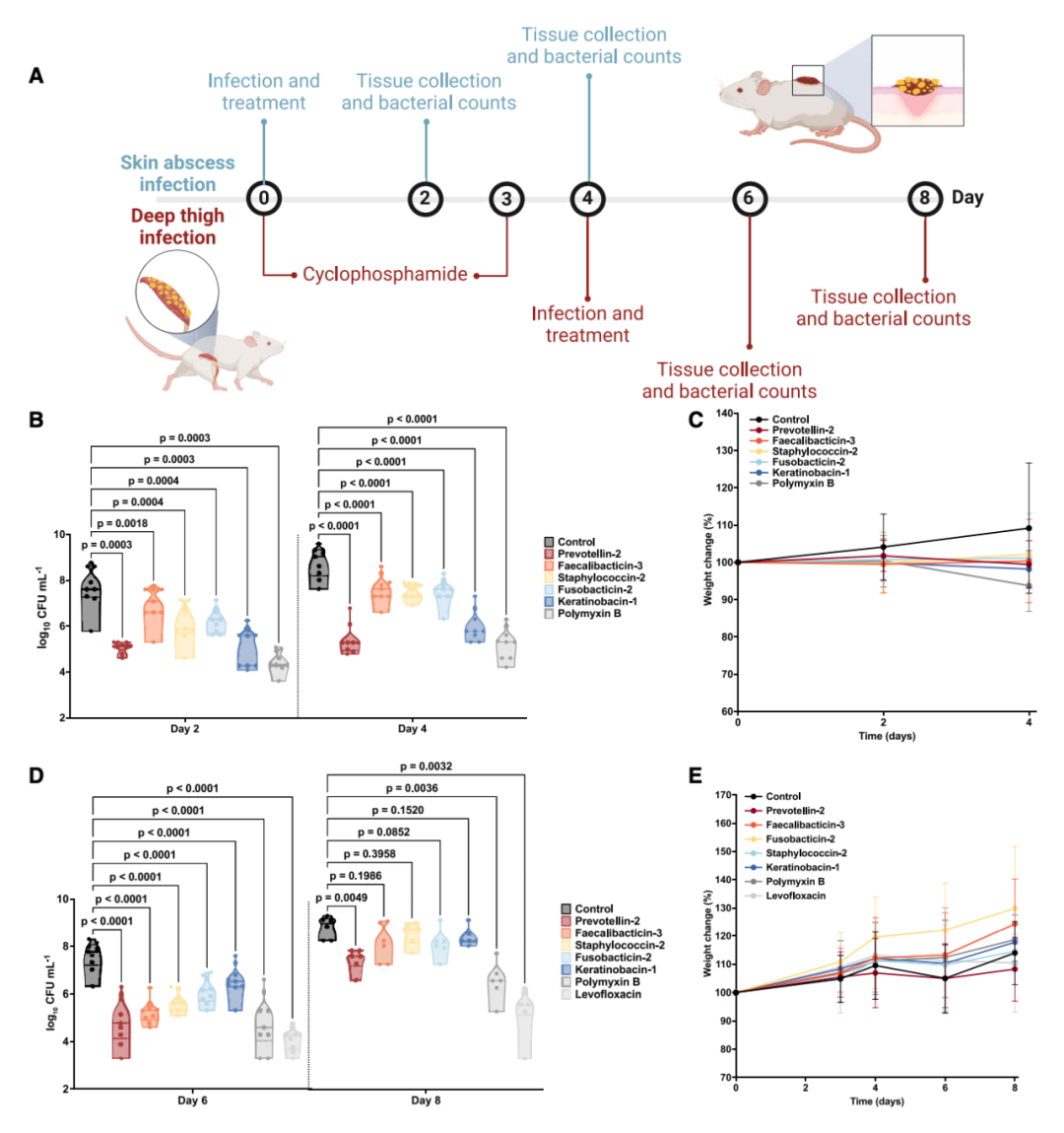
综上所述,该研究利用计算和实验筛选平台,挖掘了一个最近扩展的微生物组微型蛋白数据库,以寻找具有抗菌活性的候选抗菌肽,并鉴定了高效和特异的抗菌肽序列,支持了人类微生物组中存在成百上千种可用于临床转化的抗菌肽。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


