Nature子刊:将Transformer模型应用于蛋白翻译过程
来源:生物世界 2024-03-10 12:25
Riboformer提供了解析核糖体分布变化的有力工具,展示了面向上下文的深度学习模型捕捉生物过程复杂动态的潜力。
该研究开发了一个名为Riboformer的深度学习框架,为我们理解翻译动态过程提供了全新的方法。这项研究利用Transformer架构对不同实验组中核糖体密度在密码子分辨率上的变化进行比对和精确建模,提供了一个可以矫正数据偏差的标准化计算框架。该架构也具有可解释性,作者将其应用于衰老和病毒感染过程,发现了导致核糖体延伸停滞的肽段序列,揭示了疾病过程中翻译动力学的调控基础。
该研究首先将Riboformer框架应用于原核生物Ribosome profiling数据集的偏差矫正和标准化。在原核生物中,人们往往需要在裂解缓冲液中加入氯霉素以阻止翻译起始和延伸。但氯霉素的效果并不完美,反而会导致核糖体在特定序列上的暂停。作者利用Riboformer框架矫正了来自四个实验室的16组数据中的实验偏差,验证了该计算框架的稳健表现。通过对矫正后数据的分析,该研究揭示了同义密码子翻译速率的微妙差别,并发现了细菌在表达外源蛋白情况下翻译延长的潜在瓶颈。
除了数据标准化以外,Riboformer还可以模拟mRNA序列突变对于核糖体分布的影响,从而找到能够导致核糖体翻译停滞的特定序列,对核糖体停滞位点进行更为精细的研究。作者将该方法应用于酵母和线虫衰老数据集中,通过对年轻和年老数据进行比对,揭示了mRNA结构,脯氨酸序列和氨基酸所带电荷等因素对于翻译延伸速率的影响,而原有的基于所有核糖体停滞位点的统计分析则很难得到类似结果。
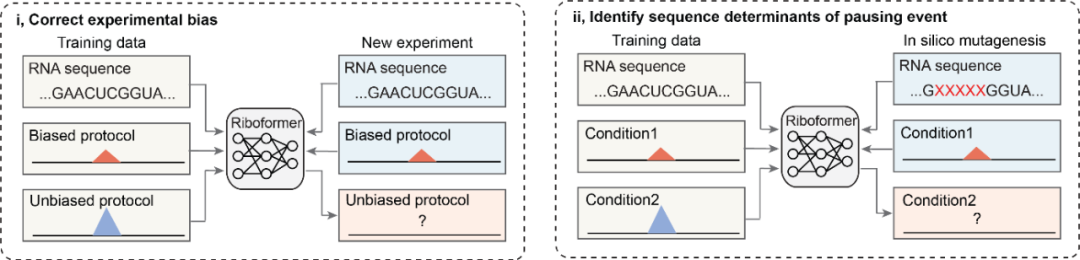
综上,Riboformer提供了解析核糖体分布变化的有力工具,展示了面向上下文的深度学习模型捕捉生物过程复杂动态的潜力。Riboformer不仅能够改进已有实验数据的分析,还能预测已有数据集在新的实验条件下的核糖体延伸速率。随着人们对翻译过程高通量定量测量水平的不断进步和新的训练数据的不断涌现,该工作有望为解决翻译调控与蛋白质稳态失衡相关的疾病提供新的视角和治疗策略。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


