盘点:2022年CRISPR/Cas研究进展
来源:生物谷原创 2022-12-27 13:37
CRISPR/Cas9是继“锌指核酸内切酶(ZFN)”、“转录激活子样效应因子核酸酶(TALEN)”之后出现的第三代“基因组定点编辑技术&r
CRISPR/Cas9是继“锌指核酸内切酶(ZFN)”、“转录激活子样效应因子核酸酶(TALEN)”之后出现的第三代“基因组定点编辑技术”。与前两代技术相比,其成本低、制作简便、快捷高效的优点,让它迅速风靡于世界各地的实验室,成为科研、医疗等领域的有效工具,而且经过不断改进后,更被认为能够在活细胞中最有效、最便捷地“编辑”任何基因。
基因组编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一,受到人们的高度重视。CRISPR是规律间隔性成簇短回文重复序列的简称,Cas是CRISPR相关蛋白的简称。CRISPR/Cas9是由一种原始的细菌免疫系统改编而成的,它的作用方式是首先在基因组的一个靶位点上切割双链DNA。
在CRISPR/Cas9系统中,酶Cas9在DNA靶位点上进行切割,其中这种靶位点是这样确定的:一种被称作CRISPR RNA(crRNA)的RNA分子利用它的一部分序列与另一种被称作tracrRNA的RNA分子通过碱基配对结合在一起,形成嵌合RNA(tracrRNA/crRNA),然后,借助crRNA的另一部分序列与靶DNA位点进行碱基配对,以这种方式,这种嵌合RNA就能够引导Cas9结合到这个靶位点上并进行切割。在实际应用时,人们可以将tracrRNA和crRNA作为两种向导RNA(gRNA)或者融合在一起形成单向导RNA(single guide RNA, sgRNA),并被用来引导酶Cas9结合到靶DNA序列上并进行切割,其中Cas9与sgRNA一起被称作Cas9-sgRNA系统。
此外,CRISPR/Cas9系统靶向识别和切割与前间隔序列邻近基序(protospacer adjacent motif, PAM)相邻的特定DNA位点。作为一种最为频繁用于基因组编辑的Cas9酶,来自酿脓链球菌(Streptococcus pyogenes)的Cas9(SpCas9)仅识别作为PAM的NGG序列(简称NGG PAM,其中N代表任何一种碱基),这就限制了基因组中能够被靶向的区域。
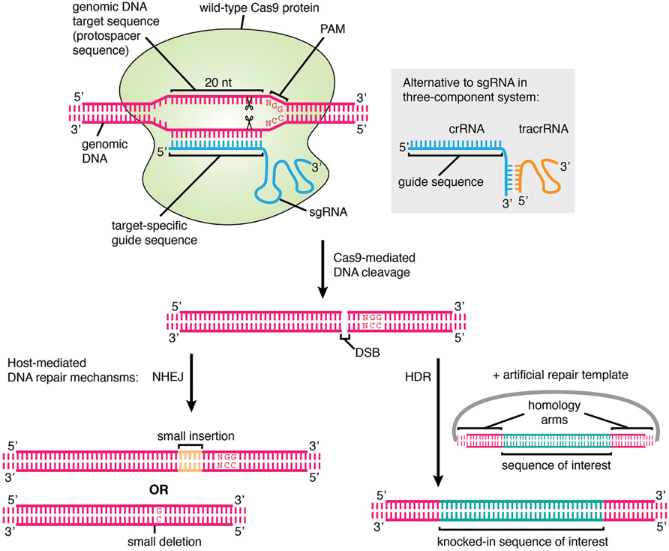
CRISPR/Cas9作用机制. 图片来自Frontiers in Genetics, 24 September 2015, doi:10.3389/fgene.2015.00300。
与CRISPR/Cas9相比,碱基编辑并不切割DNA双螺旋,而是在组成DNA或RNA的四个碱基中,利用酶精确地重新排列其中的一个碱基上的一些原子,从而将这个碱基转化为一个不同的碱基,同时不改变其周围的碱基。这种能力大大增加了改变遗传物质的选择手段。2017年,通过碱基编辑器编辑技单个碱基技术入选2017年《科学》杂志“科学十大突破”。
在2022年,科学家们在CRISPR/Cas取得重大的进展,让我们一起看看这个领域在这一年里取得的重大发现。
1.两篇Science揭示揭示RNA引导的CRISPR-Cas效应蛋白切割可触发适应性免疫反应
在第一项新的研究中,来自美国麻省理工学院的研究人员证明了CRISPR相关蛋白酶Csx29对σ因子抑制剂表现出可编程的RNA激活的内肽酶活性,以调节转录反应。活性的与底物结合的Csx29复合物的低温电镜结构显示了一种异构激活机制,该机制在靶RNA结合时重组Csx29的催化残基(Science, 2022, doi:10.1126/science.add7450)。这一发现揭示了自然界中的一种RNA引导功能,可用于体外和人类细胞中的RNA检测应用。
在第二项新的研究中,来自美国麻省理工学院和日本东京大学的研究人员发现III-E型CRISPR系统包括类似于半胱天冬蛋白酶(caspase)的蛋白酶Csx29,当Cas7-11识别靶RNA时,Csx29就被激活从而切割另一种称为Csx30的辅助蛋白。这种切割产生有毒的Csx30片段,据推测这些片段能抑制专门的σ因子RpoE,调节细菌对噬菌体感染的反应,并导致细菌生长停滞,从而清除噬菌体感染(Science, 2022, doi:10.1126/science.add7347)。
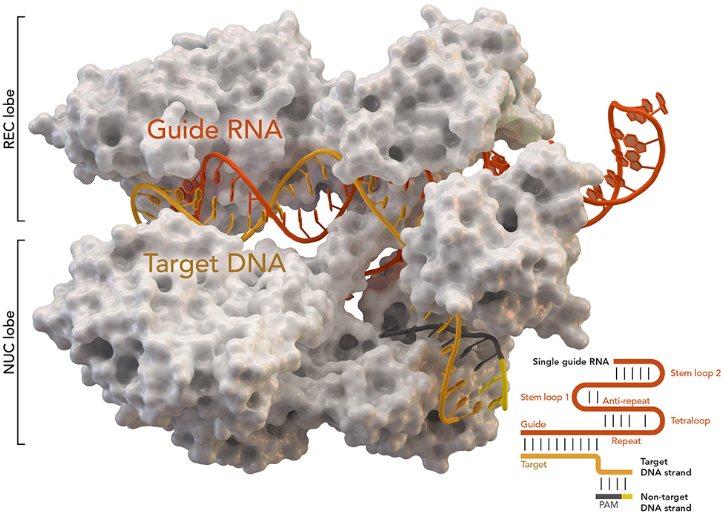
CRISPR/Cas9基因组编辑系统,图片来自Thomas Splettstoesser (Wikipedia, CC BY-SA 4.0)。
这些发现扩大了CRISPR免疫反应的已知复杂性,使得对哺乳动物细胞中基于蛋白酶的可编程RNA检测成为可能。总体来说,Cas7-11-Csx29效应蛋白是一种RNA依赖性的核酸酶-蛋白酶。
2.Cell:在数千种病毒中发现CRISPR-Cas系统,有望改善细胞基因组编辑
对病毒基因组的系统性扫描揭示了大量潜在的基于CRISPR的基因组编辑工具。CRISPR-Cas系统在细菌和古生菌的微生物世界中很常见,它们经常帮助它们的宿主细胞抵御病毒。但是,在一项新的研究中,美国加州大学伯克利分校的分子生物学家Jennifer Doudna和微生物学家Jillian Banfield及其同事们决定在感染细菌和古生菌的病毒(即噬菌体)中更全面地寻找CRISPR-Cas系统。令人吃惊的是,他们发现了大约6000种编码CRISPR-Cas系统的噬菌体,包括每一种已知类型的CRISPR-Cas系统的代表。他们认为,这些病毒利用CRISPR-Cas彼此竞争---而且有可能也是为了操纵宿主的基因活性,使之对自己有利。
图片来自Cell, 2022, doi:10.1016/j.cell.2022.10.020。
他们发现了在通常的CRISPR-Cas结构上存在着广泛的变异,一些CRISPR-Cas系统缺少组分,另一些CRISPR-Cas则异常紧凑。在法国国家科学研究中心研究噬菌体生态学和进化的Anne Chevallereau说,“即使噬菌体编码的CRISPR-Cas系统是罕见的,它们也是高度多样化和广泛分布的。大自然充满了惊喜。”
3.Science子刊:1期临床试验表明经过CRISPR基因编辑的通用CAR-T细胞可用于治疗复发性B细胞白血病
在一项新的临床研究中,来自英国大奥蒙德街儿童医院和伦敦大学学院的研究人员利用CRISPR/Cas9技术对供者T细胞进行基因改造,试图治疗患有耐药性白血病的重症儿童,这些儿童已用尽所有可用的治疗方法。这项I期临床试验是首次在人类身上使用“通用的”经过CRISPR基因编辑的T细胞,代表着在使用基因编辑细胞治疗癌症方面迈出了重要一步。作为这项临床试验的一部分,他们构建并应用了新一代的更精确的“通用”基因组编辑T细胞(Science Translational Medicine, 2022, doi:10.1126/scitranslmed.abq3010)。
这些作者使用CRISPR对T细胞进行了基因修饰,具体而言它对T细胞的DNA进行切割并插入了一段遗传代码。在这种情况下,这段遗传代码允许T细胞表达一种识别癌变B细胞表面上的一种称为CD19的标志物的嵌合抗原受体(CAR),所产生的CD19 CAR-T细胞摧毁这些癌变B细胞。他们随后利用CRISPR破坏了CD19 CAR-T细胞中的T细胞受体α链并移除它们的CD52,由此获得TT52CAR19 T细胞,这样构建出一种通用的可以“现成”使用的CAR-T细胞疗法,而无需任何供者匹配。
虽然英国国家医疗服务体系如今提供了一些CAR-T细胞疗法,但它们依赖于收集患者自身的T细胞并对它们进行基因改造。这很昂贵,并不总是可行的,也不可能在短时间内实现。目前正在研究基因组编辑,以允许供者提供的T细胞经过预先制造后用于多名患者,目的是降低成本,使得它们更容易获得。
4.Nat Biotechnol:利用改进的CRISPR-Cas9基因编辑系统将较长的DNA序列高效引入细胞基因组中的精确位点
DNA可以以单链或双链形式存在,而Cas9会附着在双链DNA上。高水平的双链DNA模板对细胞有毒,因此该方法只能用于低量的模板DNA,这会导致编辑效率低下。单链DNA对细胞的毒性较小,即使在相对高的浓度下也是如此。与传统的双链DNA模板相比,单链DNA模板可以使基因编辑的效率提高一倍以上。
在一项新的研究中,来自美国格拉斯通研究所和加州大学旧金山分校的研究人员描述了一种将改良的Cas9酶附着在单链DNA模板上的方法,只需在两端添加一小段悬空的双链DNA,从而能够以非常高的效率将特别长的DNA序列引入细胞基因组的精确位置,而不需要传统上用来携带DNA进入细胞的病毒递送系统。单链DNA分子的双链末端让人们可以使用Cas9来加强非病毒载体在细胞中的递送(Nature Biotechnology, 2022, doi:10.1038/s41587-022-01418-8)。
此外,他们使用新的DNA模板产生了超过10亿个靶向多发性骨髓瘤的CAR-T细胞。CAR-T细胞是经过基因改造的T细胞,可以有效对抗特定的细胞或癌症。有了新的由Cas9指导的单链模板,大约一半的T细胞获得了新的基因,因而被转化为CAR-T细胞。
论文共同作者、加州大学旧金山分校血液学与肿瘤学科医学助理教授Justin Eyquem博士说,“我们知道,将DNA模板靶向基因组中称为TRAC(T-cell receptor α constant, T细胞受体α恒定区)位点的特定位点,将会提高CAR-T细胞的抗肿瘤效力。这种新的非病毒方法使我们能够更有效地实现这一目标,这将加快下一代CAR-T细胞疗法的开发。”
5.Science:重大进展!揭示CRISPR RNA引导的蛋白酶的作用机制,有望提供新的抗病毒工具和组织工程工具
在一项新的研究中,来自美国康乃尔大学、荷兰代尔夫特理工大学和韩国浦项科技大学的研究人员为一系列CRISPR系统提供了新的见解,这可能导致在动物和植物中有前途的抗病毒工具和组织工程工具。他们着重关注新发现的CRISPR RNA引导的Caspase(CRISPR RNA-guided Caspase, Craspase)系统(Science, 2022, doi:10.1126/science.add5064)。论文通讯作者为康乃尔大学文理学院分子生物学与遗传学教授Ailong Ke博士和代尔夫特理工大学的Stan J.J. Brouns博士。
图片来自Science, 2022, doi:10.1126/science.add5064。
CRISPR-Cas系统是细菌中RNA引导的核酸酶,它在精确的位置切割病毒DNA或RNA靶标,以实现强大的基因组编辑应用。半胱氨酸天冬氨酸蛋白酶(Caspase)是一种蛋白酶家族,控制包括人类在内的动物体内的程序性细胞死亡。最近的一个发现是,类似于Caspase的蛋白可以与CRISPR-Cas关联在一起,这使科学界为之振奋。这种CRISPR引导的caspase被赋予了一个新的名字,即Craspase。
在这篇论文中,这些作者使用针对Craspase系统的低温电镜图来解释它们如何切割靶RNA并激活可以分解蛋白的蛋白酶。Ke说,“这些低温电镜图导致了一部高清晰度的分子电影。通过来回观看,我们准确地知道Craspase是如何识别RNA靶标的,这又是如何激活蛋白酶的,这种活性持续多长时间,以及什么最终关闭了这种蛋白酶的活性。关于如何从这个平台汲取力量的想法开始涌入。”
6.Nat Commun:当心!CRISPR/Cas9基因编辑可能导致细胞毒性和基因组不稳定性
CRISPR/Cas9,俗称“基因剪刀”,是一种精确的基因编辑技术。它允许将所需的DNA序列引入到基因组的(几乎)任何位置,从而修改或灭活一个基因。这种技术被广泛用于生物医学研究,而且一些基于CRISPR/Cas9的疗法正在进行临床试验,用于治疗人类血液疾病、某些类型的癌症和HIV感染以及其他疾病。
在一项新的研究中,西班牙巴塞罗那生物医药研究所研究员Fran Supek博士及其研究团队报告说,根据人类基因组的靶序列位点,CRISPR/Cas9基因编辑可以引起细胞毒性和基因组不稳定性。这种不想要的影响是由关键的肿瘤抑制蛋白p53介导的,并由编辑位点附近的DNA序列和周围区域的多种表观遗传因子决定(Nature Communications, 2022, doi:10.1038/s41467-022-32285-1)。
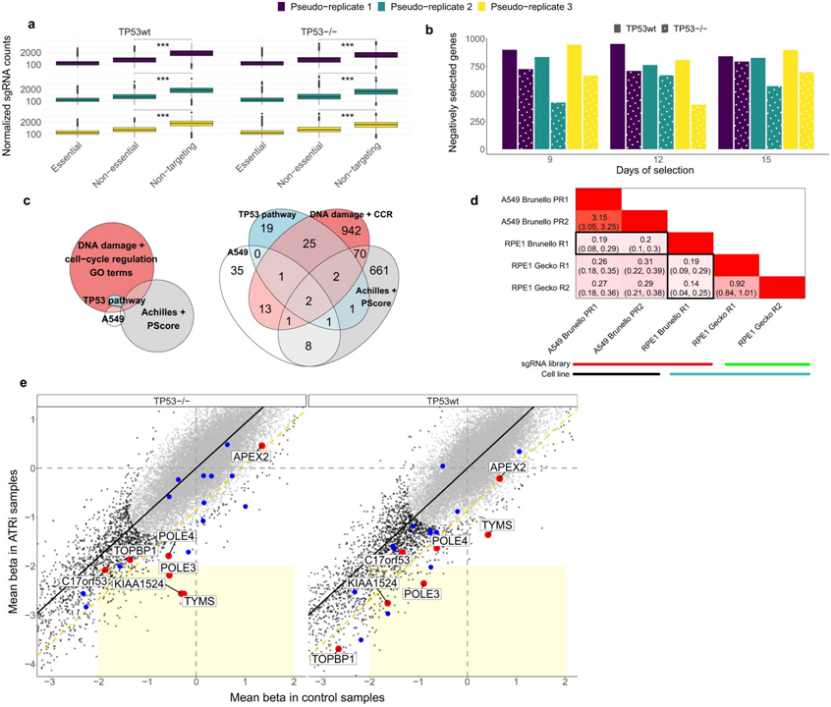
TP53野生型背景可能会混淆基因筛选中基因选择的估计,图片来自Nature Communications, 2022, doi:10.1038/s41467-022-32285-1。
利用计算方法,这些作者分析了为人类细胞设计的最受欢迎的CRISPR文库,并检测到了3300个显示出强烈毒副作用的靶序列位点。他们还报告说,大约15%的人类基因含有至少一个显示出毒副作用的编辑位点。
Supek博士解释说,“我们的研究解决了与TP53相关的Cas9毒副作用的一个重要问题---也是最近有一些争议的问题,它也提供了如何回避这个问题的指南。避免在这些‘风险’位点上进行编辑,不仅会使CRISPR编辑更有效,更重要的是更安全。”
7.Nature:利用CRISPR添加反转录子条形码记录基因在DNA中的表达顺序
在一项新的研究中,来自美国格拉德斯通研究所的研究人员开发出一种更像日记或收据簿的工具---它可以一次记录细胞的基因活动数天。这种被命名为Retro-Cascorder的工具在DNA链内记录数据,然后可以在任何时候对其进行分析以获得细胞的活动记录(Nature, 2022, doi:10.1038/s41586-022-04994-6)。
对于构建Retro-Cascorder的第一步,Shipman团队转向了反转录子(retron),即在激活时产生特定DNA序列的细菌序列。这些作者给感兴趣的基因添加了一个反转录子。每次该基因被激活时,这个反转录子也会产生一个相应的携带该基因特有条形码的DNA序列。接下来,这些作者希望有一个分子分类账簿,按时间顺序记录这些收据。为此,他们使用了CRISPR阵列,即一个较长的DNA重复序列,细菌通常在CRISPR阵列中复制它们在免疫记忆中所需要的DNA序列---按照它们接收这些信息的顺序。
通过将CRISPR阵列和反转录子整合到相同的细胞中,Shipman团队确保反转录子产生的每一个DNA收据都会插入到CRISPR阵列中。为了检索CRISPR阵列中包含的信息,他们只需对细胞的基因组进行测序,并查看CRISPR阵列中的反转录子序列的顺序。
8.Science子刊:利用CRISPR的改进版本揭示促进肺气肿和慢性阻塞性肺病产生的基因
在一项新的研究中,来自美国波士顿大学和波士顿医学中心的研究人员利用CRISPR的改进版本---CRISPR激活(CRISPRa)和CRISPR干扰(CRISPRi)---了解促进肺气肿和慢性阻塞性肺病(COPD)产生的基因的功能。他们通过关闭促进这些疾病发病机制的基因的表达,发现了它们的功能性结果(Science Advances, 2022, doi:10.1126/sciadv.abo6566)。
这些作者利用CRISPR的改进版本设计了一个系统,在iPS细胞中用CRISPRi关闭一个感兴趣的基因的表达,或用CRISPR激活(CRISPRa)过度表达一个感兴趣的基因。他们在培养皿中培养这些细胞,并使它们分化为驻留在肺部中的细胞。所研究的肺部驻留细胞类型被称为2型肺泡上皮细胞(type 2 alveolar epithelial cell, AT2),是肺泡中的祖细胞---肺泡是肺部发生气体交换的部分,是肺气肿中受损的结构。因此,通过了解全基因组关联研究中发现的基因如何影响AT2细胞,他们可以开始了解这些基因可能对影响这些细胞的疾病(如肺气肿)有什么贡献。

利用CRISPRi平台研究COPD GWAS基因概述,图片来自Science Advances, 2022, doi:10.1126/sciadv.abo6566。
一旦产生AT2细胞,这些作者就使用CRISPRi关闭9个不同的在全基因组关联研究中发现的基因的表达,并对其进行分析,以了解这些细胞如何受到影响,尤其是它们的增殖能力,这是它们在应对肺气肿等损伤时需要做到的。他们注意到,关闭一个特定的基因,即编码桥粒斑蛋白(desmoplakin, DSP)的基因,会导致这些细胞增加增殖,并增加与细胞成熟有关的基因的表达。他们发现,在烟雾暴露前关闭DSP表达的AT2细胞比对照细胞在更大程度上关闭了细胞连接(cell junction)基因的表达。这些细胞在形成新集落方面也比对照细胞更好,这是衡量祖细胞功能的一个指标。他们随后观察了从肺上皮细胞中剔除了DSP的小鼠,与具有正常DSP的对照组小鼠进行比较。他们发现,DSP缺失小鼠的AT2细胞在受伤后更容易增殖,这与针对人类iPS细胞衍生的AT2细胞获得的发现一致。
9.Nat Biotechnol:警惕!基于CRISPR技术的临床疗法或会损伤机体细胞的基因组!
异体T细胞疗法的多项临床试验能使用位点特异性的核酸酶类干扰T细胞受体(TCR)和其它基因的表达。在一项新的研究中,来自特拉维夫大学等机构的科学家们表示,CRISPR疗法或许会损伤机体的基因组,CRISPR基因组编辑手段非常有效,但并不总是安全,有时所分裂的染色体并不会恢复功能,而且基因组的稳定性也会受到影响,从长远来看,这或许会导致癌症的发生(Nature Biotechnology, 2022, doi:10.1038/s41587-022-01377-0)。
文章中,研究人员指出了使用CRISPR疗法所产生的风险,目前CRISPR疗法被广泛用于治疗诸如癌症、肝脏和肠道疾病、遗传综合征等多种疾病中,研究人员在调查这种技术对免疫系统白细胞的影响时,发现了在高达10%的所治疗的细胞中都会出现相当大比例的细胞遗传物质的丢失,这种损失或许会导致细胞基因组的不稳定,从而诱发癌症。
10.Cell:基于CRISPR技术的新型图谱将每个人类基因与其功能关联在一起
人类基因组计划(Human Genome Project)是一项雄心勃勃的举措,旨在对人类的每个DNA片段进行测序。该项目吸引了来自世界各地研究机构(包括包括怀特黑德研究所)的合作者,并最终于2003年完成。如今,二十多年过去了,怀特黑德研究所成员Jonathan Weissman及其同事们在一项新的研究中超越了人类基因组序列,首次全面地构建出人类细胞中表达的基因的功能图谱。他们将每个基因与它在细胞中的作用联系起来,这是多年来在单细胞测序方法Perturb-seq上合作的成果(Cell, 2022, doi:10.1016/j.cell.2022.05.013)。
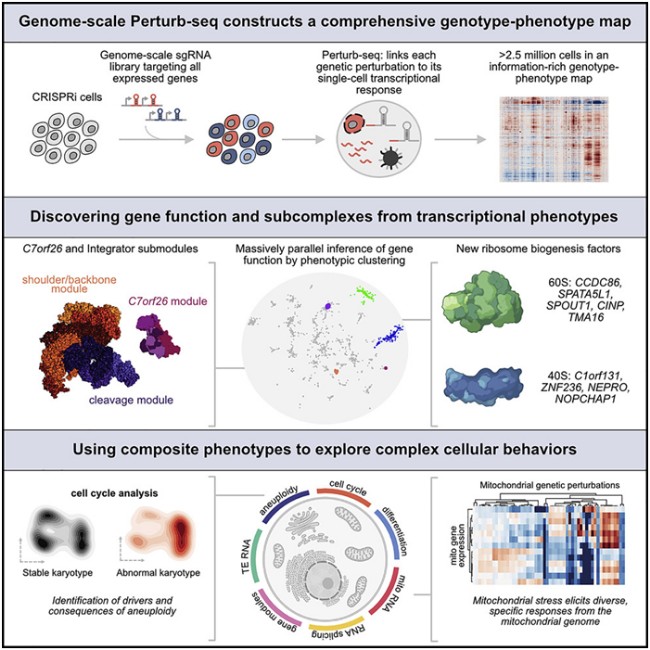
图片来自Cell, 2022, doi:10.1016/j.cell.2022.05.013。
这些数据可以在Weissman实验室的网站上找到,供其他科学家使用。Weissman说,“这是一个大资源,就像人类基因组是一个大资源一样,你可以进去并进行基于发现的研究。与其提前确定你要研究的生物学,不如拥有这张基因型-表型关系图,你可以进去筛选这个数据库,而不必做任何实验。”
这种筛选使得这些作者能够深入研究各种生物学问题。他们用它来探索功能未知的基因对细胞的影响,研究线粒体对应激的反应,并筛选出导致染色体丢失或增加的基因,一种在过去被证明很难研究的表型。论文共同通讯作者、前Weissman实验室博士后Tom Norman说,“我认为这个数据集将使那些来自生物学其他领域的人能够进行各种我们甚至还没有想到的分析,突然之间他们就有了可以利用的数据。
11. Cell:更为紧凑的靶向RNA的基因编辑工具Cas7-11S问世,有望基于此开发出新型RNA基因疗法
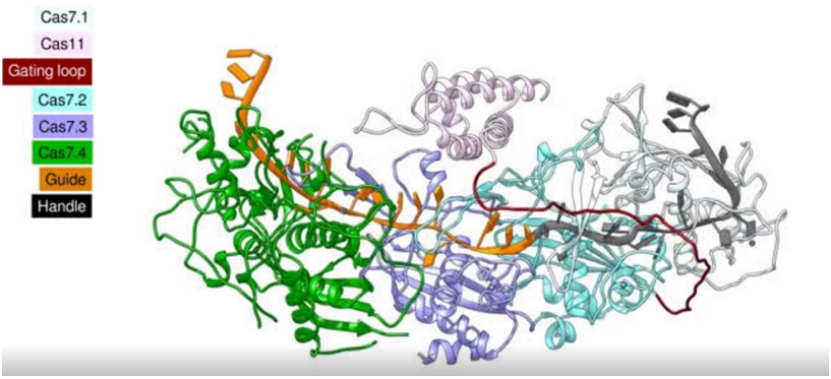
去年,来自麻省理工学院麦戈文脑科研究所的研究人员发现了III-E型CRISPR-Cas效应蛋白Cas7-11并表征了它的特征,它是第一种能够对RNA链进行精确、有指导性的切割而在此过程中不伤害细胞的CRISPR 酶(Nature, 2021, doi:10.1038/s41586-021-03886-5)。如今,在一项新的研究中,他们与东京大学的合作者合作,发现Cas7-11可以缩小到一个更紧凑的版本,使其成为编辑活细胞内RNA的一种更可行的选择。他们描述了这种新的、紧凑的Cas7-11,同时还对原始的Cas7-11进行了详细的结构分析(Cell, 2022, doi:10.1016/j.cell.2022.05.003)。
为了解析出Cas7-11的整体结构,这些作者使用了低温电镜技术,这种技术将电子束照射在冷冻的蛋白样本上,并测量电子束的传输方式。他们知道Cas7-11采用模块化架构,由Cas7.1、Cas7.2、Cas7.3、Cas7.4、Cas11、INS和CTE这个结构域和4个结构域间接头(linker)---L1、L2、L3和L4---组成,但是并不确定这些结构域究竟是如何折叠和组装在一起的。
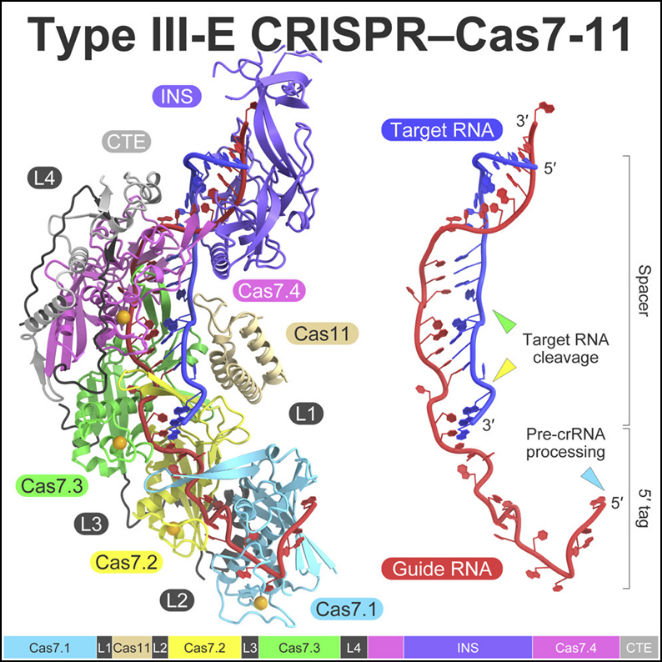
图片来自Cell, 2022, doi:10.1016/j.cell.2022.05.003。
他们发现Cas7-11通过使用Cas7.1结构域将它的前体CRISPR RNA(pre-crRNA)加工成crRNA,而且在crRNA的指导下,它还使用Cas7.2和Cas7.3结构域在两个确定的位点上切割靶RNA。该结构还揭示了Cas7-11的一个组成部分没有任何明显的功能作用。这一发现表明他们员可以将这个组成部分移除,重新设计Cas7-11,使它变得更小,而不影响它靶向RNA的能力。他们测试了去除这个组成部分的不同片段的影响,结果获得Cas7-11的一个新的紧凑版本,被称为Cas7-11S。有了Cas7-11S,他们将它装入在单个病毒载体中,将它递送到哺乳动物细胞中,并有效地靶向目标RNA。
12. Nature:首次使用非病毒精确基因组编辑开发出的neoTCR-T细胞在临床试验中治疗实体癌并取得积极进展
人类免疫系统在T细胞表面上有特定的受体,即T细胞受体(TCR),可以专门识别癌细胞并将它们与正常细胞区分开来。这些受体对每名患者来说都是不同的,因此找到一种有效的方法来分离它们并将它们导入到T细胞中,以产生一种个性化的T细胞疗法来治疗癌症,是使这种方法大规模可行的关键。
在一项新的研究中,来自美国加州大学洛杉矶分校和PACT制药公司的研究人员首次开发了一种基于CRISPR/Cas9非病毒精确基因组编辑的临床级方法,同时敲除T细胞中的两个内源性TCR基因:TCRα(TRAC)和TCRβ(TRBC),并在TRAC基因座上插入新抗原特异性TCR(neoantigen-specific TCR, neoTCR)的两条链,所使用的neoTCR是从患者自身的循环T细胞中分离出来的,由此获得的T细胞称为neoTCR-T细胞(Nature, 2022, doi:10.1038/s41586-022-05531-1)。
16名难治性实体癌(包括结肠癌、乳腺癌和肺癌)患者首次在neoTCR-T细胞剂量递增的1期临床试验(NCT03970382)中接受了多达三种不同的neoTCR-T细胞输注,每种neoTCR-T细胞都表达了患者特异性的neoTCR。一名患者出现了1级细胞因子释放综合征,一名患者出现了3级脑炎。在接受neoTCR-T细胞输注前,所有患者都接受清除淋巴细胞的化疗,并且在化疗后出现预期的副作用。在接受neoTCR-T细胞输注后,5名患者病情稳定,其他11名患者出现疾病进展。在接受neoTCR-T细胞治疗后的肿瘤活组织中检测到neoTCR-T细胞,其频率高于治疗前表达内源性TCR的T细胞。
这项新研究证实了分离和克隆多种识别突变新抗原的neoTCR、利用单步骤非病毒精确基因组编辑同时敲除内源性TCR和敲入neoTC和以临床等级制造neoTCR-T细胞的可行性,输注多达三种neoTCR-T细胞产品的安全性,以及将neoTCR-T细胞递送患者体内肿瘤中的能力。
13. Cell:我国科学家领衔开发出一种新的高通量基因靶向技术,绘制出小鼠基因扰动图谱
在一项新的研究中,来自中国上海科技大学、复旦大学和美国耶鲁大学医学院等研究机构的研究人员开发出一种称为iMAP(inducible mosaic animal for perturbation,诱导性嵌合动物扰动)的技术,作为一种转基因平台,它能够在小鼠体内平行地原位靶向至少100个基因。

图片来自Cell, 2022, doi:10.1016/j.cell.2022.06.039。
iMAP结合了Cre-loxP和CRISPR-Cas9技术的各自优点,并利用生殖系传递的转基因,该转基因携带了大量两侧引入两个loxP位点的、串联gRNA编码单元阵列。Cre介导的重组触发了这个阵列中所有gRNA的表达,但每个细胞中只有一个gRNA,从而将小鼠转化为嵌合生物,适合用于表型表征,也适合用于通过育种高通量获得传统的单基因扰动品系。通过使用gRNA作为读数,他们绘制了一个微型的Perturb-Atlas,对39个组织的90个基因的扰动进行了编目,这产生了对背景依赖性基因功能的丰富见解,并让人们得以一窥iMAP在基因组解码中的潜力。
14. Science:利用CRISPRa和CRISPRi揭示对人类T细胞功能至关重要的基因
CRISPR基因组编辑已成为剔除或改变DNA序列并研究其结果的有力工具。如今,在一项新的研究中,来自美国格拉德斯通研究所和加州大学旧金山分校的研究人员利用CRISPR-Cas9系统强行激活而不是编辑人类免疫细胞中的基因。这种称为CRISPR激活(CRISPR activation, CRISPRa)的方法让他们比以前更彻底、更迅速地发现在免疫细胞生物学中发挥作用的基因(Science, 2022, doi:10.1126/science.abj4008)。
这项研究是首次在直接从人身上分离出来的原代人类免疫细胞中大规模地成功使用CRISPRa。这些作者在不同的免疫细胞中激活了基因组中的每个基因,使他们能够平行地测试近2万个基因。这使他们能够迅速了解哪些提供了最有力的杠杆来重编程细胞功能的规则,以便最终可能导致更强大的免疫疗法。
15. Cell:新型CRISPR成像技术平台Perturb-map或能揭示控制机体肿瘤免疫力的特殊基因
尽管CRISPR筛查能帮助发现调节多种细胞内在过程的基因,但现有方法对于识别胞外基因的功能仍然并不理想,尤其是在组织背景下。

图片来自Cell, 2022, doi:10.1016/j.cell.2022.02.015。
在一项新的研究中,来自美国西奈山伊坎医学院等机构的研究人员利用基于CRISPR的成像技术平台Perturb-map发现了两条关键通路,其或许对于肿瘤生长、肿瘤架构和机体免疫细胞的招募有着非常深远的影响,其中一条通路是由细胞因子干扰素γ(IFNγ)所控制,另一条则是由肿瘤生长因子β受体(TGFβR)所控制,研究者发现,当TGFβR2基因或编码IFNγ的调节子SOCS1的基因被从癌细胞中剔除后,肺部肿瘤就会变得更大且更丰富。尽管这两种基因的缺失对肿瘤的生长会产生类似的影响效应,但通过Perturb-map平台对肿瘤进行成像结果或能揭示,SOCS1肿瘤会被T细胞高度浸润,而TGFβR肿瘤则会排斥T细胞,即使当SOCS1和TGFβR肿瘤直接接触时,前者仍然会被浸润,而后者则会被排斥,这或许是一项重要的研究发现,因为机体肿瘤中含有较少免疫细胞的患者对免疫疗法药物的反应较差。
16. Nat Biotechnol:利用AAV病毒载体递送CRISPR/Cas9,让B细胞产生抵抗HIV的广泛中和抗体
在一项新的研究中,来自以色列特拉维夫大学的研究人员利用两种腺相关病毒(AAV)载体同时递送 CRISPR-Cas9 基因编辑系统和靶向免疫球蛋白基因的修复模板,直接对小鼠内源 性B 细胞的免疫球蛋白基因位点进行编辑,从而高效地在小鼠体内对 B 细胞进行基因修饰,使它们能够产生抵抗 HIV 病毒的广泛中和抗体---3BNC117,这就使得利用一次性递送AAV病毒载体递送CRISPR/Cas9治疗HIV感染成为可能(Nature Biotechnology, 2022, doi:10.1038/s41587-022-01328-9)。
当然,这项技术还存在一些限制:抗体滴度仍要更高、更稳定才能确保治疗效果和减少免疫接种次数;需要结合多种互补的广泛中和抗体(bNAbs)提高对 HIV 的免疫预防;在没有加强免疫的情况下,这些抗体反应是否持续尚不清楚;以及最重要的安全性问题,是否会引起不良免疫反应,是否会导致因 AAV 染色体随机整合驱动的致癌事件。
17. Nature:重新设计的Cas9版本可让基因编辑更安全
在人类身上使用基于CRISPR的基因编辑所面临的巨大挑战之一是这种分子机器有时会对宿主基因组的不正确位点进行修改,这就造成了一种可能性,即试图修复基因组中一个位点的基因突变可能会意外地在另一个位点产生危险的新突变。
但是如今,在一项新的研究中,来自美国德克萨斯大学奥斯汀分校的研究人员对一种广泛使用的基于CRISPR的基因编辑工具中的一个关键组件---Cas9---进行重新设计,使得它靶向错误的DNA片段的可能性降低数千倍,同时保持与原始的Cas9版本一样的效率,使得它可能更加安全(Nature, 2022, doi:10.1038/s41586-022-04470-1)。
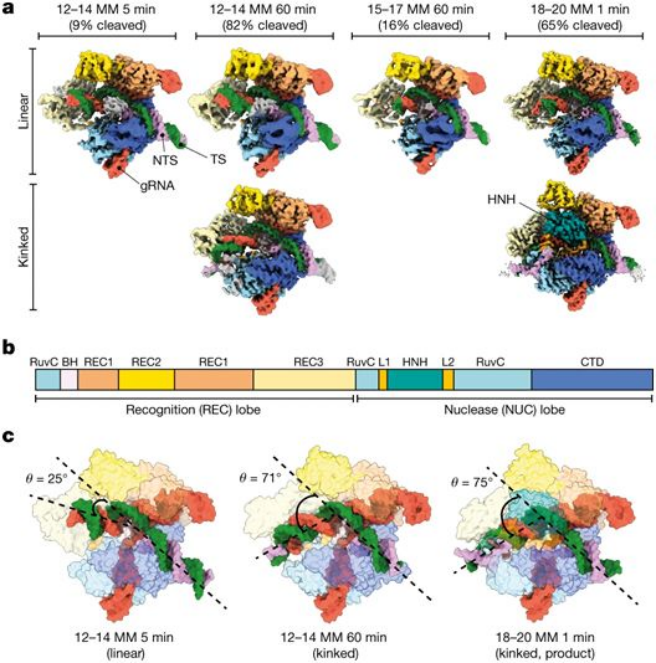
错配诱导的Cas9构象中间体,图片来自Nature, 2022, doi:10.1038/s41586-022-04470-1。
其他实验室已经重新设计了Cas9以减少脱靶相互作用,但到目前为止,所有这些版本都是通过牺牲速度来提高准确性。在这项新的研究中,这些作者开发的Cas9新版本---SuperFi-Cas9---切割脱靶位点的可能性降低了4000倍,但与天然的Cas9一样快。到目前为止,他们展示了SuperFi-Cas9在试管中的DNA上的应用。他们如今正与其他科学家合作,计划测试SuperFi-Cas9在活细胞中的基因编辑。他们还在努力开发更安全、更活跃的Cas9版本。
18. Science子刊:我国科学家领衔揭示利用新型纳米胶囊递送CRISPR-Cas9有望安全有效地治疗胶质母细胞瘤
在一项新的研究中,来自中国河南大学和澳大利亚麦考瑞大学等研究机构的研究人员开发出一种纳米胶囊,它能够穿越血脑屏障(BBB),携带CRISPR-Cas9编辑工具来治疗脑瘤。这种纳米胶囊的外壳是用一种二硫化物交联的聚合物制成的,然后用一种Angiopep-2肽进行点缀。添加该肽是为了产生中性的表面电荷,这样它就不会被核糖核酸酶攻击。这种纳米胶囊的外壳大到足以容纳CRISPR-Cas9复合物,但仍然足够小(大约30纳米长),使它能够通过血脑屏障。
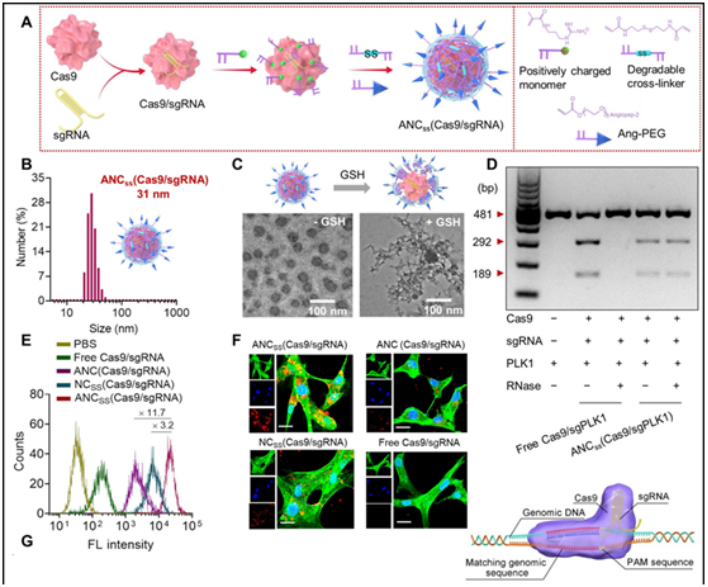
Cas9/sgRNA纳米胶囊的制备、物理性质和细胞功能,图片来自Science Advances, 2022, doi:10.1126/sciadv.abm8011。
他们在胶质母细胞瘤小鼠模型中测试了他们的纳米胶囊递送系统。每只小鼠都进行了一次尾部注射---一些小鼠接受了这种新开发的纳米胶囊递送系统,而另一些则接受了对照处理。他们发现,接受这种新疗法的小鼠的平均生存时间为68天,而对照组为24天。他们还发现在其他脑组织中,不希望发生的基因突变率不到0.5%。
19. Science子刊:开发出一种大幅降低脱靶突变和在靶突变的基因编辑工具
基因编辑工具CRISPR-Cas9可用于治疗遗传性血液疾病,但这可能会造成意外的遗传改变。在一项新的研究中,来自德国马克斯-德布吕克分子医学中心(MDC)的Klaus Rajewsky和Van Trung Chu及其研究团队提出了一种称为“spacer-nick”的新型改良方法。这种方法采用了一对改良的称为缺口酶(nickase)的分子剪刀,在DNA的两条链的不同位点上产生缺口,将这种不良后果降到最低。但是这对用于基因编辑的缺口酶能够更精确地检测和修复有缺陷的基因,这在很大程度上取决于这些作者在这种基因编辑工具中内置的一个间隔序列(spacer)。他们使用这个间隔序列来确保所产生的两个缺口相隔200到350个碱基对,并避免发生DNA双链断裂。针对造血干细胞和T细胞进行的实验表明,这是让在靶突变和脱靶突变最小化的最佳距离(Science Advances, 2022, doi:10.1126/sciadv.abm9106)。
Chu报告说,他们甚至可以定量确定他们微调的基因剪刀的优越性:“使用经典的CRISPR-Cas9方法,在超过40%的干预中会发生在靶突变。这种spacer-nick系统可以将这一比例降至2%以下。”Chu解释说,脱靶突变的成功率不能很容易和准确地确定:“我们真正能说的是,当使用经典的基因剪刀时,脱靶突变发生得相对频繁,但在我们的方法中却很少发生,甚至不存在。暂时还不清楚的是,在spacer-nick切割之后,遗传物质被修复的确切机制。Chu说,“它似乎不是通过众所周知的---而且容易出错的---NHEJ途径发生的。”
20. Science子刊:利用CRISPR-Cas13沉默神经系统中的靶基因,有望治疗一系列神经系统疾病
正如肌萎缩性脊髓侧索硬化症(ALS)或亨廷顿舞蹈病等神经退行性疾病所展示的那样,单个基因突变可以产生深远的影响。在一项新的研究中,来自美国伊利诺伊大学厄巴纳-香槟分校的研究人员在小鼠的中枢神经系统中使用了一种针对性的CRISPR技术来关闭可导致ALS和亨廷顿舞蹈病的突变蛋白的产生(Science Advances, 2022, doi:10.1126/sciadv.abk2485)。
与流行的DNA编辑CRISPR-Cas9技术不同,这种新方法使用CRISPR-Cas13,它可以靶向信使RNA(mRNA)---由DNA转录而来,携带着蛋白蓝图。在这项新的研究中,这些作者开发出Cas13系统,靶向并切割编码引发ALS和亨廷顿舞蹈病的突变蛋白的mRNA,在不干扰细胞DNA的情况下有效地沉默突变基因。

源自黄化瘤胃球菌的Cas13d(RfxCas13d)经编程后靶向hSOD1,图片来自Science Advances, 2022, doi:10.1126/sciadv.abk2485。
这些作者将CRISPR-Cas13系统注射到携带导致ALS或亨廷顿舞蹈病的基因突变的小鼠的中枢神经系统。腺相关病毒(AAV)载体将这种RNA靶向系统递送到细胞中。Gaj说,AAV载体是一种有前途的基因治疗工具,部分原因是它们能够进入脊髓和大脑中的细胞。他们发现CRISPR-Cas13有效地减少了这两种疾病的神经系统中存在的突变蛋白的数量---特别是ALS小鼠脊髓中的蛋白SOD1,以及亨廷顿舞蹈病小鼠大脑中的亨廷顿蛋白(huntingtin)。SOD1突变蛋白的减少也与更好的治疗效果相关联。与未接受治疗的小鼠相比,接受CRISPR-Cas13注射的ALS小鼠的疾病进展较慢,生存率提高,握力和运动技能的下降速度较慢。
其实,科学家们针对CRISPR-Cas基因编辑的研究不胜枚举,以上罗列的仅是其中的一小部分。当然,迄今为止,涉及CRISPR-Cas的疾病治疗研究大体上仍然局限在细胞模型和动物模型上,这是因为诸如CRISPR/Cas9之类的基因编辑技术存在着编辑效率较低和脱靶效应等缺点,因此贸然进行开展人体临床试验,会引发难以预测的结果,这不奇怪当中国科学家贺建奎声称世界上首批经过基因编辑的婴儿出生时,国内外的口诛笔伐纷至杳来。不过,在未来,科学家们将从病理学、分子生物学、基因、蛋白和组学等不同角度深入探究不同类型的CRISPR/Cas系统及其改进版本等基因编辑技术的详细作用机制,人们最终有朝一日能够利用基因编辑技术治疗HIV感染、癌症、血液系统疾病、神经系统疾病和遗传疾病等一系列疾病。
参考文献:
1. Jonathan Strecker et al. RNA-activated protein cleavage with a CRISPR-associated endopeptidase, Science, 2022, doi:10.1126/science.add7450.
2. Kazuki Kato et al. RNA-triggered protein cleavage and cell growth arrest by the type III-E CRISPR nuclease-protease, Science, 2022, doi:10.1126/science.add7347.
3. Basem Al-Shayeb et al. Diverse virus-encoded CRISPR-Cas systems include streamlined genome editors, Cell, 2022, doi:10.1016/j.cell.2022.10.020.
4. Giorgio Ottaviano et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia, Science Translational Medicine, 2022, doi:10.1126/scitranslmed.abq3010.
5. Brian Shy et al. High-yield genome engineering in primary cells using a hybrid ssDNA repair template and small-molecule cocktails. Nature Biotechnology, 2022, doi:10.1038/s41587-022-01418-8.
6. Chunyi Hu et al. Craspase is a CRISPR RNA-guided, RNA-activated protease. Science, 2022, doi:10.1126/science.add5064.
7. Miguel M. Álvarez et al. TP53-dependent toxicity of CRISPR/Cas9 cuts is differential across genomic loci and can confound genetic screening. Nature Communications, 2022, doi:10.1038/s41467-022-32285-1.
8. Santi Bhattarai-Kline et al. Recording gene expression order in DNA by CRISPR addition of retron barcodes. Nature, 2022, doi:10.1038/s41586-022-04994-6.
9. Rhiannon B. Werder et al. CRISPR interference interrogation of COPD GWAS genes reveals the functional significance of desmoplakin in iPSC-derived alveolar epithelial cells. Science Advances, 2022, doi:10.1126/sciadv.abo6566.
10. Nahmad, A.D., Reuveni, E., Goldschmidt, E. et al. Frequent aneuploidy in primary human T cells after CRISPR–Cas9 cleavage. Nature Biotechnology, 2022, doi:10.1038/s41587-022-01377-0.
11. Joseph M. Replogle et al. Mapping information-rich genotype-phenotype landscapes with genome-scale Perturb-seq. Cell, 2022, doi:10.1016/j.cell.2022.05.013.
12. Kazuki Kato et al. Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex. Cell, 2022, doi:10.1016/j.cell.2022.05.003.
13. Susan P. Foy et al. Non-viral precision T cell receptor replacement for personalized cell therapy, Nature, 2022, doi:10.1038/s41586-022-05531-1.
14. Bo Liu et al. Large-scale multiplexed mosaic CRISPR perturbation in the whole organism. Cell, 2022, doi:10.1016/j.cell.2022.06.039.
15. Ralf Schmidt et al. CRISPR activation and interference screens decode stimulation responses in primary human T cells. Science, 2022, doi:10.1126/science.abj4008.
16. Maxime Dhainaut et al. Spatial CRISPR genomics identifies regulators of the tumor microenvironment. Cell, 2022, doi:10.1016/j.cell.2022.02.015.
17. Alessio D. Nahmad et al. In vivo engineered B cells secrete high titers of broadly neutralizing anti-HIV antibodies in mice. Nature Biotechnology, 2022, doi:10.1038/s41587-022-01328-9.
18. Jack P. K. Bravo et al. Structural basis for mismatch surveillance by CRISPR–Cas9 . Nature, 2022, doi:10.1038/s41586-022-04470-1.
19. Yan Zou et al. Blood-brain barrier–penetrating single CRISPR-Cas9 nanocapsules for effective and safe glioblastoma gene therapy. Science Advances, 2022, doi:10.1126/sciadv.abm8011.
20. Ngoc Tung Tran et al. Precise CRISPR-Cas–mediated gene repair with minimal off-target and unintended on-target mutations in human hematopoietic stem cells. Science Advances, 2022, doi:10.1126/sciadv.abm9106.
21. Jackson E. Powell et al. Targeted gene silencing in the nervous system with CRISPR-Cas13. Science Advances, 2022, doi:10.1126/sciadv.abk2485.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


