Cell:高彩霞/刘俊杰/张勇等揭示CRISPR系统起源的关键分子机制
来源:生物世界 2025-10-02 09:32
首次发现并定义了连接转座子与 CRISPR 之间长期缺失的关键进化中间体,将其命名为——TranC(Transposon-CRISPR intermediate)。
CRISPR-Cas 系统是原核生物的获得性免疫系统,能够在 CRISPR RNA 的指导下特异性切割入侵的外源核酸。其中,分别以 Cas9 和 Cas12 为效应蛋白的 type II 类和 V 类 CRISPR 系统已成为当前基因组编辑的重要工具,广泛应用于基础研究、医学和农业等多个领域。
已有研究表明,Cas12 起源于 IS200/605 和 IS607 转座子家族编码的 TnpB 核酸酶。TnpB 蛋白广泛存在于细菌和古菌的转座子中,是原核生物中最庞大、最丰富的转座子相关核酸酶家族之一。
TnpB 和 Cas12 在种类和结构上具有高度多样性,从 TnpB 到 Cas12 的进化被认为是“多次独立起源”的转座子-免疫系统复杂进化事件。阐明 CRISPR 系统从转座子起源的分子机制,是该领域长期悬而未解的科学难题。
2025 年 9 月 29 日,中国科学院遗传与发育生物学研究所高彩霞团队联合清华大学生命科学学院刘俊杰副教授、中国科学院动物研究所张勇研究员,在国际顶尖学术期刊 Cell 期刊发表了题为:Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons 的研究论文。
研究团队历经 7 年深入探索,首次发现并定义了连接转座子与 CRISPR 之间长期缺失的关键进化中间体,将其命名为——TranC(Transposon-CRISPR intermediate),弥合了 CRISPR 进化历程中的缺口。
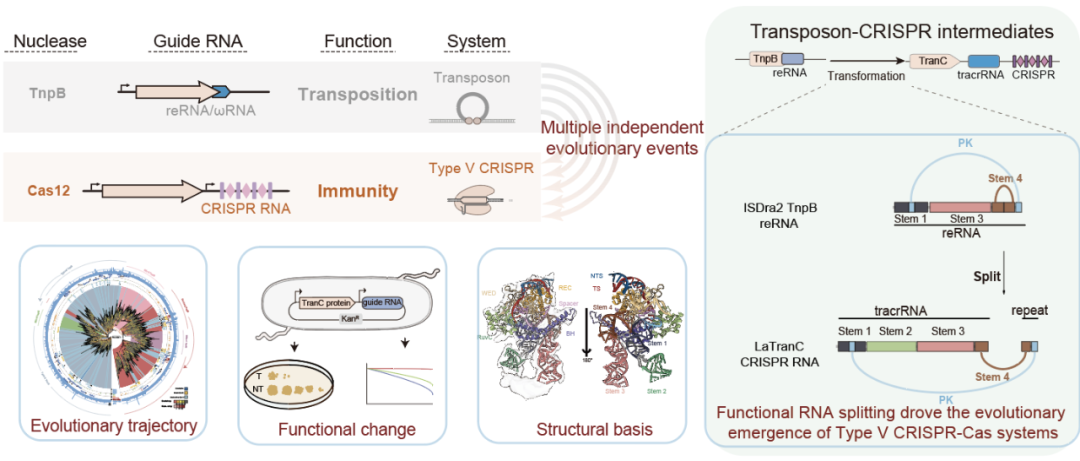
该研究揭示,驱动 TnpB 转座酶向 Cas12 系统演化的核心机制源于引导 RNA 的“功能性分裂”,而非蛋白质结构的根本性改变。这一发现不仅破解了 Cas12 起源的分子机制之谜,也首次以实验证据阐明了 RNA 层面的创新如何驱动复杂分子机器的进化进程。

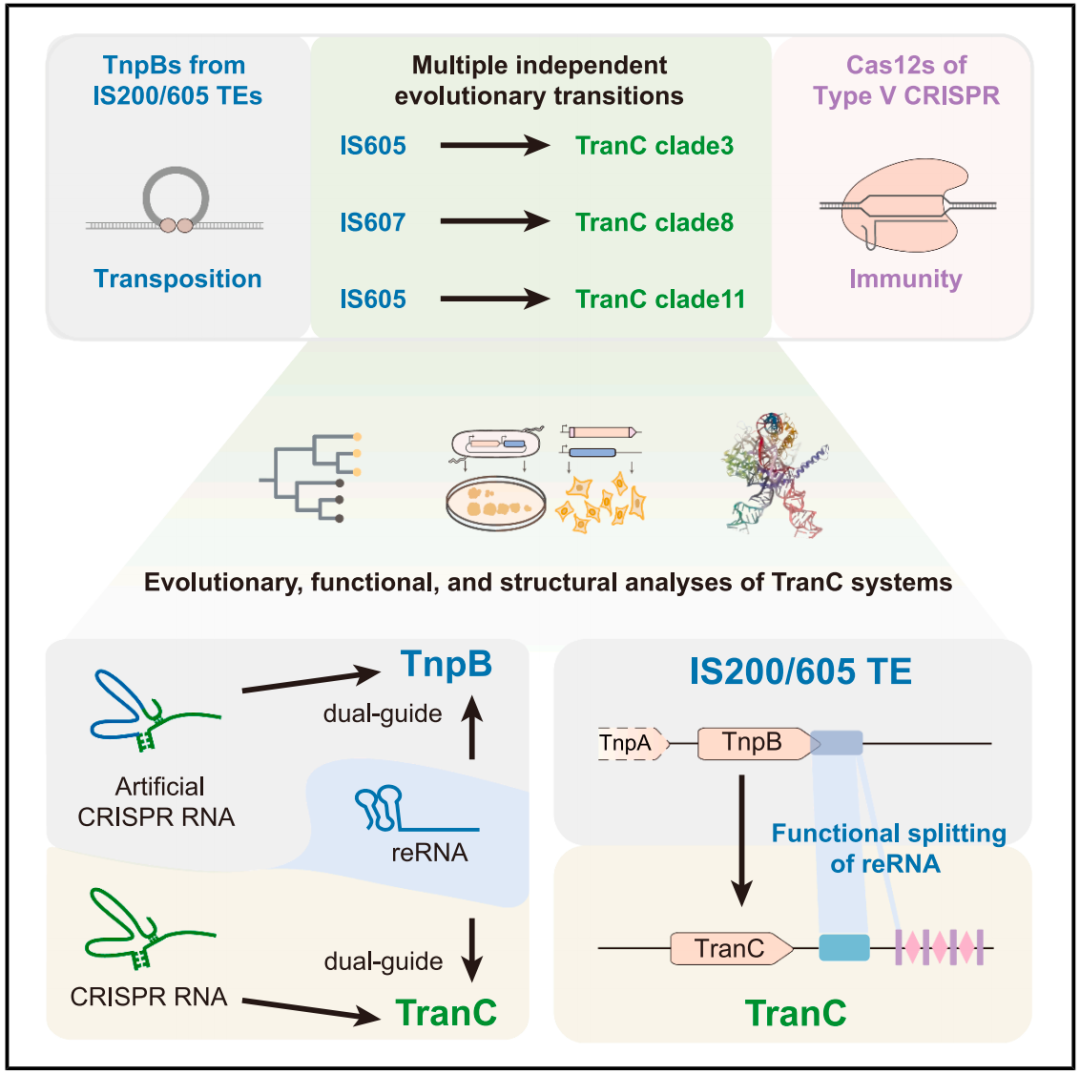
在这项最新研究中,研究团队首先结合序列相似性、共享结构域特征和保守催化基序三重搜索方法,从原核生物基因组与宏基因组数据中,鉴定出 146 个与 TnpB 亲缘关系较近的 CRISPR 偶联候选蛋白。通过系统发育分析、AlphaFold 结构预测与功能元件对比,最终从中鉴定出 6 个演化中间家族,命名为 TranC。这些 TranC 系统均与特定的 TnpB 分支构成姊妹群体,代表了 TnpB 向 Cas12 演化过程中的多个独立起源路径。其中,除此前已报道的 Clade 8(Cas12n,源自 IS607 家族)外,研究新鉴定的 Clade 3、11、12、13、14 源自 IS605 转座子家族,进一步描绘了 Cas12 系统多次“独立演化”的复杂场景。
功能实验证实,TranC 系统具有独特的“双 RNA 导向机制”。在大肠杆菌体系中,来自多个支系的五个代表性 TranC 系统不仅能够使用自身编码的 CRISPR RNA(tracrRNA + crRNA)进行靶向切割,还保留了祖先 TnpB 使用 reRNA(transposon-derived right-end RNA,亦称作 ωRNA)作为向导的能力。进一步在人类细胞体系开展的基因组编辑实验显示,来自 Clade 3、源于人类肠道细菌 Lawsonibacter sp. 的 LaTranC 系统,能够同时使用 CRISPR RNA 与 ISDra2 TnpB 的 reRNA 进行基因组编辑。这种兼具两种 RNA 识别能力的“双重导向”机制,是 TranC 系统作为 TnpB 向 Cas12 演化中间体的重要功能标志,也为理解 CRISPR 系统的起源提供了关键证据。
进一步的结构生物学分析揭示,TranC 蛋白与其祖先 TnpB 蛋白在三维结构上高度保守,其差异主要体现在 RNA 层面。冷冻电镜解析表明,LaTranC 与 CRISPR RNA(tracrRNA + crRNA)形成的复合体,与其姊妹分支 ISDra2 TnpB 与 reRNA 形成的复合体在结构上高度一致。结构对比首次观测到,TnpB 系统中由单一 reRNA 构成的向导 RNA,在 TranC 中演化为功能分离的 tracrRNA 与 crRNA 两个模块,从而建立起典型的 CRISPR 双 RNA 导向结构。这一“RNA 功能性分裂”的现象不仅在 LaTranC 中被验证,也通过 AlphaFold 结构预测和 RNA 共变异分析在其他 TranC 支系中被普遍观察,提示其为 Cas12 系统多次独立起源过程中的趋同进化特征。
为进一步验证 RNA 分裂在 CRISPR 系统起源中的关键作用,研究团队通过实验模拟了从 TnpB 到 TranC 的演化路径。结果表明,仅通过将 ISDra2 TnpB 的 reRNA 模块功能性拆分为嵌合型 tracrRNA 与 LaTranC 来源的 crRNA 两部分,即可赋予其利用 CRISPR 阵列进行靶向识别与基因组切割的能力。换而言之,该系统已从单 RNA 导向的 TnpB 机制转变为双 RNA 导向的类 CRISPR 机制。
TranC 系统的发现揭示了 CRISPR 起源的关键分子机制
综上所述,该研究通过多学科方法,首次明确指出,RNA 层面的功能性分裂与模块化创新,而非蛋白结构的根本性改变,是驱动 CRISPR-Cas 系统由转座子演变为免疫系统的核心分子机制。
中国科学院遗传与发育生物学研究所高彩霞研究员、清华大学生命科学学院刘俊杰副教授、中国科学院动物所张勇研究员为该论文共同通讯作者。中国科学院遗传与发育生物学研究所已出站博士后靳帅、已毕业博士生朱子旭、博士生李运嘉和清华大学已出站博士后张寿悦为该论文共同第一作者。中国科学院遗传与发育生物学研究所已毕业博士生刘怡静、博士生程志恒、博士生杨广磊、助理研究员李洪超、助理研究员梁荣洪与副研究员张瑞,清华大学生命科学学院已毕业博士生李丹苑、中国科学院动物研究所李源清博士、中国科学院微生物研究所骆迎峰研究员、邱金龙研究员,齐禾生物科技有限公司首席技术官赵天萌博士、高强博士等在课题研究中做出了重要贡献。上海科技大学季泉江教授、中国科学院微生物研究所吴边研究员为该研究提供了重要实验材料。研究得到农业农村部项目、国家重点研发计划、国家自然科学基金、中国科学院稳定支持基础研究领域青年团队项目和新基石科学基金项目的资助。
此外,齐禾生物科技有限公司赵天萌团队与中国科学院遗传与发育生物学研究所高彩霞团队合作,对 LaTranC 基因组编辑系统进行了系统性的蛋白定向进化与向导 RNA 工程改造,成功获得具备完全自主知识产权的高效变体——TranC11a(574个氨基酸)。在真核细胞的基因组编辑实验中,TranC11a 显著优于现有小型核酸酶系统(例如 TnpB 和 IscB),并在部分位点展现出与 SpCas9 系统相当的编辑效率。该研究以:Directed evolution of compact TranC11a systems for efficient genome editing 为题,在预印本平台 bioRxiv 上线。

TranC 系列相关核心专利已通过国家知识产权局的自由实施(FTO)审查,为其在生物医药和农业育种等关键领域的转化应用奠定了坚实基础。目前,TranC11a 质粒已通过 Addgene 平台面向全球科研人员开放获取,并可通过齐禾生科获得专业技术支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


