诺奖团队最新论文:利用CRISPR系统,实现活细胞内单分子RNA成像
来源:生物世界 2025-02-20 11:47
研究团队利用 smLiveFISH 技术分析了两种不同 mRNA(NOTCH2 和 MAP1B)的行为,这两种 mRNA 分别编码一种细胞表面受体蛋白和一种微管相关蛋白。
要了解单细胞中单个 RNA 分子的多种动态行为,就需要实时以高分辨率对其进行可视化。然而,目前还没有能够以通用的方式实现对未经修饰的内源性 RNA 进行单分子活细胞成像。
2025年2月18日,诺贝尔化学奖得主、CRISPR 基因编辑技术先驱 Jennifer Doudna 教授团队在 Nature Biotechnology 期刊发表了题为:Single-molecule live-cell RNA imaging with CRISPR–Csm 的研究论文。
该研究开发了一种基于 CRISPR–Csm 系统的单分子活细胞荧光原位杂交(smLiveFISH)技术,能够直接且高效地在可视化各种细胞类型中的任何未经修饰的单个 RNA 分子。利用 smLiveFISH 技术,研究团队在活细胞中追踪单个天然 NOTCH2 和 MAP1B 的 mRNA 转录本,并确定了其 mRNA 的不同的定位机制。

RNA 直接参与蛋白质合成,并在转录和转录后水平调控基因表达。除了 RNA 序列之外,个体转录本的时空分布也控制着这些活动。事实上,RNA、RNA结合蛋白(RBP)和其他细胞机制之间的动态和协调的相互作用发生在特定的亚细胞区域和时间点。
活细胞 RNA 成像方法已开始揭示单个细胞内的 RNA 动态,彰显了此类研究的重要价值。然而,基于茎环结构(Stem Loop)或适体(Aptamer)的标记方法需要在 RNA 特定区域插入基因序列,或依赖外源表达标记 RNA——这些操作既耗时又可能干扰 RNA 的天然行为。使用分子信标或 CRISPR-Cas 系统实现未修饰内源 RNA 可视化的方法,则受限于单分子分辨率不足(通常仅限于高丰度重复 RNA )以及内体滞留探针或过表达荧光蛋白融合体产生的严重背景噪声。
尽管现有方法在特定案例中已获成功,但开发通用型单分子水平活细胞原生 RNA 成像平台的需求日益迫切。
在这项最新研究中,研究团队开发了一种单分子活细胞荧光原位杂交(single-molecule live-cell fluorescence in situ hybridization,smLiveFISH)技术,该技术利用来自嗜热链球菌的 RNA 靶向的 III-A 型 CRISPR-Csm 系统以及多重向导 RNA,能够直接且高效地在包括原代细胞在内的多种细胞类型中对任何未经修饰的单个 RNA 分子进行可视化成像,从而追踪不同种类活细胞中的单个 mRNA 分子。
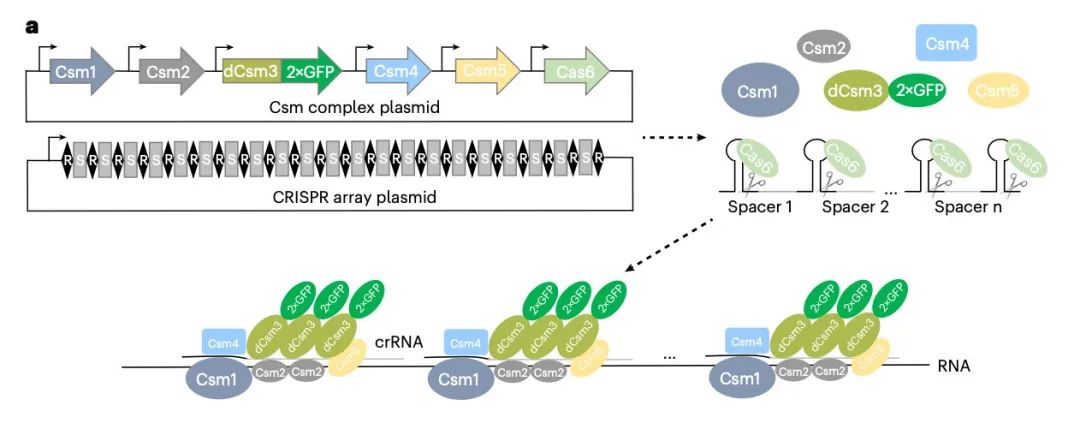
smLiveFISH成像天然单个 mRNA 分子的原理
研究团队利用 smLiveFISH 技术分析了两种不同 mRNA(NOTCH2 和 MAP1B)的行为,这两种 mRNA 分别编码一种细胞表面受体蛋白和一种微管相关蛋白。结果显示,NOTCH2 的 mRNA 包含两种具有不同动态特性的群体,这与共翻译多肽在内质网(ER)中的跨膜转运有关。相比之下,MAP1B 的 mRNA 表现出几种不同的行为,包括以不依赖翻译的方式进行定向运输。

活细胞中单个 NOTCH2 mRNA 的动态变化
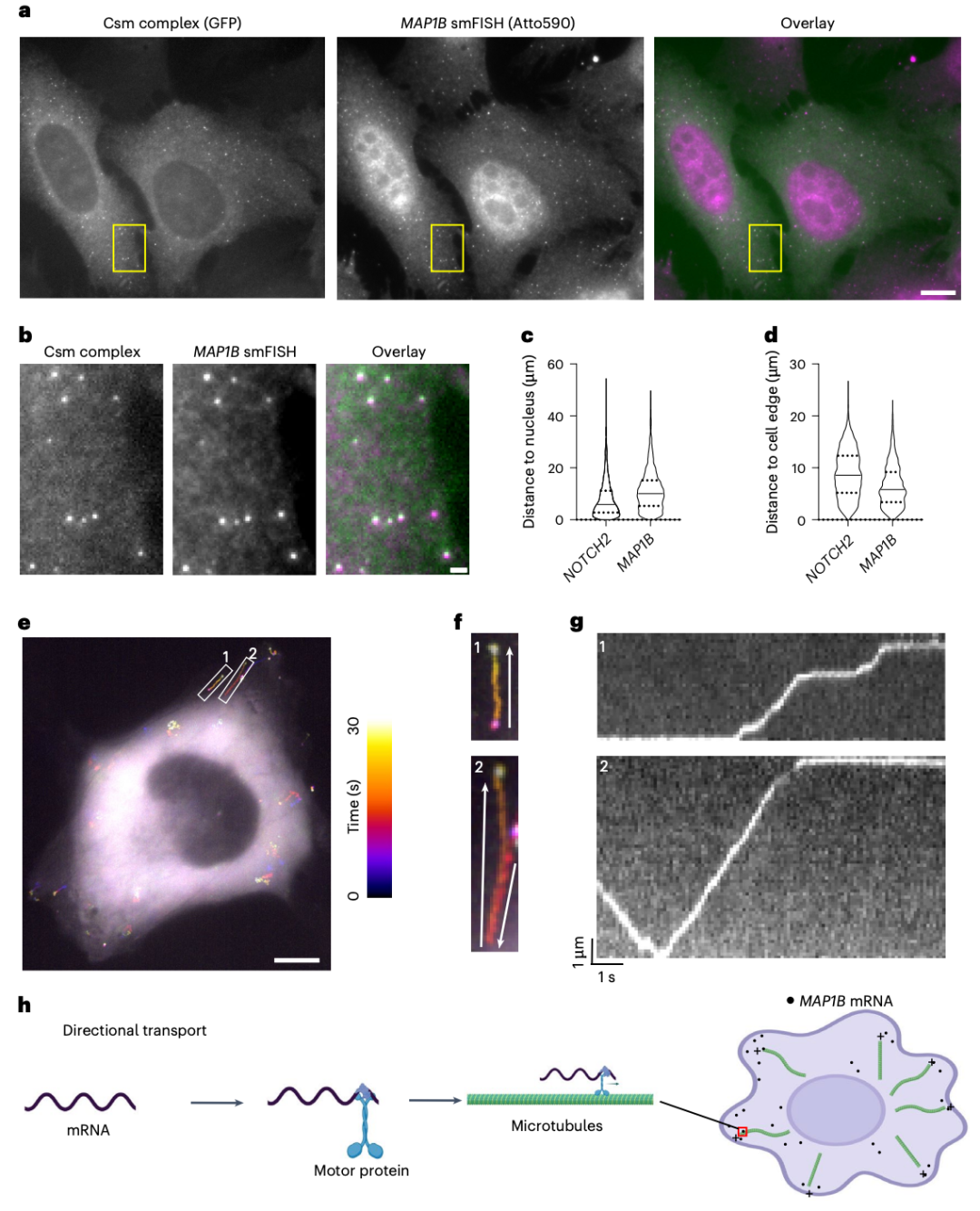
活细胞中单个 MAP1B mRNA 的动态变化
研究团队进一步证明,smLiveFISH 能够检测出单个转录本在对小分子作出反应时定位上的差异,例如被整合入 mRNA 处理小体(也叫做 P 小体),这突显了 smLiveFISH 技术在评估单个内源性 RNA 行为中的实用性。因此,smLiveFISH 技术有可能揭示健康和疾病状态下天然转录本的时空组织原理。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


