科研人员开发uCoTargetX技术实现单细胞多组蛋白修饰与转录并行检测
来源:网络 2024-01-16 15:48
研究者在此基础上进一步优化革新,发展了uCoTargetX技术,在国际上率先实现单细胞多种组蛋白修饰和转录组的并行检测,为深入研究各种生理病理状态下细胞命运的表观调控提供了强有力的技术工具。
北京大学未来技术学院、北京大学-清华大学生命科学联合中心何爱彬教授团队于Science Advances期刊发表了题为“ Single-cell joint profiling of multiple epigenetic proteins and gene transcription”的文章,报道了一种名为uCoTarget(ultra-high throughput Combined TAgmenting enRichment for multiple epiGEneTic proteins in the same cells)的单细胞多维表观检测新技术,能够在单个细胞同时捕获高达5种不同的蛋白质-染色质互作信息。研究者在此基础上进一步优化革新,发展了uCoTargetX技术,在国际上率先实现单细胞多种组蛋白修饰和转录组的并行检测,为深入研究各种生理病理状态下细胞命运的表观调控提供了强有力的技术工具。
何爱彬团队前期已经积累了丰富的单细胞表观多组学技术开发经验【6—8】。针对上述技术瓶颈,研究者优化实验体系,采取逐步添加带有特定标签的抗体-PAT-T7复合物的方案,规避了其他技术中共价交联和纯化的繁琐步骤,并且不依赖物种特异的一抗和特殊的微流控设备,极大提升了uCoTarget技术的简易性和可推广性(图1)。研究者首先在K562和Kasumi-1细胞系中同时对多达5种激活或抑制性组蛋白修饰(H3K27ac、H3K4me3、H3K4me1、H3K36me3和H3K27me3)进行检测,证明了uCoTarget实验的真实性、可靠性和稳定性。为了揭示转录因子和组蛋白修饰在造血干祖细胞产生过程中细胞命运决定的协同或异步作用,研究者结合体外分化模型,利用uCoTarget同时捕获RUNX1、标记活性增强子的组蛋白修饰H3K27ac以及抑制性组蛋白修饰H3K27me3。RUNX1-H3K27ac染色质速率(chromatin velocity)和轨迹分析,能在一定程度上预测心脏和造血谱系的分化路径,并提示RUNX1可能先于H3K27ac发挥驱动作用。进一步,研究人员巧妙地将uCoTarget与基于组合标签的转录组捕获技术相结合,开发出uCoTargetX,率先实现单细胞全基因组范围内多种蛋白-染色质的相互作用和mRNA同时捕获。同一细胞内多维度多种类的分子信息,可以真实准确地反映多维组蛋白修饰模式如何协同或拮抗调控基因表达,有助于我们全面理解生命发育及基因表达调控的多样性和复杂性。
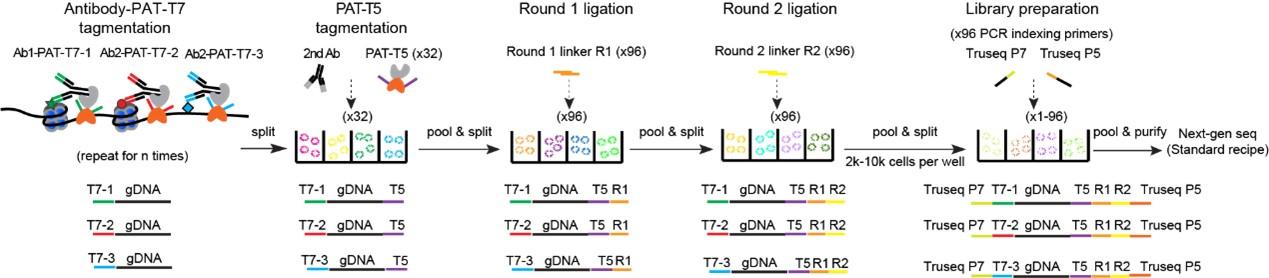
图1. 超高通量单细胞表观多组学技术uCoTarget流程图
北京大学未来技术学院已毕业博士王千昊与中国医学科学院血液学研究所熊海清研究员为论文共同第一作者。何爱彬和熊海清为本文的共同通讯作者。该研究获得了科技部干细胞专项、国家自然科学基金委和生命科学联合中心的支持。
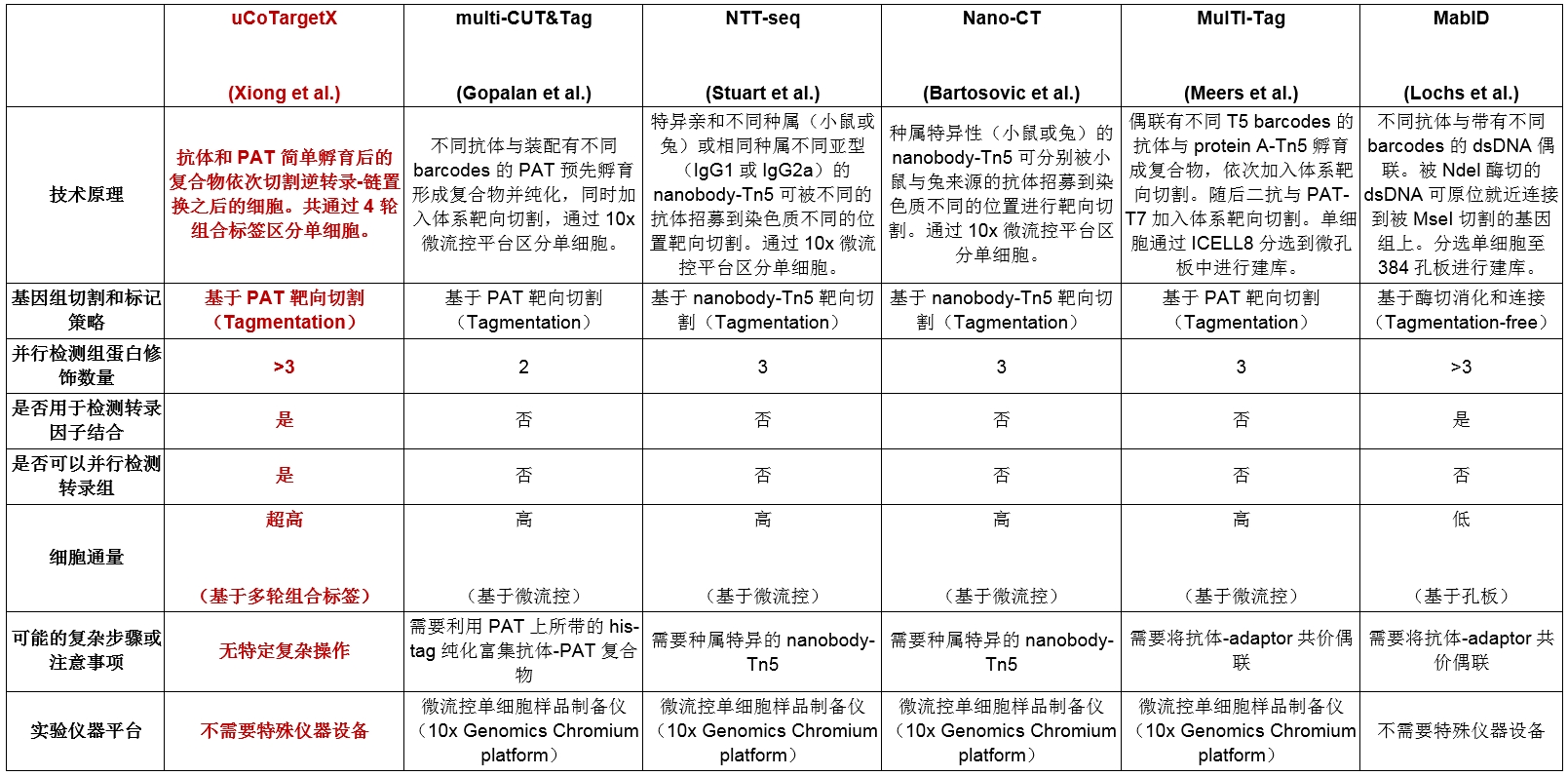
附:不同单细胞多组蛋白修饰技术特点比较
参考文献:
1. Gopalan S, Wang Y, Harper N W, et al. Simultaneous profiling of multiple chromatin proteins in the same cells. Molecular cell, 2021, 81(22): 4736-4746. e5.
2. Stuart T, Hao S, Zhang B, et al. Nanobody-tethered transposition enables multifactorial chromatin profiling at single-cell resolution. Nature Biotechnology, 2023, 41(6): 806-812.
3. Bartosovic M, Castelo-Branco G. Multimodal chromatin profiling using nanobody-based single-cell CUT&Tag. Nature Biotechnology, 2023, 41(6): 794-805.
4. Meers M P, Llagas G, Janssens D H, et al. Multifactorial profiling of epigenetic landscapes at single-cell resolution using MulTI-Tag. Nature Biotechnology, 2023, 41(5): 708-716.
5. Lochs S J A, van der Weide R H, de Luca K L, et al. Combinatorial single-cell profiling of major chromatin types with MAbID. Nature Methods, 2023: 1-11.
6. Wang Q, Xiong H, Ai S, et al. CoBATCH for high-throughput single-cell epigenomic profiling. Molecular cell, 2019, 76(1): 206-216. e7.
7. Ai S, Xiong H, Li C C, et al. Profiling chromatin states using single-cell itChIP-seq. Nature Cell Biology, 2019, 21(9): 1164-1172.
8. Xiong H, Luo Y, Wang Q, et al. Single-cell joint detection of chromatin occupancy and transcriptome enables higher-dimensional epigenomic reconstructions. Nature Methods, 2021, 18(6): 652-660.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


