李博文等利用机器学习+组合化学,加速发现用于mRNA递送的可电离脂质
来源:生物世界 2024-05-20 14:26
该研究通过将机器学习和组合化学相结合,建立了一种加速发现和评估可电离脂质的方法,促进了用于精准递送mRNA的脂质纳米颗粒(LNP)的开发。
多伦多大学李博文、麻省理工学院 Daniel Anderson 等在 Nature 子刊 Nature Materials 上发表了题为:Accelerating ionizable lipid discovery for mRNA delivery using machine learning and combinatorial chemistry 的研究论文。
该研究通过将机器学习和组合化学相结合,建立了一种加速发现和评估可电离脂质的方法,从中发现了可电离脂质119-23,其在转染数种组织的肌肉和免疫细胞方面的效果优于已确立的基准脂质,该研究促进了用于精准递送mRNA的脂质纳米颗粒(LNP)的开发。

在过去几年中,脂质纳米颗粒(LNP)取得了实质性的临床进展,美国FDA于2018年批准了来自Alnylam的第一种siRNA药物Onpattro(治疗遗传性转甲状腺素蛋白淀粉样变性引起的周围神经病变),并于2020年分别批准了来自Moderna和辉瑞/BioNTech的两种新冠mRNA疫苗。值得注意的是,FDA批准这三种LNP配方均含有一种独特的可电离脂质,可电离脂类的结构设计空间巨大,其化学性质的微小变化可极大地影响生物功能。
Daniel Anderson/李博文团队等一直在探索高通量合成(HTS)策略,以加快设计和鉴定用于RNA递送的新型可电离脂质。只有少数化学反应与生物相容性溶剂中的脂质的热力学稳定性相兼容,可以在不使用催化剂或繁琐的保护/去保护步骤的情况下进行。其中两个值得注意的例子包括迈克尔加成反应,即胺与丙烯酸酯的1,4-加成,以及胺与环氧化物的开环反应。
然而,由于其二维结构,这些反应在可实际产生的可电离脂质的多样性和结构灵活性方面存在固有的限制。为了克服这一限制,Daniel Anderson/李博文团队于2023年3月在Nature Biotechnology 期刊发表论文【1】,设计了一种三组分反应(3CR)体系,其中硝基蓖麻油酸丙烯酸酯(NRA)连接剂与脂肪醇(脂质尾)偶联,然后与含有伯胺、仲胺或叔胺的头部基团连接。与传统的将胺类头部基团直接与脂质尾结合的双组分反应相比,3CR体系简化了繁琐的合成过程,增加了脂质结构多样性,快速生成了结构多样化的生物可降解电离脂质组合库。并在此基础上筛选和鉴定了RCB-4-8 LNP,这种新型LNP具有更好的生物可降解性和安全性,可重复气管内给药,首次成功利用LNP实现高效肺上皮细胞CRISPR基因编辑,为先天性肺部疾病的基因治疗开辟了新途径。

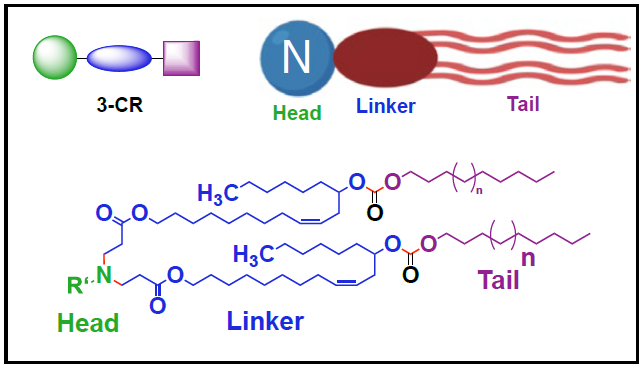
在这项发表于 Nature Materials 期刊的最新研究中,研究团队构建了基于四组分反应(4CR)的新型高通量合成(HTS)平台,使可电离脂质的设计和生成更加高效。从概念上讲,可电离脂质结构被划分为四个独立元件:胺头基、连接子、尾1和尾2,分别对应于4CR反应物中的胺、异氰、醛和羧酸。这种4CR方法是之前3CR方法的改进,允许增加维度和提高产率,从而促进了更多可电离脂质候选物的生成。
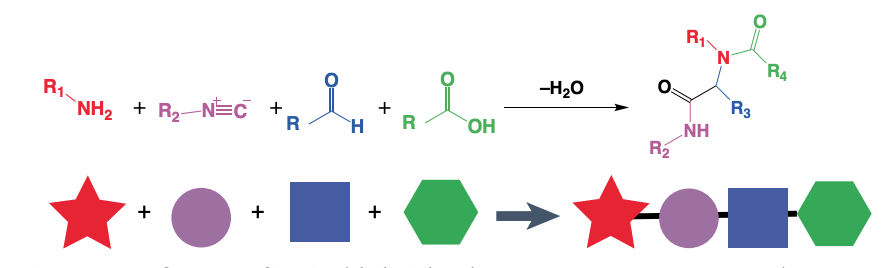
该研究介开发了一种将机器学习与前沿组合化学工具相结合的方法,以加速发现用于mRNA递送的有效可电离脂质。从一个简单的四组分反应(4CR)平台开始,研究团队创建了一个化学多样化的584个可电离脂质库。筛选了含有这些可电离脂质的脂质纳米颗粒(LNP)的mRNA转染效率,并将这些数据作为基础数据集用于训练各种机器学习模型。
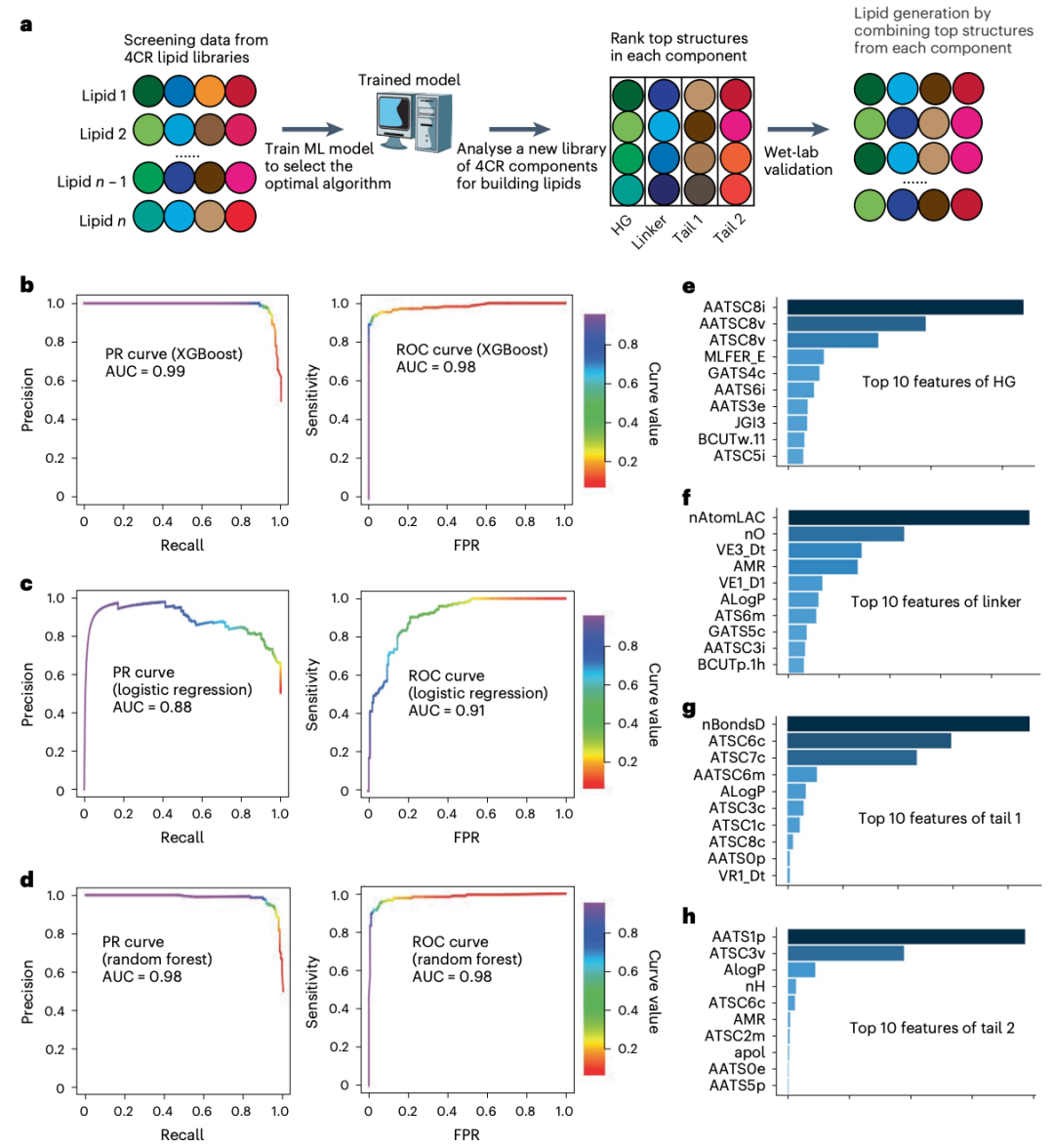
使用脂质筛选数据进行机器学习算法训练
然后,研究团队选择了其中性能最好的机器学习模型来探索一个包含40000种脂质的庞大的虚拟脂质库,进而合成并实验评估标记的前16个可电离脂质。研究团队发现了可电离脂质119-23,其在转染数种组织的肌肉和免疫细胞方面的效果优于已确立的基准脂质。
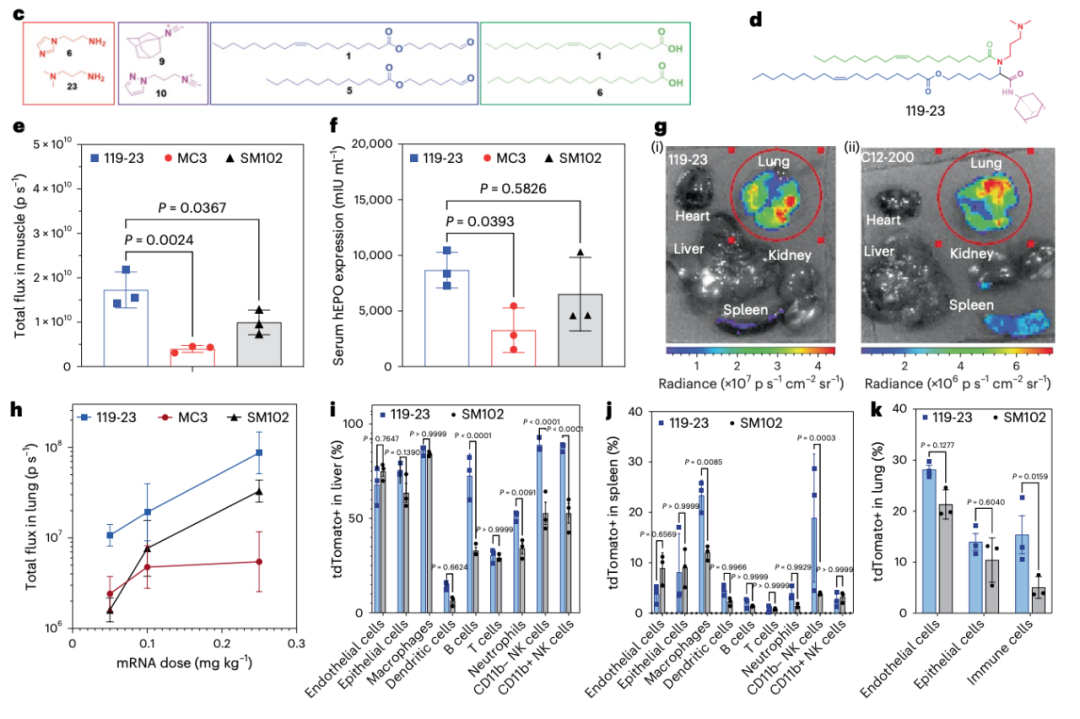
脂质库的机器学习辅助筛选和最佳表现脂质的验证
总的来说,该研究通过将机器学习和组合化学相结合,建立了一种加速发现和评估可电离脂质的方法,促进了用于精准递送mRNA的脂质纳米颗粒(LNP)的开发。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


