Cell Systems封面论文:曾坚阳团队提出蛋白质口袋结构表征新方法
来源:生物世界 2023-08-21 10:35
具体而言,对于输入的蛋白质结构,首要任务是确定其表面上anchor的位置。这些anchor位置是通过对蛋白质表面潜在结合配体的空间区域进行采样、过滤、聚类等操作后得到的。
曾坚阳课题组在 Cell Systems 期刊的 Focus on Protein Engineering and Design 专题中发表了题为:PocketAnchor: Learning structure-based pocket representations for protein-ligand interaction prediction 的封面论文,提出了蛋白质口袋结构表征新方法。


为了解决上述问题,曾坚阳团队设计了一种基于深度学习的蛋白质口袋结构表征新方法,命名为:PocketAnchor,用于捕捉蛋白质口袋的局部环境和空间特征。研究团队将“anchor”定义为伸入蛋白质空腔以及位于蛋白质表面附近的探针点,并设计了一种信息传递网络模型用以从氨基酸中各原子和蛋白质表面收集局部环境信息,以进一步更新anchor特征。这些anchor特征可应用于解决与蛋白质-配体相互作用预测相关的若干问题,如口袋检测和亲和力预测等(图1)。
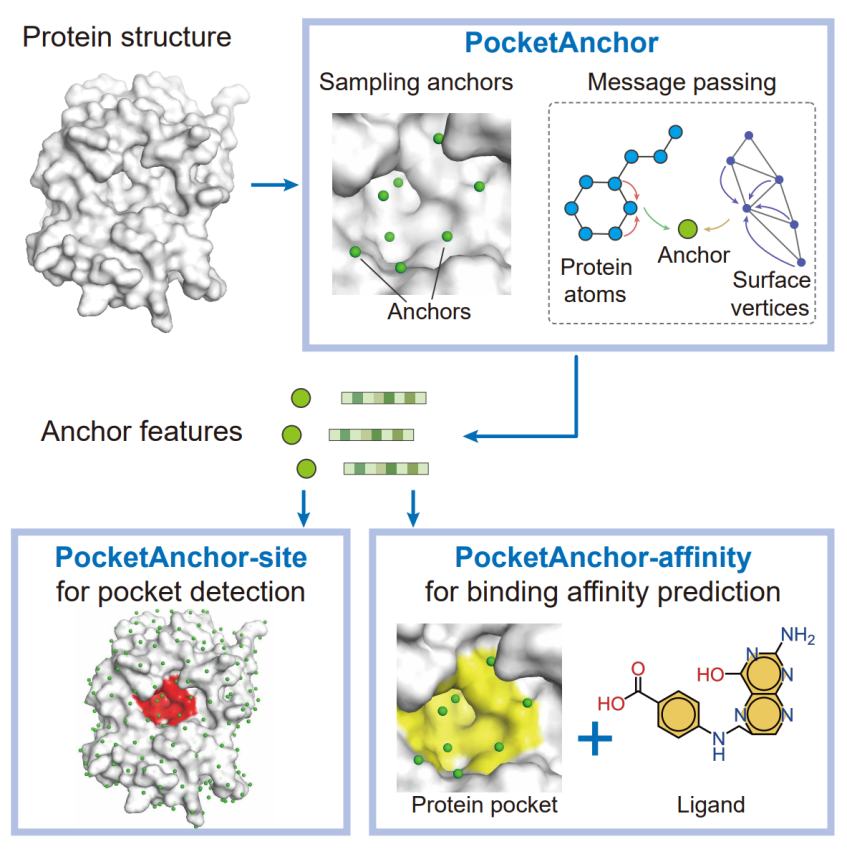
图1:PocketAnchor方法的概念及其应用流程
具体而言,对于输入的蛋白质结构,首要任务是确定其表面上anchor的位置。这些anchor位置是通过对蛋白质表面潜在结合配体的空间区域进行采样、过滤、聚类等操作后得到的。在这里,研究团队将anchor视为代表潜在可结合配体区域中的虚拟节点,并用于表示该位置的子口袋环境特征。相应地,该研究开发了PocketAnchor模块,以端到端的方式学习蛋白质子口袋结构的特征,并设计了一种新型信息聚合策略,将来自蛋白质表面和原子的邻居信息整合到蛋白质口袋的环境特征中(图2)。
此外,研究团队还设计了不同的预测模块,可以与PocketAnchor模块结合以解决各种下游任务。其中,PocketAnchor-site模型在口袋检测任务中取得了成功的应用,这表明基于anchor的蛋白质结构表征方法能够提供有效的结合口袋特征表示,从而改进相关预测任务的效果。
同时,通过集成小分子配体表征模块和亲和力预测模块设计的PocketAnchor-affinity 模型,可以成功应用于蛋白质-配体结合亲和力预测任务,尤其是对数据集中没有出现过的新蛋白质具有良好的泛化能力。
因此,该模型可以潜在地用于对新蛋白质靶标进行药物发现这一应用场景中。此外,通过对学习到的anchor特征进行分析发现,这些特征捕捉到了蛋白质子口袋区域的几何和化学特性,以及与小分子配体片段相互作用倾向性等重要信息,为蛋白质-配体相互作用的相关任务建模提供了有价值的特征信息。
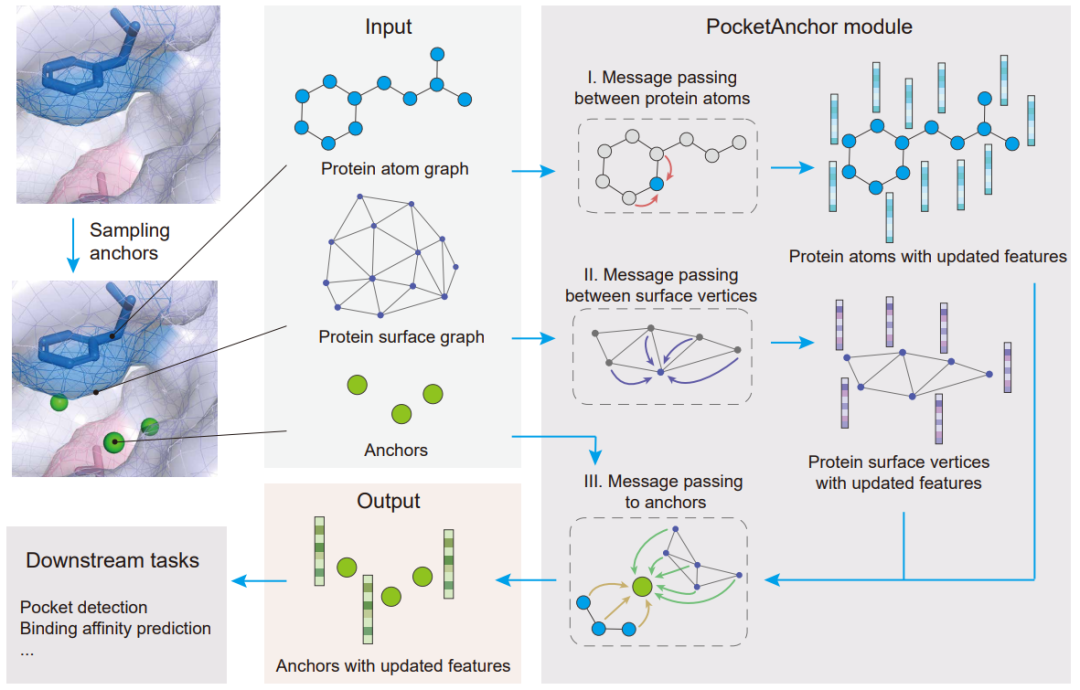
图2:PocketAnchor利用神经网络模型从蛋白质口袋局部提取信息并进行表征
清华大学交叉信息研究院博士毕业生李舒雅、田庭忠为论文共同第一作者。原清华大学交叉信息研究院副教授曾坚阳(现为西湖大学工学院教授)和助理研究员赵诞为论文共同通讯作者。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


