《自然·遗传学》:迄今最大规模外显子水平研究发现阿尔茨海默病新风险基因!
来源:奇点糕 2022-12-19 14:04
在这项研究中,Henne Holstege团队首次在外显子水平上利用罕见变异报道了新的AD风险基因——ATP8B4和ABCA1,并且还对以往GWAS报道的风险基因
阿尔茨海默病(AD)是痴呆症的主要原因,根据以往研究估计,AD的遗传度高达70%[1]。
一直以来,受限于基因分型技术,以往的大型队列只能采用SNP芯片结合基因型推断方法来对个体进行基因分型,因此无法有效检测在群体中突变频率较低的罕见变异,这也就导致GWAS发现的AD相关遗传变异都是次等位频率(MAF)>0.05的常见变异(大部分疾病/表型都受到同样影响)。
从进化的角度来讲,群体中的常见遗传变异一般不会对个体的生存繁殖产生较大的有害性,而极其有害的往往是罕见变异。因此GWAS发现的AD相关遗传变异往往影响力很小[2],仅仅关注这些变异是不够的,这只是造成AD具有70%遗传度的一部分原因,另一部分在遗传学研究中通常称之为“丢失的遗传度(Missing Heritability)”[3]。构成这部分遗传度的因素有很多,罕见变异是其中一种。
目前在二代测序技术的帮助下,罕见变异的检测精度已经得到显著提升,也是时候研究一下罕见变异所代表的AD遗传度了。
近日,由荷兰阿姆斯特丹自由大学的Henne Holstege、Marc Hulsman和法国巴斯德研究所的Jean-Charles Lambert以及法国鲁昂大学的Gaël Nicolas四位科学家领衔的研究团队在《自然·遗传学》期刊发表重要研究成果。
他们利用16036例AD患者和16522个健康人的外显子组测序数据,比较了AD患者和健康人之间基因的有害罕见变异负荷,发现TREM2、SORL1、ABCA7、ATP8B4和ABCA1在两者之间有显著差异。

论文截图
Henne Holstege团队分为两个阶段进行了这项研究,他们首先在12652例AD患者(包含4060例早发性AD患者[EOAD]和8592例晚发性AD患者[LOAD])和8693个健康人中识别具有显著差异的罕见变异负荷的基因,随后在3384例AD患者和7829个健康人中进行了验证。
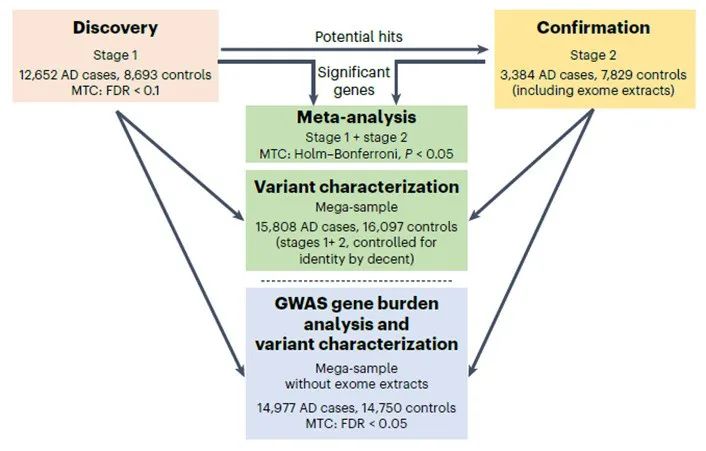
研究流程
在基因识别阶段,Henne Holstege团队选择了从外显子组测序数据中检测到的MAF<1%的罕见变异,并利用LOFTEE和REVEL(两个变异有害性注释数据库,REVEL分数介于0~100之间,越大表明变异是一个有害性更高的错义变异)对这些变异的有害性进行了注释,然后根据不同的有害性阈值得到了四组变异:LOF(由LOFTEE注释的导致基因功能丧失的变异,n=57543)、LOF+REVEL分数≥75(n=111755)、LOF+REVEL≥50(n=211665)、LOF+REVEL≥25(n=409733)。
由于这四组变异所涵盖的基因数量不同,因此Henne Holstege团队分别比较了四组不同数量的基因在AD患者和正常人之间的变异负荷,最终发现SORL1、TREM2、ABCA7、ATP8B4、ADAM10以及ABCA1的变异负荷在至少一组变异上有显著差异,表明这些基因与AD风险有关。
随后在验证阶段Henne Holstege团队对上述识别到的变异负荷差异基因进行了验证,这些基因仍然在至少一组变异上有显著差异的负荷。他们接着把两个阶段的数据整合到一起做了一次荟萃分析,发现SORL1、TREM2、ABCA7、ATP8B4以及ABCA1仍然在AD患者和正常人之间有显著差异的变异负荷,但ADAM10的变异负荷差异不再显著。
对此,研究人员推测原因可能是ADAM10上的罕见变异太少,统计力不足,在更大样本量的队列中应该可以验证ADAM10和AD的关联性。
上述基因中SORL1、TREM2、ABCA7、ABCA1以及ADAM10在以往的GWAS研究中也都被发现与AD相关[2],因此Henne Holstege团队想探究GWAS发现的AD相关基因是否会出现显著差异的罕见变异负荷。他们将两个阶段的样本合并成了一个更大的队列,并且比较了最近发表的AD相关GWAS研究报道的75个基因在AD患者和正常人之间的罕见变异负荷。
除了前面提到的基因,Henne Holstege团队还发现RIN3、CLU、ZCWPW1以及ACE罕见变异负荷在AD患者中更高,表明这些基因上的罕见变异与AD风险升高有关。
之后Henne Holstege团队还分析了位于上述基因上的罕见变异的特点,在大部分基因上,有害变异在EOAD患者中的频率更高,这很好理解,早期发病的主要因素就是遗传。并且具有最大影响力的LOF变异位于SORL1、ADAM10、CLU以及ZCWPW1上,携带这些变异的个体有最低的中位发病年龄,表明这些基因是AD病因的关键基因。
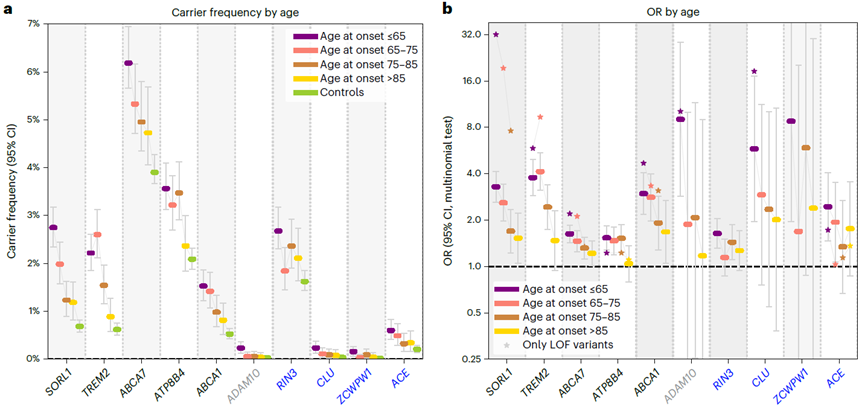
有害罕见变异在EOAD患者中频率更高(图a);基因的有害变异负荷OR在EOAD患者中更大(图b)
在整个合并队列中,9%的健康人在上述10个基因中的一个上发生了至少一个有害罕见变异,而在EOAD和LOAD患者中则分别为18%和14%。同时在EOAD患者中,2%的患者患病可以归因于SORL1、TREM2以及ABCA7上的有害罕见变异,1%归因于ATP8B4、ABCA1以及RIN3上的有害罕见变异。
总的来讲,在这项研究中,Henne Holstege团队首次在外显子水平上利用罕见变异报道了新的AD风险基因——ATP8B4和ABCA1,并且还对以往GWAS报道的风险基因RIN3、CLU、ZCWPW1以及ACE进行了罕见变异负荷分析,发现这些基因很可能是GWAS研究中的驱动基因,表明在GWAS无法衡量AD相关基因重要性的情况下,直接影响基因功能的罕见变异可以帮助我们找到更重要的基因。
参考文献:
[1] Gatz M, Reynolds CA, Fratiglioni L, et al. Role of genes and environments for explaining Alzheimer disease. Arch Gen Psychiatry. 2006;63(2):168-174. doi:10.1001/archpsyc.63.2.168
[2] Bellenguez C, Küçükali F, Jansen IE, et al. New insights into the genetic etiology of Alzheimer's disease and related dementias. Nat Genet. 2022;54(4):412-436. doi:10.1038/s41588-022-01024-z
[3] Génin E. Missing heritability of complex diseases: case solved?. Hum Genet. 2020;139(1):103-113. doi:10.1007/s00439-019-02034-4
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


