Cell Reports封面论文:从不确定中寻求确定,戴俊彪/蔡毅之团队实现串联重复的真核rDNA的深度精简与重塑
来源:生物世界 2024-03-03 14:30
该研究实现了串联重复的rDNA阵列的高效定制合成,并引入人工重排系统,成功将单倍体酵母菌株中rDNA拷贝数精简至约8个拷贝,是目前研究发现的可支持生存的最低rDNA拷贝数。
近日,中国科学院深圳先进技术研究院戴俊彪团队和曼彻斯特大学蔡毅之团队在 Cell Reports 期刊发表了题为:High plasticity of ribosomal DNA organization in budding yeast 的研究论文,并被选为2024年2月27日同期的封面文章。
该研究开发了高效的重复阵列构建方法CAT,并将人工重排系统引入串联重复的rDNA阵列中,成功实现了rDNA区域的深度精简,发现8个拷贝的rDNA序列即可维持菌株生存;同时,首次构建了包含三个阵列的酵母模型,探索了rDNA阵列数目的影响。
该研究揭示了酵母rDNA区域组织形式的高度可塑性,也是戴俊彪团队近期取得的Sc3.0计划的又一重要成果。

为了将人工控制系统引入rDNA区域,研究人员开发了一种高效的阵列构建方法CAT,能够在基因组任意位置快速构建具有定制序列的合成型rDNA阵列。利用该方法,研究人员成功地在3号(短染色体)或15号(长染色体)染色体上构建了异位rDNA阵列。随后,利用合成型rDNA阵列中的引入的loxPsym序列和诱导表达的Cre重组酶,成功实现了3号染色体上短rDNA阵列的深度精简,发现8个拷贝的rDNA单元即可支持酿酒酵母的生存。
高等生物基因组中通常含有多个rDNA阵列,分布在不同染色体上。为了模拟高等生物rDNA的组织形式,探究rDNA阵列数目的影响,研究人员结合CAT与减数分裂,成功构建了多个两阵列菌株和三阵列菌株。结果显示,额外引入的rDNA阵列虽然显著改变了基因组3D构象,但对单核仁结构的形成、菌株的正常生长和基因组表达水平没有显著的破坏性影响,充分显示了酵母基因组对于rDNA阵列组织形式的包容性。
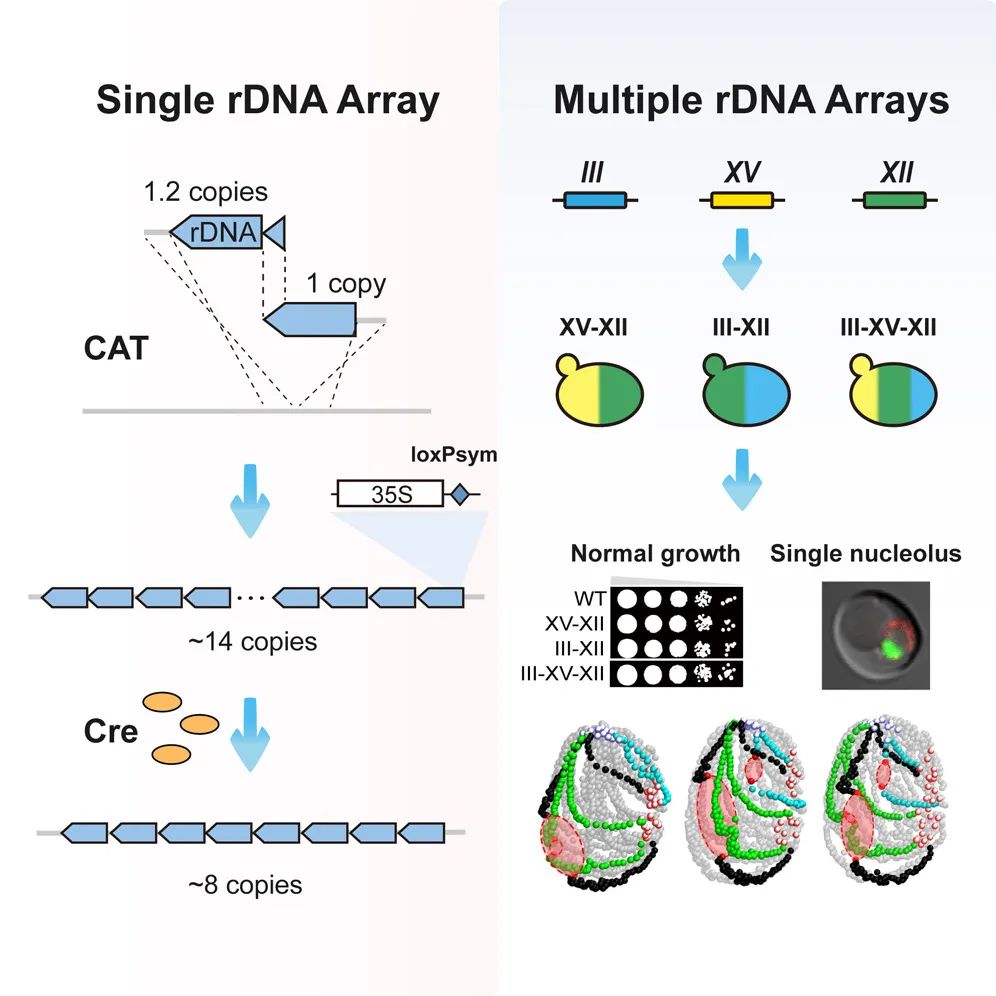
综上所述,该研究实现了串联重复的rDNA阵列的高效定制合成,并引入人工重排系统,成功将单倍体酵母菌株中rDNA拷贝数精简至约8个拷贝,是目前研究发现的可支持生存的最低rDNA拷贝数;同时,该研究构建了多rDNA阵列的酵母模型,解析了额外rDNA阵列的影响,揭示了酵母细胞对多阵列组织形式的包容性。本研究利用合成基因组学理念,对于高度重复的rDNA阵列进行了深度精简和合成重塑,不仅拓展了现有理论认知,为后续最小酵母基因组和稳健的简化酵母底盘的构建奠定理论基础,也为后续rDNA的调控解析提供了重要的研究工具。
中国科学院深圳先进技术研究院副研究员姜双英、研究助理蔡泽霖、深圳华大生命科学研究院王云副研究员和先进院研究助理曾诚为论文共同第一作者。戴俊彪研究员、姜双英副研究员与蔡毅之教授为论文共同通讯作者。该研究获得国家重点研发计划、国家自然科学基金、广东省合成基因组学重点实验室、广东省自然科学基金、中国科学院国际大科学计划、深圳市杰出人才培养基金、英国皇家学会牛顿高级学者基金等多个项目的支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


