Nature Methods | 解码基因调控新机制:系统性发现人类转录组中的RNA结构开关
来源:生物探索 2024-07-20 10:20
该研究展示了RNA结构开关在基因表达调控中的重要性,并强调了开发新的RNA开关鉴定方法的必要性。未来,SwitchSeeker有望被应用于更广泛的转录组研究中,揭示更多关于RNA结构和功能的奥秘。
RNA结构开关(RNA structural switches)是基因表达的重要调控因素,广泛存在于细菌中。然而,在真核生物中,RNA结构开关的研究和鉴定却相对有限。在细菌中,一些RNA开关被称为核糖开关(riboswitches),它们通过结合小分子配体诱导RNA构象变化,从而控制基因表达。尽管植物和真菌中已鉴定出一些硫胺素焦磷酸感应核糖开关,但在人类中已知的RNA开关仅有两个:一个是依赖于蛋白质的血管内皮生长因子A(VEGFA)RNA开关,另一个是基于m6A修饰的RNA开关。因此,RNA开关在高等真核生物基因表达中的总体影响仍不清楚。
7月16日Nature Methods的研究报道“A systematic search for RNA structural switches across the human transcriptome”介绍了SwitchSeeker,一种综合计算和实验方法,用于系统鉴定功能性RNA结构开关。研究人员将SwitchSeeker应用于人类转录组,并识别出245个潜在的RNA开关。为了验证该方法,他们在RORC(RAR-related orphan receptor C)转录本的3'非翻译区(3' UTR)中表征了一个以前未知的RNA开关。使用体内二甲基硫酸(DMS)突变谱测序(DMS-MaPseq)结合低温电子显微镜(cryo-EM)确认了其存在,并发现该RNA开关在两种不同的结构构象中表现出不同的调控功能。此外,利用基因组范围的CRISPR筛选,鉴定出通过该RNA结构开关调控基因表达的反式因子(trans factors),并发现无义介导的mRNA降解(nonsense-mediated mRNA decay, NMD)以构象特异性方式作用于该元素。
SwitchSeeker提供了一种无偏见、实验驱动的方法,用于发现塑造真核生物基因表达图景的RNA结构开关。这一研究不仅扩展了我们对RNA开关在基因调控中的理解,还为未来全面表征不同细胞类型和生物体中的RNA开关奠定了基础。
这项研究展示了RNA结构开关在基因表达调控中的重要性,并强调了开发新的RNA开关鉴定方法的必要性。SwitchSeeker作为一种迭代的预测和验证平台,通过结合计算模型和高通量实验技术,能够有效地鉴定并表征RNA开关。这一框架的应用不仅限于3' UTR,还可以扩展到其他类型的RNA开关研究中,为揭示真核生物RNA调控机制提供了新的视角。

基因表达的调控在所有生命王国中都是至关重要的过程。自从发现核糖体(ribosome)和RNA聚合酶(RNA polymerase)等分子机器以来,研究人员逐渐认识到RNA在基因调控中的核心作用。特别是一些古老的RNA调控机制,如核酶(ribozymes)和RNA结构开关,展示了RNA分子在细胞内复杂的功能。
在细菌中,核糖开关(riboswitches)通过结合小分子配体(ligands),诱导RNA构象变化,从而控制基因表达。例如,核糖开关可以感知环境中的代谢物浓度,并通过改变其二级结构(secondary structure)调控相关基因的转录或翻译。然而,在真核生物中,尽管植物和真菌中已鉴定出一些硫胺素焦磷酸(thiamine pyrophosphate)感应核糖开关,但在人类中已知的RNA开关仅有两个:一个是依赖于蛋白质的血管内皮生长因子A(VEGFA)RNA开关,另一个是基于m6A修饰的RNA开关。因此,RNA开关在高等真核生物基因表达中的总体影响仍不清楚。
SwitchSeeker:系统性鉴定RNA结构开关的方法
为了填补这一知识空白,研究团队开发了SwitchSeeker,一种综合计算和实验的方法,用于系统鉴定功能性RNA结构开关。SwitchSeeker结合了计算模型和高通量实验技术,通过迭代的预测和验证过程,有效地鉴定并表征RNA开关。
首先,研究团队开发了一种名为SwitchFinder的计算模型,该模型能够从头(de novo)预测RNA开关。SwitchFinder通过生成RNA序列的二级结构及其对应的能量景观(energy landscape),识别具有RNA开关特征的序列。这些特征包括两个局部能量最小值(local minima)和一个相对较小的能量屏障(energy barrier)。通过这种方式,SwitchFinder能够在不依赖已知序列模式的情况下,识别出新的RNA开关。
在验证SwitchFinder的性能时,研究团队将其应用于一组已知的RNA开关家族,并观察到该模型在这些家族中的预测准确性比现有的方法更高。接下来,他们将SwitchFinder与高通量实验技术相结合,创建了一个端到端的预测和验证平台,命名为SwitchSeeker。
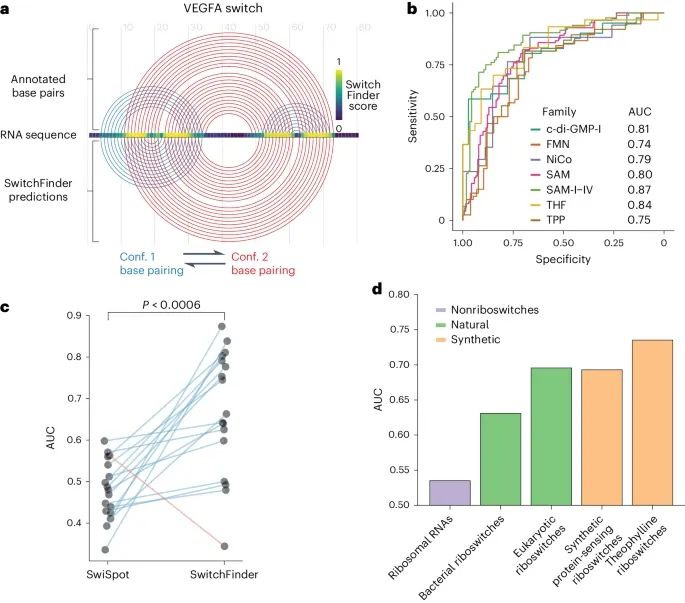
SwitchFinder在识别人类基因组中的候选RNA开关时的结果(Credit: Nature Methods)
图a. SwitchFinder如何在VEGFA(血管内皮生长因子A)mRNA序列中定位RNA开关。示例显示了RNA开关在序列中的具体位置。
图b. SwitchFinder对常见Rfam家族RNA开关预测的ROC曲线。SwitchFinder应用于真实序列和乱序对照序列的混合物,ROC曲线衡量其正确选择真实序列的能力。结果表明,SwitchFinder在不同RNA开关家族中的预测表现良好。
图c. 比较了SwitchFinder和SwiSpot两种模型在Rfam家族中的RNA开关预测性能。每个点代表一个Rfam家族,结果显示SwitchFinder在大多数RNA开关家族中的预测准确性优于SwiSpot。
图d. 展示了SwitchFinder在各种天然和合成核糖开关(riboswitches)群体中的预测表现,包括细菌、真核和合成的核糖开关。结果表明,SwitchFinder在识别这些不同类型的核糖开关时表现出色。
在人类转录组中的应用
研究团队将SwitchSeeker应用于人类转录组,以鉴定潜在的RNA开关。首先,他们对人类转录组的3'非翻译区(3' UTR)进行了分析,选择了长度不超过186个核苷酸的3,750个候选开关元素。为了进一步筛选出在细胞中既功能性又结构上双稳态(bi-stable)的RNA开关,他们分别进行了两项高通量体内筛选实验:结构筛选和功能筛选。
结构筛选
在结构筛选中,研究团队利用DMS-MaPseq技术对表达候选RNA开关文库的HEK293细胞进行了分析。DMS-MaPseq数据中的单核苷酸可及性(accessibility)反映了多个RNA分子在Gibbs自由能景观中的不同能量最小值。如果一种构象占主导地位,它将在DMS-MaPseq反应性谱中表现出来;如果多种构象共存,它们将共同贡献于反应性谱。通过这种方法,SwitchSeeker能够识别出在体内共存于两种构象之间的RNA开关。
功能筛选
在功能筛选中,研究团队实施了大规模并行报告基因分析(MPRA),在HEK293细胞中功能性地探测RNA开关。他们将3,750个候选RNA开关序列或相应的乱序对照序列克隆到一个双荧光报告基因(eGFP-mCherry)中,使用eGFP荧光测量候选RNA开关对基因表达的影响,同时使用未受影响的mCherry荧光作为内源对照。通过流式细胞仪对按eGFP
表达比例分类的细胞进行排序和测序,研究团队发现536个候选RNA开关显著下调了eGFP的表达,538个候选RNA开关显著上调了eGFP的表达。
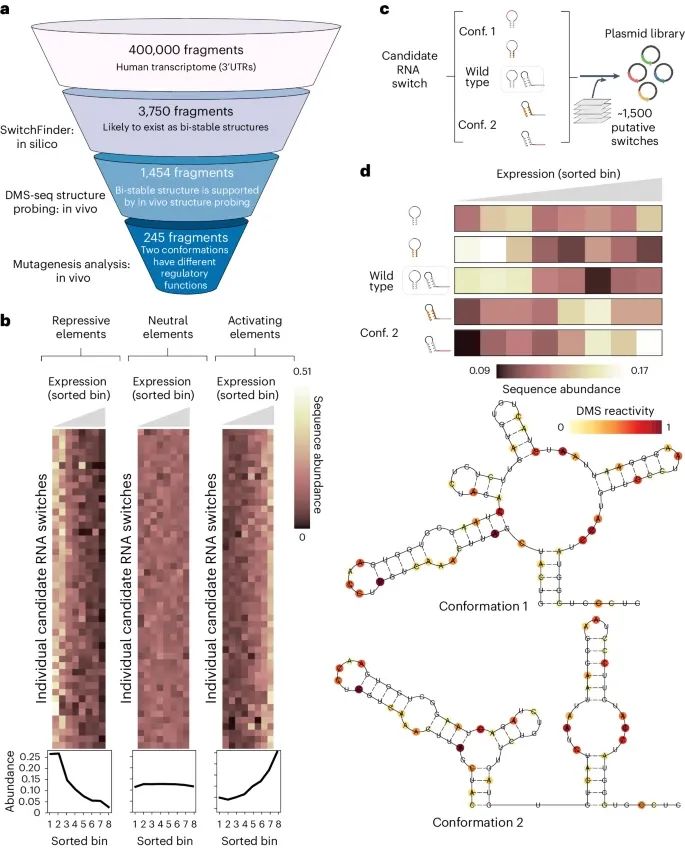
大规模并行报告基因分析(MPRA)在捕捉候选RNA开关不同构象之间功能差异时的结果(Credit: Nature Methods)
图a. 概述了SwitchSeeker平台在识别人类转录组中3'非翻译区(3'UTR)候选RNA开关的流程图。从人类转录组中选择出约3,750个候选RNA开关,通过结构筛选和功能筛选进一步筛选出1,454个候选RNA开关,并最终确认245个具有功能差异的双稳态结构开关。
图b. 展示了功能筛选的结果。每一行代表一个候选RNA开关,每一列代表通过报告基因表达(eGFP荧光,相对于mCherry荧光)定义的单个分类。在排序的八个分类中,展示了下调、中性和上调基因表达的候选RNA开关的相对丰度。
图c. 描述了大规模平行突变分析(MPRA)设置。对于每个候选RNA开关,设计了四个突变序列变体,其中两个锁定在构象1,另外两个锁定在构象2。通过这种方式,生成了一个序列库,每个候选RNA开关由四个突变序列变体和参考序列表示。
图d. 展示了通过MPRA确定的高置信度候选RNA开关的具体示例。底部展示了SwitchSeeker预测的两种备选构象,结合了RNA二级结构探测数据。顶部展示了候选RNA开关在不同构象锁定时对报告基因表达的影响。每一行代表一个锁定RNA开关在特定构象的序列变体,每一列代表基因表达定义的一个分类。
关键发现:RORC RNA开关
为了验证SwitchSeeker的预测,研究团队选择了位于RORC(RAR-related orphan receptor C)转录本3' UTR中的一个开关进行深入分析。通过DMS-MaPseq结构探测和单颗粒低温电子显微镜(cryo-EM),他们确认了该RNA开关在两种不同的分子构象中存在,并通过基因组范围的CRISPR干扰(CRISPRi)筛选发现其中一种构象通过激活非经典的无义介导mRNA降解(NMD)途径减少了RORC基因的表达。
研究团队设计了突变-恢复(mutation-rescue)实验,以确认RORC RNA开关的两种构象在基因表达中的不同作用。他们发现,一些突变可以稳定其中一种构象,而另一些突变则可以稳定另一种构象。例如,位于Box 1、Box 2和Box 3的突变对RORC RNA开关的结构和功能有显著影响。进一步的实验表明,这些突变在体外和体内都展示了类似的构象变化,验证了该RNA开关在不同环境中的稳定性。
通过SwitchSeeker的应用,研究团队最终报告了245个高置信度的功能性RNA结构开关。这一研究不仅扩展了我们对RNA开关在基因调控中的理解,还为未来全面表征不同细胞类型和生物体中的RNA开关奠定了基础。
SwitchSeeker作为一种迭代的预测和验证平台,通过结合计算模型和高通量实验技术,能够有效地鉴定并表征RNA开关。这一框架的应用不仅限于3' UTR,还可以扩展到其他类型的RNA开关研究中,为揭示真核生物RNA调控机制提供了新的视角。
该研究展示了RNA结构开关在基因表达调控中的重要性,并强调了开发新的RNA开关鉴定方法的必要性。未来,SwitchSeeker有望被应用于更广泛的转录组研究中,揭示更多关于RNA结构和功能的奥秘。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


