研究揭示真核生物uORF的分布特征和演化规律
来源:北京大学 2021-02-22 17:02
近日,北京大学生命科学学院陆剑课题组在Nature Communications发表题为“Determinants of genome-wide distribution and evolution of uORFs in eukaryotes”的论文,系统解析了真核生物uORF在不同物种以及不同基因之间的差异分布形成的驱动力,以及u
近日,北京大学生命科学学院陆剑课题组在Nature Communications发表题为“Determinants of genome-wide distribution and evolution of uORFs in eukaryotes”的论文,系统解析了真核生物uORF在不同物种以及不同基因之间的差异分布形成的驱动力,以及uORF的序列演化特征。
传统观点认为,细胞中的蛋白质由核糖体翻译mRNA的编码区(CDS)产生。近年来,人们通过捕获核糖体正在翻译的RNA片段进行高通量测序发现,核糖体还可以翻译mRNA中CDS上游或下游的非翻译区(UTR)以及一些非编码RNA。许多研究表明,这些由UTR和非编码RNA上翻译事件构成的非经典翻译组,在生物体的发育调控、胁迫响应和免疫应答过程以及肿瘤等人类疾病中发挥着重要功能。作为非经典翻译组的主要组成部分,uORF是起始密码子位于5' UTR中的开放阅读框。陆剑课题组对uORF的功能和演化进行了长期的研究,首次发现uORF的演化受到正选择驱动[1],并解析了uORF在果蝇发育过程中的调控作用[1],揭示了uORF和miRNA如何组合使用调控基因表达[2]。在此基础上,课题组受邀发表综述文章,总结并展望这一领域的研究[3]。
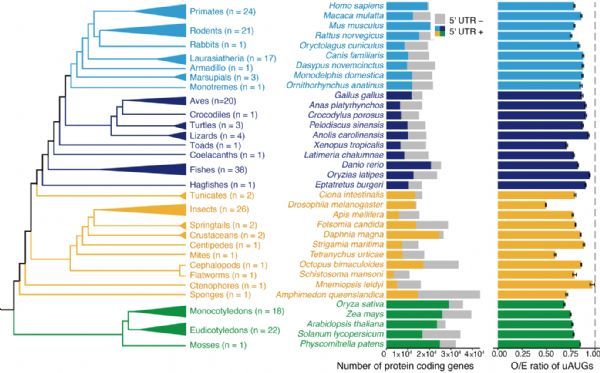
虽然uORF在真核生物中普遍存在,uORF在不同物种以及不同基因中的分布规律还缺乏系统的研究,有多少uORF的翻译产物可能编码有功能的蛋白质也尚不清楚。为了解决这些基本问题,课题组的研究人员分析了478个物种的高质量基因组注释。通过对这些物种中鉴定到的大约1700万个uORF的分析,研究人员发现真核生物中的uORF整体上受到纯化选择(216种动植物中的概况见下图)。比较分析表明,不同物种间uORF的相对多少主要由有效种群大小决定,有效种群越小的物种中,uORF受到的纯化选择较弱,实际观测到的uORF跟预期的uORF数目更接近。同一个物种的不同基因携带uORF数目呈现很大差异,他们发现,一个基因携带uORF数目的多少主要受基因表达量影响,表达越高的基因携带的uORF数目越少。另一方面,物种中新固定下来的uORF也受到正选择驱动,而且这一趋势在高表达的基因中更明显。
研究中涉及到的216种多细胞动植物的概况。左图为这些物种的系统发生关系;中间为不同进化支上35个代表性物种中蛋白质编码基因的5' UTR注释情况;右图为代表性物种中实际观察到的uORF起始密码子uAUG的数目跟预期的数目的比值。
为了区分uORF的主要功能是调控作用还是编码蛋白质,研究人员对uORF的起始密码子和开放阅读框的序列进行了比较。他们发现,uORF的起始密码子远比其阅读框本身更加保守,uORF的开放阅读框中非同义突变和同义突变的比例也更加符合中性演化。基于此,研究人员得出uORF的主要功能是调控作用而不是编码小肽。他们进一步推测可能编码有功能的蛋白质的uORF所占的比例不到1%,这也跟质谱数据中很难检测到uORF编码的蛋白质的结果相符。
综上所述,该研究揭示了有效种群大小和基因表达量如何跟自然选择共同作用,决定uORF在不同物种和不同基因中的分布。通过对uORF序列演化和质谱数据的分析,研究发现uORF的主要功能是作为顺式调控元件,而不是编码蛋白质。此外,该研究还发现,相对于以AUG作为起始密码子的经典uORF,以CUG、GUG等密码子起始的非经典uORF更不保守,对CDS翻译的抑制作用也更弱。这些发现为我们理解uORF的演化和功能提供了很多新的见解。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。