Nature子刊:左二伟团队利用AlphaFold2开发出新型碱基编辑器
来源:生物世界 2024-06-05 09:19
研究团队使用AlphaFold2预测了1483个胞苷脱氨酶的三维结构,根据结构相似性对这些脱氨酶进行聚类,并从聚类中选择具有代表性的272个脱氨酶进行碱基编辑活性检测。
中国农业科学院深圳农业基因组研究所左二伟团队在 Nature Biomedical Engineering 期刊发表了题为:Structure-guided discovery of highly efficient cytidine deaminases with sequence-context independence 的研究论文。
该研究创新性地运用AlphaFold2介导的蛋白结构预测和结构相似性聚类方法来挖掘新型胞嘧啶脱氨酶,并开发了高效、无序列偏好性的胞嘧啶碱基编辑工具。

胞嘧啶脱氨酶,如载脂蛋白B mRNA催化编辑蛋白酶(APOBEC)和激活诱导的胞嘧啶脱氨基化酶(AID),是催化基因组DNA中C碱基脱氨为U碱基并诱导单碱基替换的基因编辑酶。研究人员利用大鼠APOBEC1(rAPOBEC1)、人APOBEC3A(hAPOBEC3A)和人AID(hAID)等构建了多种胞嘧啶碱基编辑器(CBE)。
虽然通过各种工程进化手段可以优化脱氨酶的编辑性能,但受制于脱氨酶的原有序列基础,优化后的脱氨酶在某些方面的编辑特性仍存在缺陷。比如,不能兼顾高编辑效率和低脱靶效应,诱导产生较多插入/缺失突变(indels),C-to-G的编辑效率和编辑纯度较低等。更重要的是,现有的编辑器都具有明显的序列偏好性,目前缺乏可以对任意NC基序(AC/TC/CC/GC)无完全偏好的胞嘧啶脱氨酶,极大的限制了CBE在某些重要位点的编辑效率。因此,发掘具有新功能特性的胞嘧啶脱氨酶对于升级和扩展CBE的编辑能力具有重要意义。
在这项研究中,研究团队使用AlphaFold2预测了1483个胞苷脱氨酶的三维结构,根据结构相似性对这些脱氨酶进行聚类,并从聚类中选择具有代表性的272个脱氨酶进行碱基编辑活性检测。通过这种方法,研究团队筛选到了多种具有鲜明特征性的脱氨酶。在HEK293T细胞中的实验结果显示,这些代表性的脱氨酶表现出多种独特的编辑特征。比如,有的具有超高的C-to-T编辑活性,有的具有高编辑特异性(编辑效率和脱靶效应的比值),有的具有多样性的编辑窗口。最有意思的是,研究团队发现了几种对编辑位点序列无偏好性的胞嘧啶脱氨酶,它们对靶位点的不同基序(AC/GC/TC/CC)表现出无选择偏好性编辑。此外,通过蛋白/DNA互作模型,研究团队在候选脱氨酶上预测了多个影响单链DNA结合能力的氨基酸残基位点,对这些位点的突变进一步降低了脱氨酶的脱靶效应。
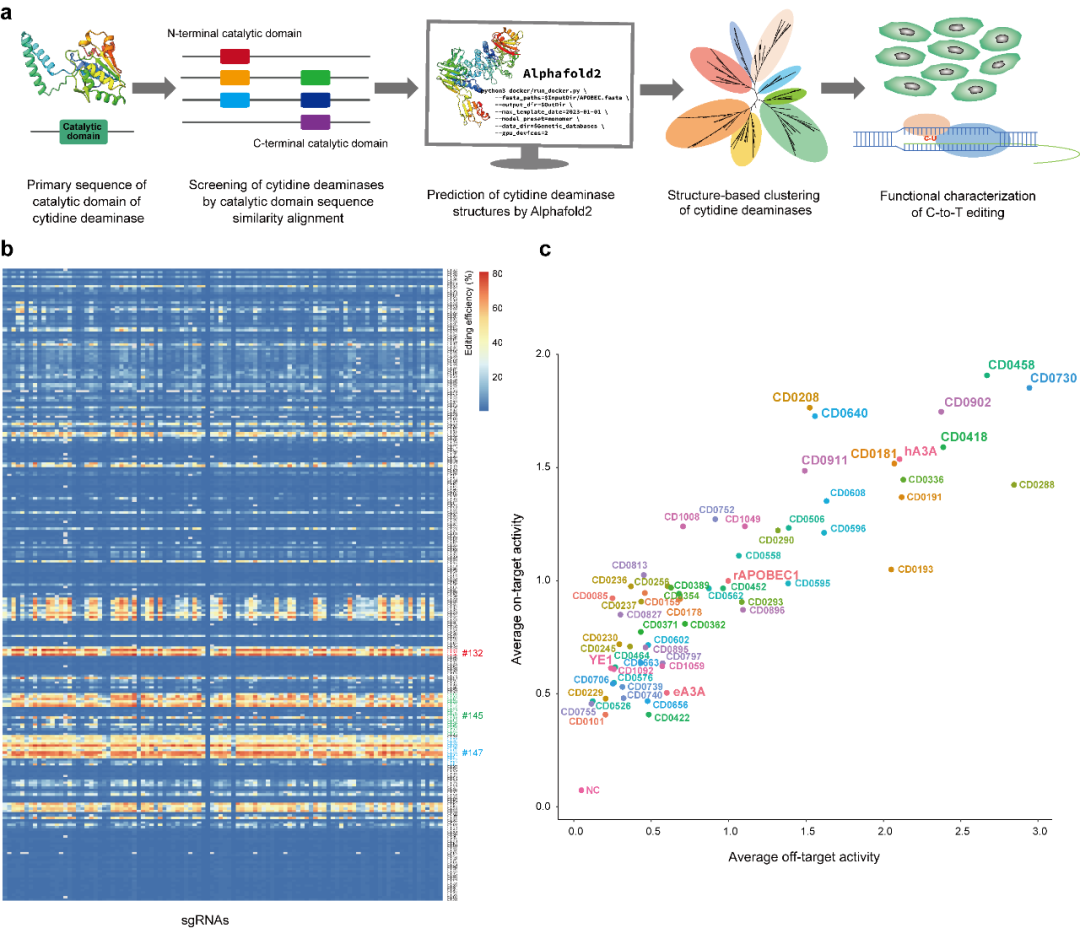
基于AlphaFold2蛋白结构预测和结构相似性聚类的方法深度挖掘新型胞嘧啶脱氨酶。a,胞嘧啶脱氨酶挖掘工作流程。b,272个代表性胞嘧啶脱氨酶在sgRNA文库中的平均编辑效率。c,利用R-loop实验来评估候选脱氨酶的off-target效应。
研究团队还在多种哺乳动物细胞(人HEK293T、小鼠N2A、小鼠mESCs和猪PK-15)基因组的内源性位点上检测了胞嘧啶碱基编辑器(CBE)的编辑效果。结果显示,新开发的CBE可以对这些哺乳动物细胞的单拷贝或多拷贝基因进行高效的C-to-T编辑,并诱导产生终止密码子。此外,优化后的胞嘧啶脱氨酶还可与多种Cas蛋白(nSpCas9、nSpCas9-NG、nSaCas9和dCpf1)兼容,极大的提高了CBE的靶向范围和编辑产物纯度。
这项研究显示出基于AI的蛋白结构预测工具在挖掘和筛选新功能蛋白的方面的巨大潜力,并提供了高效率、适用广泛的候选胞嘧啶脱氨酶用于构建CBE工具,有力推进CBE的生物医学和农业育种上等方面的应用。
中国农业科学院深圳农业基因组研究所左二伟研究组博士后徐奎、冯虎、张海航、贺晨飞和博士生康会芳为论文共同第一作者,左二伟研究员为论文通讯作者。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


