功能蛋白自然多样性挖掘方法方面取得进展
来源:农业资源中心 2022-09-26 10:21
中国科学院遗传与发育生物学研究所农业资源研究中心研究员李小方团队,通过基于序列的宏基因组学和功能基因组学技术,从全球不同生境挖掘到大量新颖铜抗性copA功能基因。
蛋白质自然多样性的概念在进化生物学的研究以及蛋白质工程改造中起着至关重要的作用。例如,研究者广泛利用DNA定向RNA聚合酶β亚基(RpoB)和固氮酶铁蛋白(NifH)的自然多样性来研究微生物的系统发育,特别是那些不可以人工培养的微生物。在这项研究中,研究团队开发了基于序列的功能宏基因组学流程,通过结合宏基因组组装、本地BLAST、进化踪迹分析、化学合成以及传统功能基因组学技术,挖掘了全球微生物宏基因组数据中的铜抗性基因copA的多样性。此研究共收集了来自世界各地的87个宏基因组数据,从数亿蛋白序列中筛查到近100000条候选序列,进一步手工筛查获得175个高置信新颖copA基因序列。系统发育分析表明,几乎所有预测到的CopA蛋白都与已知CopA蛋白有近缘关系,其中55个序列来自完全未知的物种。通过化学合成10个预测copA基因和3个已知copA基因并转化铜敏感大肠杆菌,研究发现其中5个预测copA基因不仅提高了宿主的铜抗性,同时促进了宿主对铜的吸收。该研究不仅扩展了人们对环境微生物CopA功能多样性的认识,也为基因工程修复生物材料的开发提供了宝贵的基因资源。
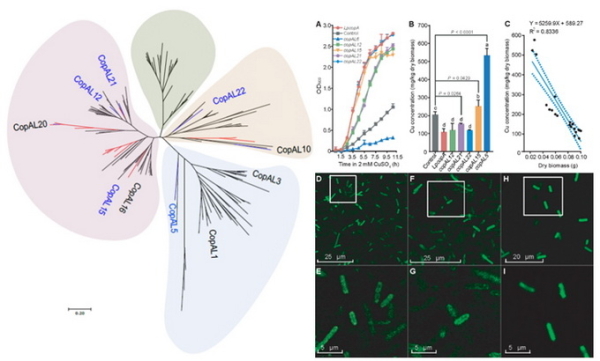
获得的新颖铜抗性蛋白序列多属于全新的世系,代表性蛋白表现功能活性并正确定位
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


