Cell:开发出将多种单细胞数据集结合在一起的新工具,有助确定细胞类型
来源:本站原创 2019-06-17 12:51
2019年6月17日讯/生物谷BIOON/---单细胞研究揭示了可能在其他分析中被忽略的细胞细节。生物学家当前使用一系列方法来收集不同组织和物种的单细胞数据。一些科学家可能使用基于组织的原位方法来研究小鼠神经元中的DNA甲基化,而另一些科学家可能使用液滴方法来探究人神经元中的RNA表达。这些研究创建了许多独特的不易结合在一起的数据集。但是,将这些数据结合在一起来比较多个个体、组织和物种的相似细胞的
2019年6月17日讯/生物谷BIOON/---单细胞研究揭示了可能在其他分析中被忽略的细胞细节。生物学家当前使用一系列方法来收集不同组织和物种的单细胞数据。一些科学家可能使用基于组织的原位方法来研究小鼠神经元中的DNA甲基化,而另一些科学家可能使用液滴方法来探究人神经元中的RNA表达。
这些研究创建了许多独特的不易结合在一起的数据集。但是,将这些数据结合在一起来比较多个个体、组织和物种的相似细胞的所有方面对于真正理解细胞类型的不同作用是必要的。
在一项新的研究中,美国斯坦利精神病学研究中心准会员Evan Macosko、访问学者Joshua Welch及其团队开发出一种将这些数据集结合在一起的新工具。这种称为基因组实验关系关联推理(Linked Inference of Genomic Experimental Relationships, LIGER)的工具将不同的单细胞数据集整合在一起;将来自不同受试者、物种或分子衡量指标的类似细胞进行分组;针对不同组彼此之间的关系构建图谱。相关研究结果发表在2019年6月13日的Cell期刊上,论文标题为“Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity”。
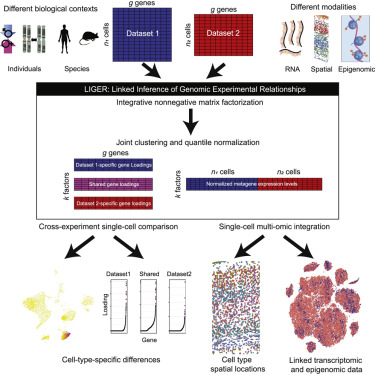
其他常见的单细胞数据集集成方法消除了不同数据集之间的差异。但是,这些差异在生物学上很重要,比如,在比较健康的和受疾病影响的细胞时。LIGER既可以保留差异,也可以识别相似之处,从而实现更丰富的分析。
当这些研究人员在LIGER中加载单细胞数据集时,这种工具使用一种称为“整合性非负矩阵分解(integrative non-negative matrix factorization)”的统计方法,基于两个定义的特征---一组对细胞原始数据集是独特的因子和一组在不同数据集之间都共有的因子(通常对应于生物学上有意义的信号)---来识别和聚类细胞。这种方法使得人们能够更清晰地查看来自不同来源的细胞之间的共有特征和独特特征。
这些研究人员以四种方式测试了LIGER。首先,他们使用这种工具来定量确定不同大脑区域和不同动物之间的小鼠脑细胞类型差异。其次,他们使用LIGER通过比较来自人类和小鼠相同大脑区域的脑细胞来探究不同物种之间的相似性和差异。第三,他们使用这种工具研究单细胞RNA测序(scRNA-seq)和原位RNA测序分析的小鼠脑细胞的空间关系,从而将受测试的细胞与它们的原始大脑位置相匹配在一起。第四,这些研究人员使用LIGER通过将scRNA-seq和DNA甲基化谱合并在一起来研究小鼠脑细胞的表观基因组特征。(生物谷 Bioon.com)
参考资料:
Joshua D. Welch et al. Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity. Cell, 2019, doi:10.1016/j.cell.2019.05.006.
这些研究创建了许多独特的不易结合在一起的数据集。但是,将这些数据结合在一起来比较多个个体、组织和物种的相似细胞的所有方面对于真正理解细胞类型的不同作用是必要的。
在一项新的研究中,美国斯坦利精神病学研究中心准会员Evan Macosko、访问学者Joshua Welch及其团队开发出一种将这些数据集结合在一起的新工具。这种称为基因组实验关系关联推理(Linked Inference of Genomic Experimental Relationships, LIGER)的工具将不同的单细胞数据集整合在一起;将来自不同受试者、物种或分子衡量指标的类似细胞进行分组;针对不同组彼此之间的关系构建图谱。相关研究结果发表在2019年6月13日的Cell期刊上,论文标题为“Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity”。

图片来自Cell, 2019, doi:10.1016/j.cell.2019.05.006。
其他常见的单细胞数据集集成方法消除了不同数据集之间的差异。但是,这些差异在生物学上很重要,比如,在比较健康的和受疾病影响的细胞时。LIGER既可以保留差异,也可以识别相似之处,从而实现更丰富的分析。
当这些研究人员在LIGER中加载单细胞数据集时,这种工具使用一种称为“整合性非负矩阵分解(integrative non-negative matrix factorization)”的统计方法,基于两个定义的特征---一组对细胞原始数据集是独特的因子和一组在不同数据集之间都共有的因子(通常对应于生物学上有意义的信号)---来识别和聚类细胞。这种方法使得人们能够更清晰地查看来自不同来源的细胞之间的共有特征和独特特征。
这些研究人员以四种方式测试了LIGER。首先,他们使用这种工具来定量确定不同大脑区域和不同动物之间的小鼠脑细胞类型差异。其次,他们使用LIGER通过比较来自人类和小鼠相同大脑区域的脑细胞来探究不同物种之间的相似性和差异。第三,他们使用这种工具研究单细胞RNA测序(scRNA-seq)和原位RNA测序分析的小鼠脑细胞的空间关系,从而将受测试的细胞与它们的原始大脑位置相匹配在一起。第四,这些研究人员使用LIGER通过将scRNA-seq和DNA甲基化谱合并在一起来研究小鼠脑细胞的表观基因组特征。(生物谷 Bioon.com)
参考资料:
Joshua D. Welch et al. Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity. Cell, 2019, doi:10.1016/j.cell.2019.05.006.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


