Nat Biotechnol | RNA结构解析新纪元:一种全新的自动化、高效率的冷冻电镜图谱分析方法
来源:生物探索 2024-03-08 11:20
该研究开发了一种基于深度学习的方法,从冷冻电镜图中自动构建全长全原子RNA结构,称为EMRNA。
华中科技大学黄胜友及肖奕共同通讯在Nature Biotechnology 在线发表题为“All-atom RNA structure determination from cryo-EM maps”的研究论文,该研究开发了EMRNA,一种从冷冻电子显微镜图中准确、自动测定全长全原子RNA结构的方法。EMRNA集成了基于深度学习的核苷酸检测、考虑序列和二级结构信息的三维骨架追踪和评分以及RNA结构的全原子构建。在2.0-6.0 Å分辨率的140种不同RNA图谱上验证了EMRNA,范围从37到423 nt,并将EMRNA与auto-DRRAFTER、phenix.map_to_model和CryoREAD对71例案例里进行了分析进行了比较。
对于全长RNA结构,相比于auto-DRRAFTER的中位精度为6.66 Å和0.58TM评分,EMRNA的中位精度为2.36 Å均方根偏差和0.86 TM评分。EMRNA在构建的模型中也获得了较高的残基覆盖率和序列匹配度,分别为93.30%和95.30%,而phenix.map_to_model的残基覆盖率和序列匹配度分别为58.20%和42.20%。CryoREAD的为56.45%和52.3%。EMRNA速度快,可以在3分钟内构建100 nt的RNA结构。

冷冻电子显微镜(cryo-EM)是目前测定大分子结构最重要的实验方法之一。数据收集和图像处理的自动化导致发表的冷冻电镜图数量呈指数级增长。随着实验技术的进步和计算算法的创新,结构生物学领域在过去十年中得到了长足的发展。许多大分子复合物的三维(3D)结构已经从冷冻电镜图中确定并存储在蛋白质数据库(PDB)中。然而,从冷冻电镜图中确定结构仍然是一项具有挑战性和费力的任务,特别是从中分辨率图中建立模型。因此,冷冻电镜图和模型结构之间的差距越来越大,这就需要先进的方法来从冷冻电镜图中精准确定结构。
RNA自身或与蛋白质相互作用在各种生物过程中发挥着重要作用,因此确定其三维结构对了解其功能具有重要价值。然而,与利用cryo-EM图谱测定蛋白质结构的进展相比,利用cryo-EM图谱建立RNA结构模型的进展还远远落后。由于RNA比蛋白质具有更高的内在异质性和动态性,在RNA-蛋白质复合物的冷冻电镜图谱中,RNA的图谱质量通常低于蛋白质。即使在3-4 Å分辨率下,RNA主干的连通性仍然不清楚,而在>4 Å分辨率的图谱上,只有整体的褶皱和凹槽是可见的。这对RNA结构的测定提出了很大的挑战。因此,在冷冻电镜(cryo-EM)构建RNA -蛋白组装模型时,有时会忽略RNA区域的结构。
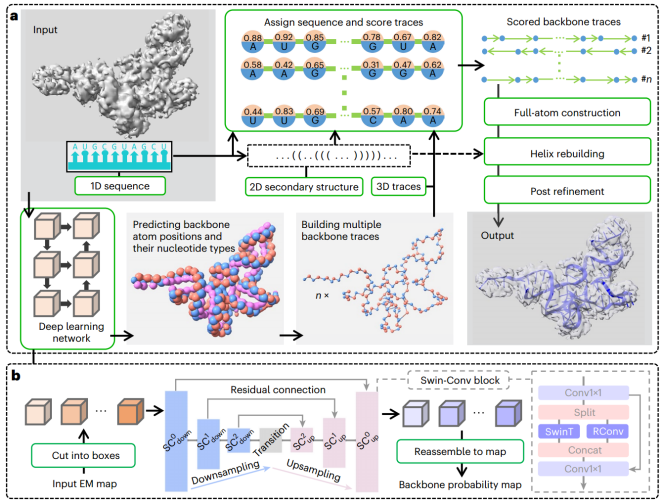
EMRNA框架(Credit: Nature Biotechnology)
最近开发了几种方法来从冷冻电镜图中模拟RNA结构,包括Brickworx, phenix.map_ to_mode、Deeptracer和CryoREAD。然而,它们都是为构建初始RNA片段而不是全长结构而开发的。DRRAFTER是第一种通过人工放置从冷冻电镜图谱中构建蛋白质-RNA复合物全原子结构的方法。它的扩展版本auto-DRRAFTER,设计用于自动构建全长RNA-only结构与RNA二级结构(SSs)从M2-seq实验。然而,它的性能很大程度上取决于所提供的RNA SSs的准确性。此外,auto-DRRAFTER非常耗时,由于其Monte-Carlo搜索特性,可能不一定能找到正确的RNA片段组装。
该研究开发了一种基于深度学习的方法,从冷冻电镜图中自动构建全长全原子RNA结构,称为EMRNA。基于Swin-Conv-UNet架构,结合多个学习目标。EMRNA通过整合四个建模步骤自动从图谱中构建完整的RNA结构:使用深度学习预测RNA的MCPs和核苷酸类型,通过TSP算法迭代地从MCPs中贯穿主链轨迹,通过将RNA序列与满足预测RNA SS的主链轨迹进行比对来构建RNA主干,以及从RNA主干构建全原子模型。对140个RNA图谱的广泛评估表明,EMRNA具有较高的准确性、稳健性和普遍适用性。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


