NAR:浙大侯廷军团队报道蛋白降解靶向嵌合体数据库PROTAC-DB 2.0
来源:生物世界 2022-11-04 15:48
因此,在2.0版本中,作者利用自主开发的PROTAC-Model建模方法为活性较好的PROTAC分子构建了相应的三元复合物计算模型,辅助研究者进行基于结构的PROTAC理性设计。
蛋白降解靶向嵌合体(Proteolytic targeting chimera,PROTAC)是一种新兴的药物治疗手段,其可介导靶蛋白和E3泛素连接酶间的相互作用,并通过泛素-蛋白酶体系统实现对靶蛋白的特异性降解。
凭借着不同于传统小分子抑制剂的作用机制,PROTAC有望靶向过去被认为是无法成药的蛋白以及克服现有药物耐药,因此近年来受到工业界与学术界的广泛关注。但是,受限于PROTAC的分子特性,设计出高选择性、高活性、可口服的PROTAC分子仍困难重重。因此,迫切需要构建一个全面地PROTAC信息系统用于帮助研究者进行PROTAC的理性设计。
近日,浙江大学药学院侯廷军教授课题组在 Nucleic Acids Research 期刊发表了题为:PROTAC-DB 2.0: an updated database of PROTACs 的论文,对初代PROTAC-DB做出了重大更新。
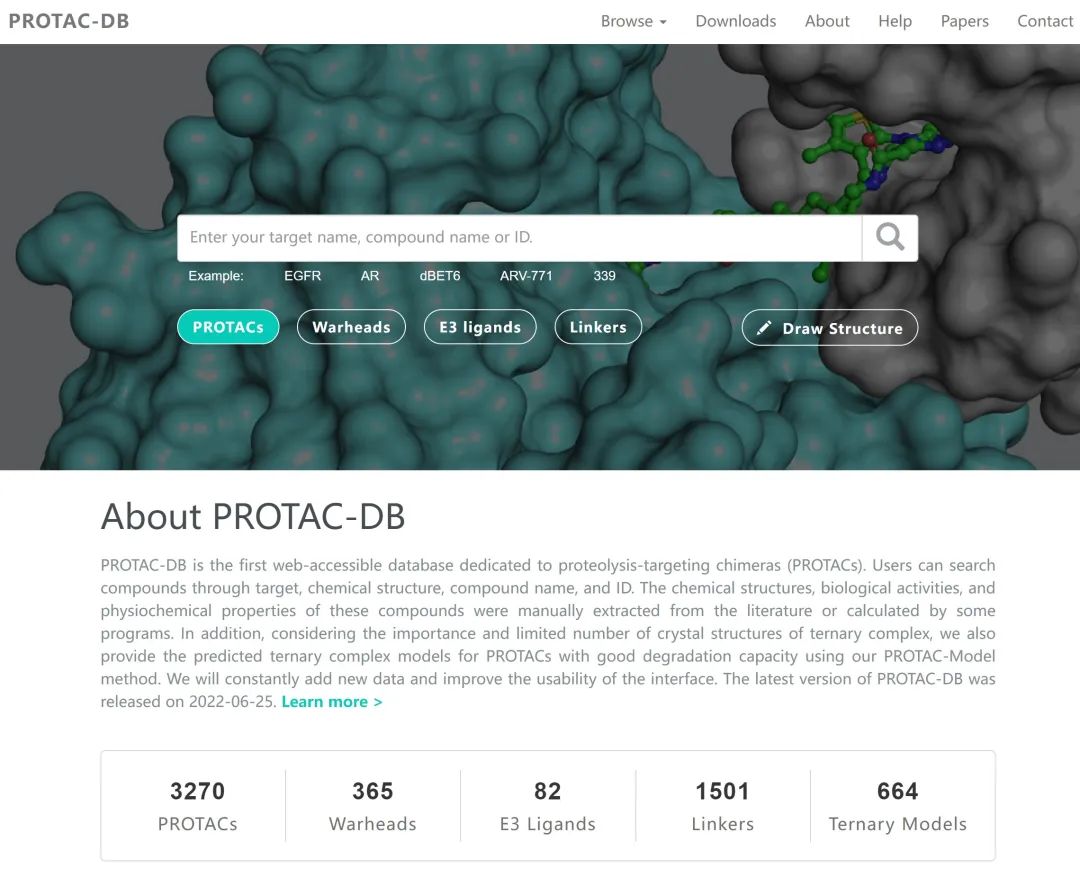
PROTAC-DB始建于2020年,是首个PROTAC在线数据平台,囊括了化学结构、生物活性、理化性质在内的多种数据,现已成为PROTAC领域的重要数据资源。该平台在发表之初,便受到了PROTAC技术创始人Craig Crews教授的推荐,并多次被 Nature Reviews Drug Discovery、Chemical Society Reviews 等国际权威期刊正面引用和介绍,现平台访问量已突破75,000次,为ESI高被引论文。

PROTAC 2.0做出了三大更新。首先,PROTAC 2.0大幅增加了收录的PROTAC分子数目以及相应的多种数据(从初代1662个PROTAC分子增至3270个)。其次,考虑到实验上仅解析出少量的PROTAC介导三元复合物晶体结构,而其结构信息可为PROTAC的理性设计提供巨大的帮助。
因此,在2.0版本中,作者利用自主开发的PROTAC-Model建模方法为活性较好的PROTAC分子构建了相应的三元复合物计算模型,辅助研究者进行基于结构的PROTAC理性设计。最后,PROTAC 2.0进一步优化了交互界面,为用户带来了更友好的使用体验。所有数据均可通过http://cadd.zju.edu.cn/protacdb/进行访问。
浙江大学药学院为本论文的第一署名单位,博士研究生翁高棋为第一作者,浙江大学药学院侯廷军教授和李丹副教授为共同通讯作者。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


