Genome Medicine:浙江大学良渚实验室沈宁团队发布实现单细胞水平肿瘤细胞群突变与表达异质性的同步研究算法
来源:生物世界 2023-12-27 11:00
该研究为癌症领域单细胞水平基因型与表型关联的异质性与进化研究提供方便有效的工具,为临床诊断提供新的方向。
浙江大学良渚实验室研究员沈宁、博士生张天韵等人在 Genome Medicine 期刊发表了题为:De novo identification of expressed cancer somatic mutations from single-cell RNA sequencing data 的研究论文。
该研究报道了一种高精确度(precision)直接从scRNA-seq数据中提取体细胞突变的算法框架——RESA(Recurrently Expressed SNV Analysis),该算法在多种类型的不同来源的癌症单细胞数据集上进行测试,均取得了较高的精确度。
该算法可以应用于癌症的肿瘤异质性与耐药机制研究,实现单细胞水平表达量变化与体细胞突变的双重检测,推进肿瘤单细胞水平基因型到表型变化的理解与认知,对肿瘤分子机制研究、临床诊断、以及个性化治疗方案的制定都有重要意义。

由于癌细胞以克隆方式实现快速复制,因此表达的体细胞突变具有cross-cell recurrence,而伪信号则更偏向随机分布且同时在多个细胞中出现的概率较低。基于该假定,研究人员开发了RESA(Recurrently Expressed SNV Analysis)算法。
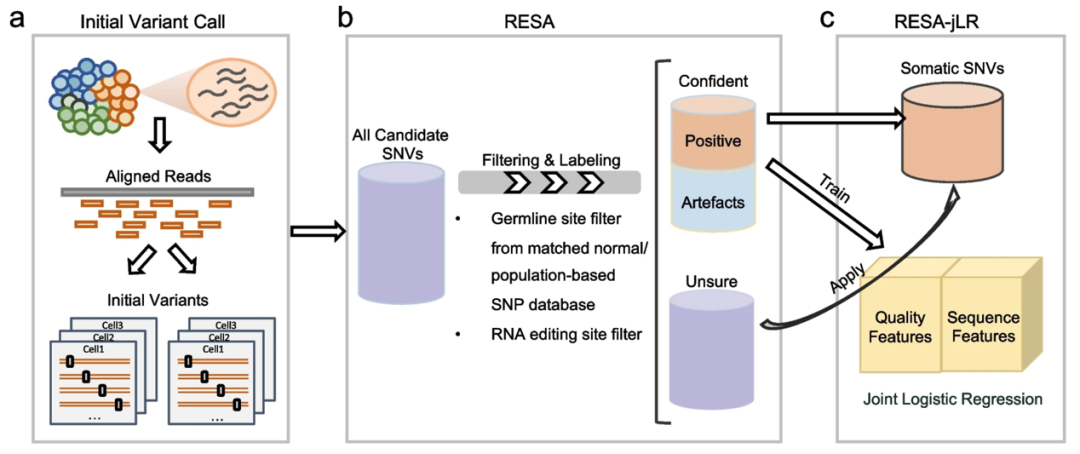
RESA算法主要分为三个阶段:初步变异检测,过滤和识别突变,以及建模再过滤。第一步将测序原始数据与基因组进行比对,并调用突变识别算法。在这一阶段,为了最小化比对和突变鉴定算法的bias,研究人员应用了两组独立read mapping和variant calling组合,确保初步鉴定的候选突变并非由数据处理算法偏差导致。第二步,研究人员通过对一系列条件进行过滤筛选,包括cross-cell recurrence数量,将候选突变分为三组,高度可信的体细胞突变或噪音信号,等待后续建模预测的候选突变以及被过滤突变。最后,研究人员将高可信度的体细胞突变定义为positive,可信的噪音定义为artefacts,建立分别包含质量相关特征和序列相关特征的联合逻辑回归模型,并将该逻辑回归分类器应用于候选突变,进一步精准识别体细胞突变从而提高整个生信模型框架的灵敏度。
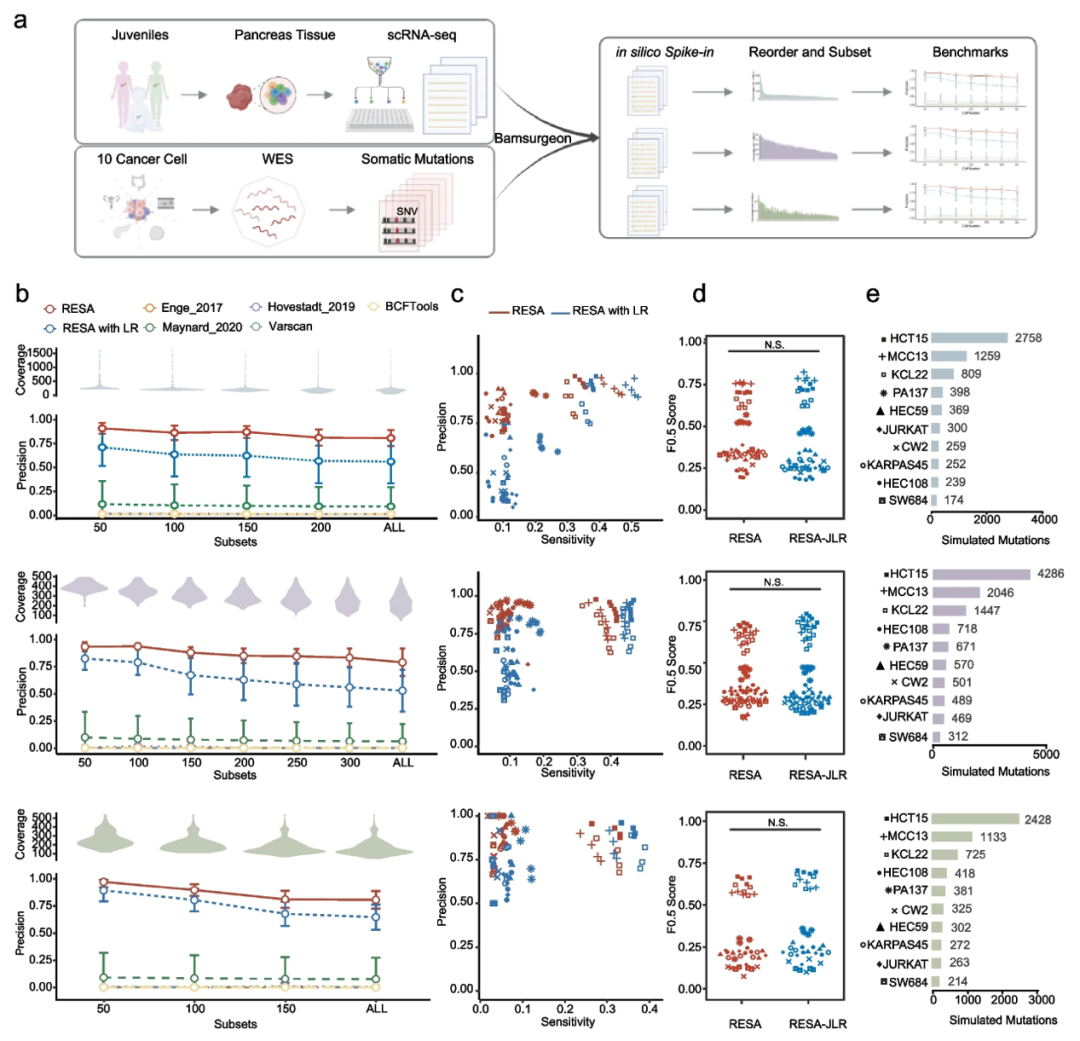
研究人员分别在三个组织样本的模拟数据集中对RESA进行了评估,发现RESA可以稳定的实现0.86的精确度。此外,在涵盖不同实验技术和条件下产生的15个癌症细胞系数据中,RESA的精确度也要远优于其他体细胞检测算法。这表明了RESA在适应特定细胞类型方面的灵活性,以及RESA在实验条件下的鲁棒性。
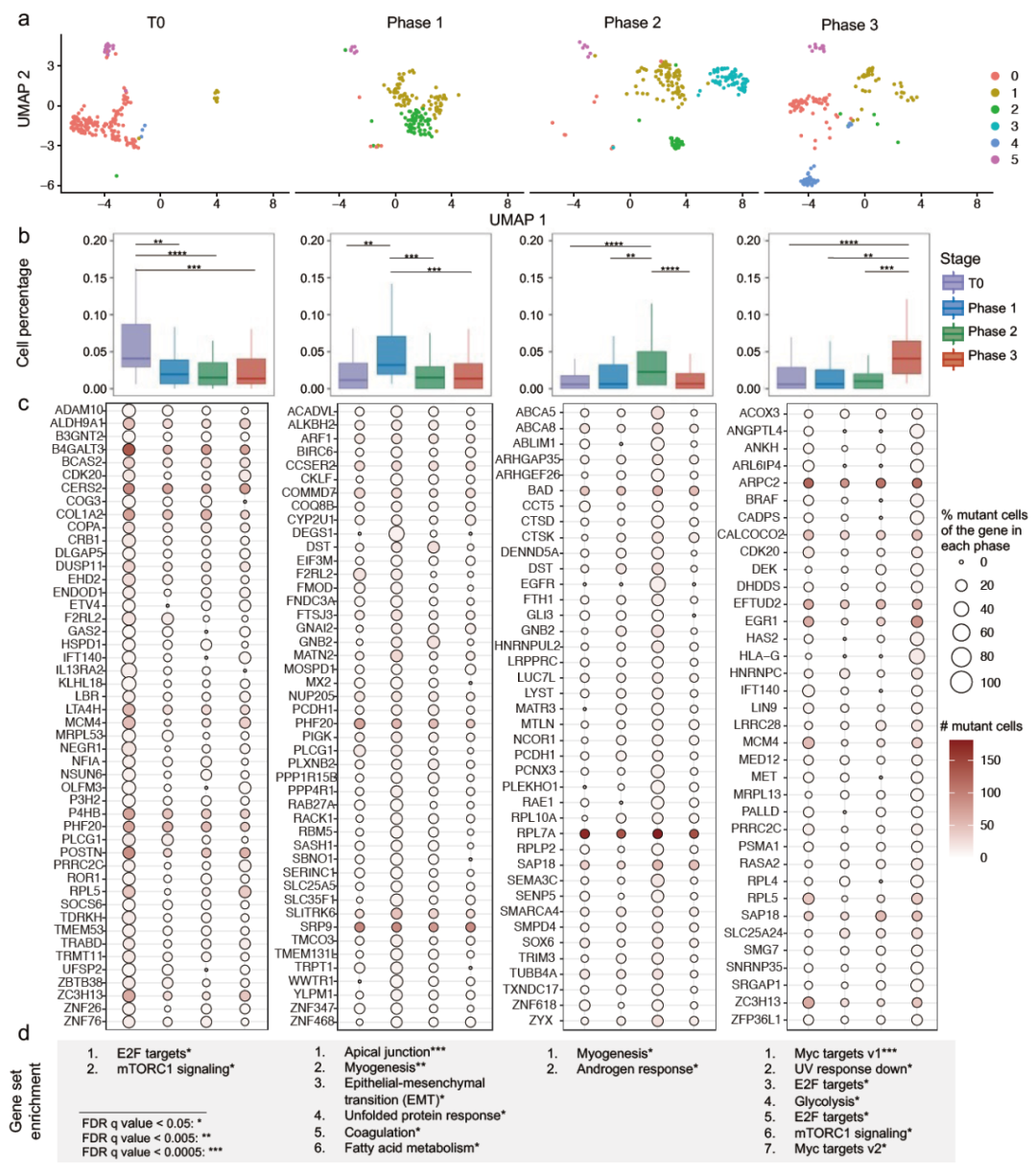
研究人员进一步分析了黑色素瘤的基于人源性肿瘤移植模型(Patient-derived xenograft,PDX),模型对四个时间点的单细胞数据集进行RAF/MEK抑制耐药性研究。这四个时间点T0、phase1、phase2、phase3,分别对应肿瘤治疗前,肿瘤缩小期,微小残留病(MRD)和复发期。通过RESA,研究人员成功检测到了575个体细胞突变,并对应到了524个基因上,其中包含黑色素瘤的重要驱动突变BRAFV600E。为了更好研究具有表达特征的突变,我们识别了阶段特异性的含有体细胞突变的基因,并对检测到的基因进行癌症特征基因富集,发现了包括紫外线反应,上皮到间充质转化(Epithelial to Mesenchymal Transition,EMT)通路等。综上所述,表达的体细胞突变可能以阶段特异性和突变特异性的方式扰乱肿瘤通路和特征,揭示了表达与突变的肿瘤内异质性之间的复杂关系。
综上,该研究建立了一个整合的生物信息算法框架,实现从单细胞转录组测序数据中直接并高精确度提取体细胞突变信息,可以用于单细胞水平体细胞突变与转录组表达变化的整合分析,并且证明了该算法在肿瘤异质性与耐药机制研究中的价值。该研究为癌症领域单细胞水平基因型与表型关联的异质性与进化研究提供方便有效的工具,为临床诊断提供新的方向。
浙江大学良渚实验室研究员沈宁为本文的通讯作者,沈宁团队博士生张天韵为本文的第一作者。研究受到了浙江大学计算机学院王海帅研究员,浙江大学爱丁堡大学联合学院郭伟教授,北京大学席瑞斌教授,良渚实验室郭红山研究员的大力支持。研究获得了浙江省领军型创新创业团队、浙江大学启动经费的支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


