空间转录组学新突破:摆好聚光灯,让RNA自己说话!
来源:生物探索 2022-10-18 17:14
在多细胞生物中,不同类型的细胞在组织内会以特殊的模式组织起来。在显微镜下观察,这些形形色色的细胞可能因为占据了独特的位置、表现为不寻常的形状或表达特定的生物标志物分子而千姿百态。
在多细胞生物中,不同类型的细胞在组织内会以特殊的模式组织起来。在显微镜下观察,这些形形色色的细胞可能因为占据了独特的位置、表现为不寻常的形状或表达特定的生物标志物分子而千姿百态。当然,这些细胞之间还存在着更深层意义的差别,即基因表达的差别。研究人员通过转录组测序,即对细胞内存在的多种RNA分子进行测序,来更深入地了解细胞的类型和基因表达模式。
基因表达具有时间特异性和空间特异性,即不同的组织或细胞在特定发育阶段或功能状态下,基因表达会存在变化,比如胚胎发育过程中生物分子水平存在飞快的动态变化,同时细胞之间的空间关系导致了子细胞之间对称性的破坏、决定了细胞未来的命运。通过在不同时间点使用单细胞转录组测序技术进行采样,能够了解某一细胞或组织基因表达的时间特异性。而空间特异性信息则相对较难获得,原因是常规转录组测序和单细胞转录组测序在还原细胞所处的原始位置信息上存在困难,而传统的原位杂交技术(In Situ Hybridization,ISH)又很难实现高通量检测,一次最多只能分析少数几个基因。为此,空间转录组学技术应运而生。
2021年8月11日,美国的研究团队在Nature发表回顾常见空间转录组学技术的综述文章“Exploring tissue architecture using spatial transcriptomics”[1],将空间转录组学技术分为两大类:(1)基于新一代测序(Next-Generation Sequencing,NGS)的方法,即在NGS前将位置信息编码到转录本上;(2)基于成像的方法,该方法又分为基于原位测序(In Situ Sequencing,ISS)的方法——直接在组织中对转录本进行扩增和测序,以及基于ISH的方法——成像探针在组织中被依次杂交(图1)。这些方法虽然不同,但最终都可以看作是构建了一个基因表达矩阵,该矩阵对每个点(一个像素、一个细胞或一组细胞)的转录组进行捕获。
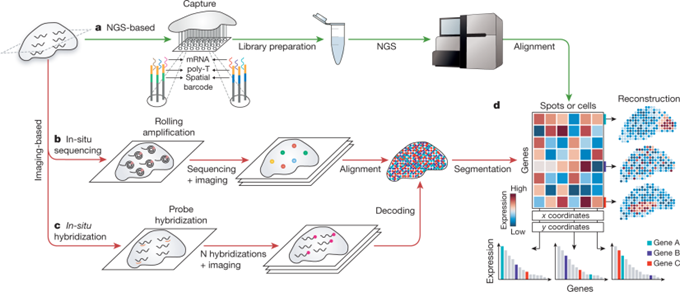
不过,现有的空间转录组学技术依然存在一些限制,比如:仅能捕获细胞总RNA分子的一小部分;达不到单细胞测序方法所能提供的分析深度和质量;破坏了原始细胞组织的环境使得对样本进行后续分析变得不可能;以及需要专门的仪器或试剂导致成本高昂。这些缺陷阻碍了对不连续的细胞群或罕见且难以分离的细胞(比如某些特殊的脑细胞,或侵入肿瘤的免疫细胞)的研究。
为此,哈佛大学Wyss仿生工程研究所的团队开发了一项名为“Light-Seq”的DNA纳米驱动技术。研究成果以“Light-Seq: light-directed in situ barcoding of biomolecules in fixed cells and tissues for spatially indexed sequencing”为题于2022年10月10日发表于Nature Methods(图2)[2]。

图2 研究成果(图源:[2])
该技术允许研究人员通过光交联过程选择特定的目标细胞,然后利用DNA标签(barcode)对目标细胞全部的RNA序列进行“地理标记”,随后在DNA驱动技术的帮助下,翻译成DNA单链以进行NGS测序。对于同一样本中的不同细胞群体,可以使用不同的DNA标签重复Light-Seq过程,以便后续分析(图3)。
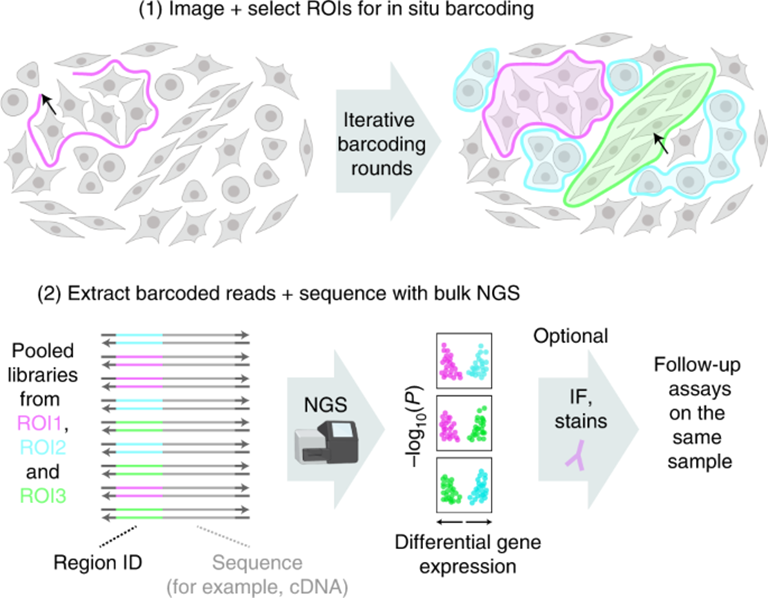
图3 Light-Seq的概览(图源:[2])
Light-Seq项目由Jocelyn(Josie)Kishi博士、Sinem Saka博士和Ninning Liu博士以及Emma West博士牵头。2019年,Kish、Saka和West共同开发了一种名为SABER-FISH的空间转录组学方法,该方法用于直接在完整组织中对基因表达进行成像,能够在固定的细胞和组织中将RNA和DNA FISH信号放大5到450倍。尽管如此,“距离捕捉细胞完整的基因表达程序还有好几个数量级的差距。每个细胞有数千个不同的RNA分子,RNA分子过于密集,无法用目前的成像技术完整捕获。”Kishi说,“而Light-Seq借助更高分辨率的DNA标签和NGS全转录组测序,两全其美地解决了这个问题。”
首先,DNA引物与细胞中的RNA分子进行碱基配对,以RNA为模板扩建出RNA的副本,即互补DNA(complementary DNA,cDNA)。然后,通过紫外光的照射,将含有超快光交联剂的标签DNA通过光交联反应“粘到”目标区域的cDNA上,而未被紫外光照射的区域,标签DNA会在之后的步骤中被洗脱,这样就对感兴趣的区域打上了标记。若是使用不同的标签配合照射不同区域的紫外光重复该过程,则能标记更多的区域。
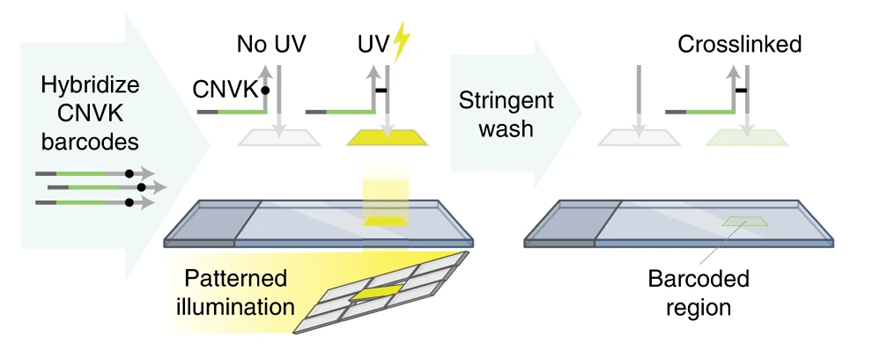
图4 DNA光刻进行空间寻址标记(图源:[2])
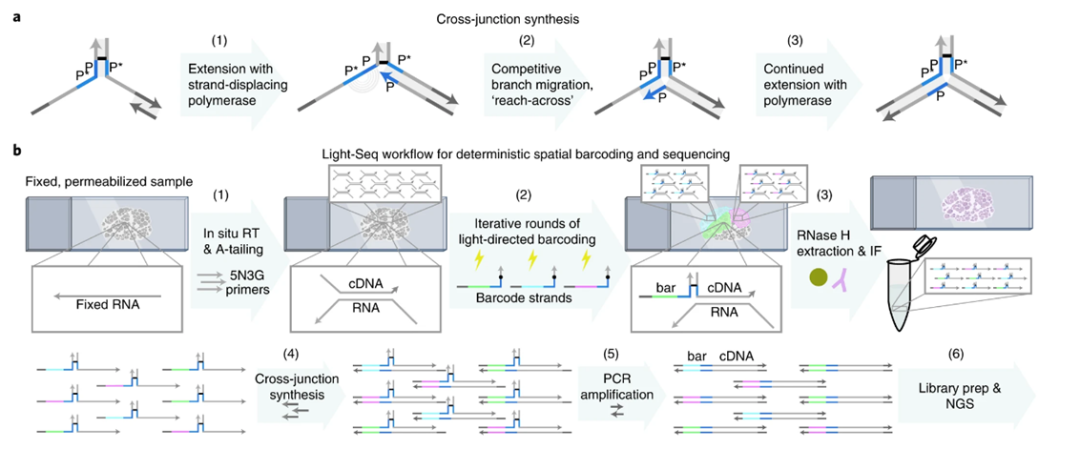
图5 拼接反应与Light-Seq的工作流程(图源:[2])
为了验证该技术的细胞选择能力和“贴标签”的能力,研究人员将小鼠3T3细胞和稳定表达eGFP的人类HEK细胞混合进行测试。基于eGFP表达和细胞形态信息,研究人员对细胞进行了手动选择并标记以不同的“标签”。结果表明,小鼠和人类图谱对于其各自的“标签”具有良好的区分率,尤其是eGFP reads归类到人类“标签”的正确率高达93±0.5%。序列提取后,又对样本进行了多重免疫荧光,验证了样本具备进行二次分析的完整性。
在培养细胞中首次验证Light-Seq之后,研究人员希望将其应用于更复杂的组织,因为组织样本中特定细胞群的RNA测序仍然充满挑战性,尤其是当靶细胞很少或难以分离时。为此,Kishi,Saka,Liu和West联手,将Light-Seq应用于小鼠的视网膜切片。研究人员手动划分了视网膜经典具备不用功能的三个细胞层。结果表明,Light-Seq达到了与单细胞测序方法相当的序列覆盖率,并发现视网膜的三个主要层之间富集了数千个RNA(图6)。此外,样本保持了完整,仍能进一步对蛋白质和其他生物分子进行成像。
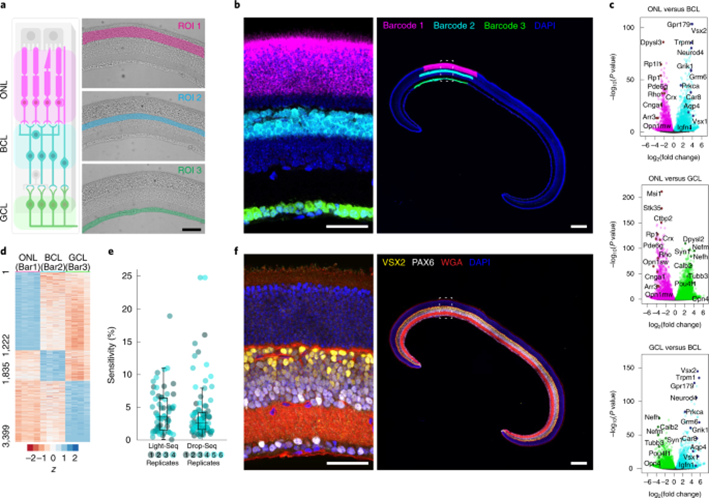
图6 Light Seq应用于小鼠视网膜切片(图源:[2])
将Light-Seq发挥到极致,研究人员分离出了一种非常罕见的细胞类型的完整转录组,这种细胞与视网膜内的其他细胞联系极其复杂,因此难以分离。“Light-Seq还提取了该细胞中表达水平极低的特异性RNA,以及据我们所知前人从未描述过的数十种特异性生物标志物RNA,这为研究这种罕见细胞类型开辟了新的机会。”West说。
将空间转录组学领域向NGS敞开还带来了有关单个RNA种类水平的信息。Kishi表示:“Light-Seq可以确定RNA结构的自然变异。”展望未来,Kishi对Light-Seq应用于更好地了解免疫系统、疾病传播细胞以及基因和细胞治疗等不同治疗策略之间的相互作用充满兴趣,现在,她正积极地与合作者们一起寻求将Light-Seq商业化的道路。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


