Nature子刊:西湖大学贾洁敏团队发现神经元调控大脑血流新路径——类突触连接
来源:生物世界 2024-01-04 10:29
这项研究在小鼠中证实了证明单个谷氨酸能轴突通过类突触——神经-小动脉平滑肌细胞接头(NsMJ)之间的突触样传输扩张其神经支配的小动脉。
西湖大学生命科学学院贾洁敏团队在神经科学顶级期刊 Nature Neuroscience 上发表了题为:Synaptic-like transmission between neural axons and arteriolar smooth muscle cells drives cerebral neurovascular coupling 的研究论文。
该研究发现了一座横架在神经元与血管之间的“新桥梁”——类突触连接(NsMJ)——通过它,谷氨酸能神经元可以直接作用于动脉血管平滑肌细胞,导致动脉舒张,诱发大脑功能性充血。这一发现是对现有脑血流调控机制的进一步完善和补充,也是对实现脑血流快速和精准调控的一个全新认知,为临床上有效治疗缺血性低灌注损伤提供了潜在策略。

基于电子显微镜的最新研究揭示了星形胶质细胞终足(astrocytic endfeet)上覆盖着毛细血管的周细胞区域的缝隙,表明轴突可能与周细胞有物理接触。周细胞和aSMC具有相同的胚胎起源,并被统称为壁细胞。如果轴突末端能够通过终足缝隙,它们就可能形成神经-小动脉平滑肌细胞接头(NsMJ),就像骨骼神经肌肉接头一样。随着当前大规模组织的连续切片扫描电子显微镜(SBF-SEM)的应用,这一假设可以直接得到验证。
大量研究表明,神经递质谷氨酸在神经活动期间对脑血流量的调节中发挥重要作用。虽然谷氨酸受体中间细胞,例如中间神经元、星形胶质细胞和锥体神经元,已被证明通过释放一氧化氮、花生四烯酸衍生物和前列腺素来调节或部分调节神经血管耦合(NVC),但尚未考虑谷氨酸直接作用于aSMC并放松小动脉的可能性。
最近的研究表明,中枢神经系统的内皮细胞中表达的功能性GluN1介导了NVC。小鼠和人类单细胞RNA测序(RNA-seq)研究报告,大脑aSMC和内皮细胞表达类似水平的Grin1 mRNA,并且aSMC在脑细胞中具有第二高的Grin1 mRNA丰度水平,尽管仍远低于神经元。这表明aSMC本身可能具有谷氨酸感知能力,而是否谷氨酸能轴突属于一种血管周围神经末梢仍有待确定。
急性缺血性卒中由于过度谷氨酸释放而产生兴奋毒性,并产生扩散去极化(SD),导致随后的小动脉收缩。然而,过度谷氨酸引起的aSMC中NMDA受体的过度激活并没有直接与SD后的小动脉收缩相关。此外,在兴奋毒性期间特异性阻断血管NMDA受体激活是否可以缓解SD引起的血管收缩完全未知。收缩性aSMC依赖于胞内Ca2+振荡来控制血管运动活动。如果aSMC表达Ca2+通透性NMDA受体,研究团队推测过多的谷氨酸可能破坏aSMC的Ca2+稳态,这可能与SD期间aSMC收缩行为有关,从而扩大梗死面积。
在这项研究中,贾洁敏团队结合多种前沿技术,证实了半个多世纪前提出的突触样传输假说。
首先,使用大体积三维扫描电镜和光电联合技术和RNA-seq,克服了电子显微镜的吞吐量问题,并明确识别了不同类型的NsMJ超微结构和aSMC表达的多功能神经递质受体。
其次,使用胶体金电子显微镜,证明了小鼠、猴子和人类aSMC中存在NMDA受体。
第三,体外和体内功能性钙成像和全细胞膜片钳记录揭示了这些NMDA受体的功能,根据谷氨酸水平产生生理和病理结果。
第四,单轴突光遗传学和自然感觉输入验证了谷氨酸能轴突NsMJ(Glu-NsMJ)传输在介导NVC方面至少与PGE2扩散一样重要。
最后,具有aSMC特异性NMDA受体干扰的小鼠在缺血性卒中后表现出减弱的血管收缩和改善的结局。
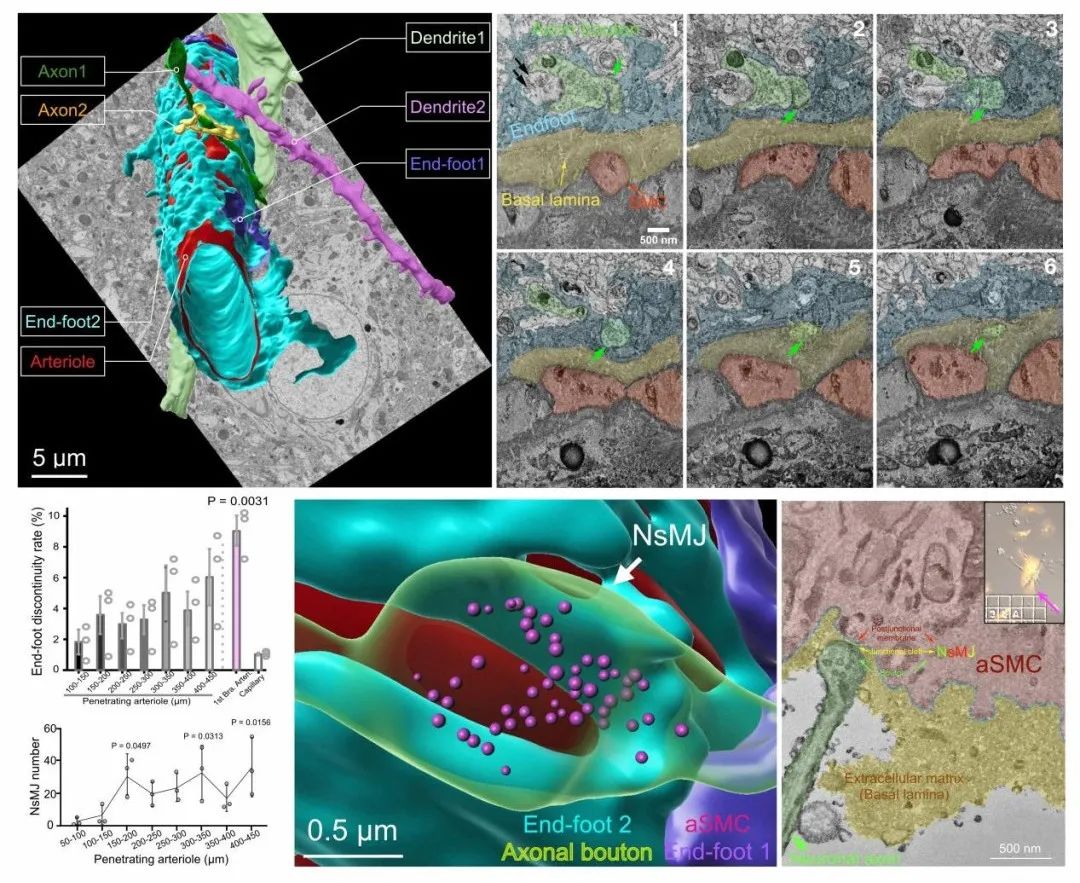
神经元与动脉血管平滑肌细胞之间形成一种“类突触”(NsMJ)
总的来说,这项研究在小鼠中证实了证明单个谷氨酸能轴突通过类突触——神经-小动脉平滑肌细胞接头(NsMJ)之间的突触样传输扩张其神经支配的小动脉。突触前亲子键对突触后树突和小动脉平滑肌细胞(aSMC)进行双向神经支配,其中包括低水平的谷氨酸NMDA受体亚单位1(Grin1)。通过aSMC特异性敲除GluN1来中断NsMJ传输,减少了光遗传学和胡须刺激引起的小鼠大脑功能性充血。值得注意的是,aSMCs中缺少GluN1亚基通过防止缺血诱导的扩散去极化引起的小动脉收缩期间aSMC中的Ca2+过载,减少了脑缺血后的脑萎缩。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


