Nat Genet:通过表观遗传疗法激发转座子新抗原,推进癌症免疫治疗
来源:生物探索 2024-09-06 14:59
研究团队以胶质细胞瘤为模型,成功证明了表观遗传疗法激活转座子,从而产生更多转座子新抗原的可能性,为将来表观遗传疗法和免疫疗法的联合治疗提供了理论基础。
近年来,免疫疗法已在多种癌症的治疗中取得了突破性的进展。这种疗法通过利用肿瘤中特异性表达的新抗原(neoantigen),使免疫细胞能够精准地锁定并消灭癌细胞【1】。传统上,人们认为这些新抗原主要源于编码基因的突变。然而,近年研究表明,基因组中非编码区域在肿瘤中的异常表达也可能导致新抗原的产生,其中转座子(Transposable elements, TEs)是一个重要的来源【2】。转座子占人类基因组的近一半,尽管在大多数正常成人组织中被抑制,但在肿瘤中它们常常被重新激活【3,4】。
最新研究发现, 转座子可以在肿瘤中成为基因的选择性启动子,进而产生转座子-基因嵌合转录本(TE-gene chimeric transcripts, TETs)【5-7】。在某些情况下,这些转录本会被翻译成同时具有转座子和基因序列的嵌合蛋白,并具有提供新抗原的潜能。
在转座子新抗原的研究领域,圣路易斯华盛顿大学遗传系王艇教授团队自2017年以来已在Nature Genetics发表了三篇重要的研究文章【6-8】,并于今年初在Nature Reviews Cancer上撰写综述,总结了这一领域的研究进展及未来展望【4】。前两篇文章阐明了转座子在肿瘤中被激活、作为启动子产生转座子-基因嵌合转录本的现象,并展现出这一现象的在不同癌症中的高度一致性,为开发通用型癌症免疫疗法提供了理论基础。第三篇文章进一步证明了表观遗传疗法能够解除转座子序列上的表观遗传抑制,重新激活转座子的启动子活性。
受此前研究的启发,2024年9月2日,王艇教授团队在Nature Genetics上发表了“第四部曲”,题为Epigenetic therapy potentiates transposable element transcription to create tumor-enriched antigens in glioblastoma cells。该研究深入描述表观遗传疗法激活更多肿瘤特异性转座子-基因嵌合转录本的可能性,为将来表观遗传疗法和免疫疗法的联合应用提供了有力的理论基础。

在过去研究中,王艇团队发现,DNA甲基转移酶抑制剂(DNMT inhibitor)以及组蛋白去乙酰化酶抑制剂(HDAC inhibitor)的协同作用,能够最大程度地激活转座子【8】(图1A)。因此,本次研究使用了这两种试剂进行表观遗传疗法处理。在之前的泛癌分析中,研究者发现胶质细胞瘤中转座子的激活程度相对偏低【7】。同时,由于胶质细胞瘤基因突变较少,使得传统免疫疗法效果有限【9】。为了验证表观遗传疗法对转座子的激活效果,并为胶质细胞瘤提供新的治疗方法,研究者对3个胶质细胞瘤干细胞系进行表观遗传疗法处理(图1A,B),并同时对原代胶质细胞和原代纤维母细胞进行相同的处理,以分析疗法对正常组织的影响。
研究者应用自主开发的算法TEProf2(TE Promoter Finder 2)【7】,从转录组数据中识别出转座子-基因嵌合转录本,并通过使用原代胶质细胞和原代纤维母细胞为参照,筛选出73个仅在肿瘤细胞中被激活的TETs(图1B,2A)。与之前的研究成果相似,这些TETs在三个细胞系中高度重合【7】(图2A)。通过HLA-pulldown质谱数据,研究者成功检测到5个被呈递到HLA上的转座子新抗原。为了进一步提高检测灵敏度,研究者使用了PacBio长测序技术,从其中一个胶质细胞瘤干细胞系中发现了1,161个TETs,并从质谱中发现了18个转座子新抗原。鉴于表观遗传疗法会对全基因组产生影响并造成潜在的副作用,研究者成功利用CRISPRa技术对转座子进行定点激活。在两个胶质细胞瘤干细胞系中,他们成功激活了三个不同的转座子拷贝,观察到了显著的基因表达变化和蛋白表达,并通过质谱数据验证了其中一个TET产生的蛋白上包含转座子提供的氨基酸序列。
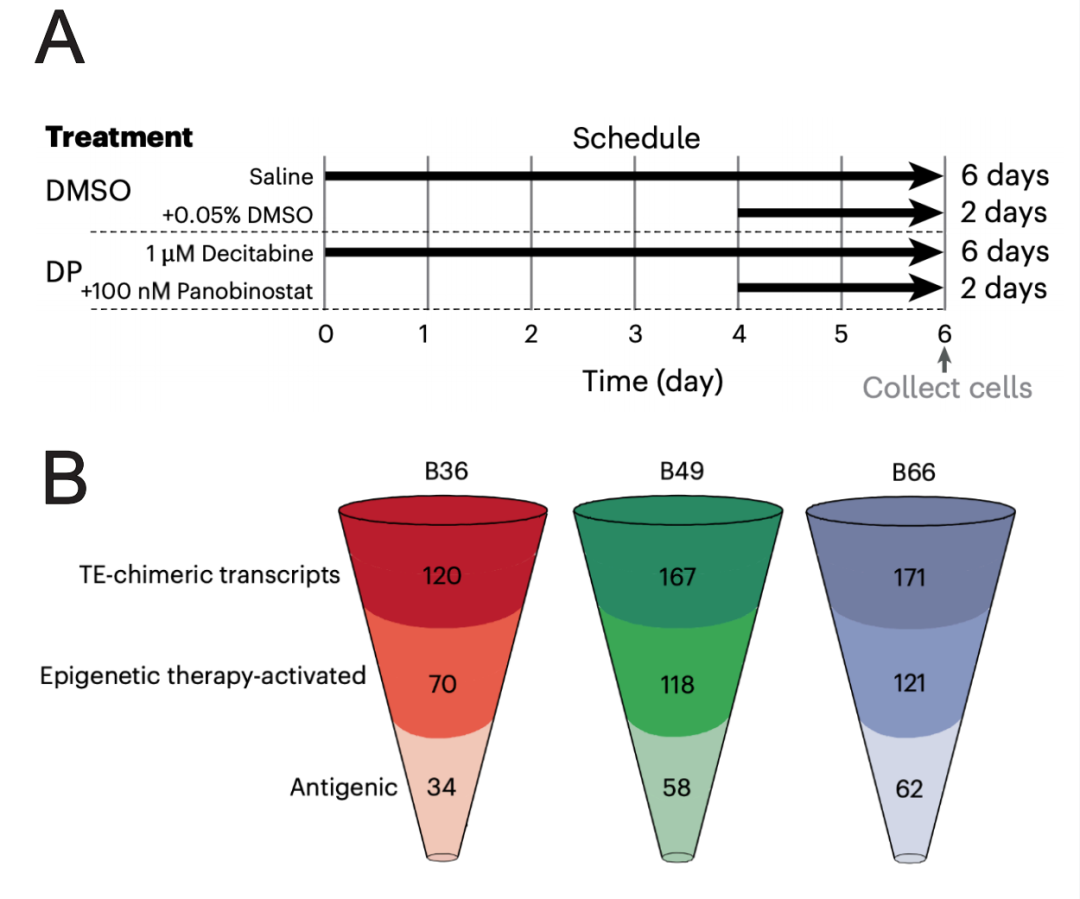
图1. (A)实验设计示意图(B)表观遗传疗法激活的转座子-基因嵌合转录本及其可能产生的抗原的数量分布(Credit: Nature Genetics)
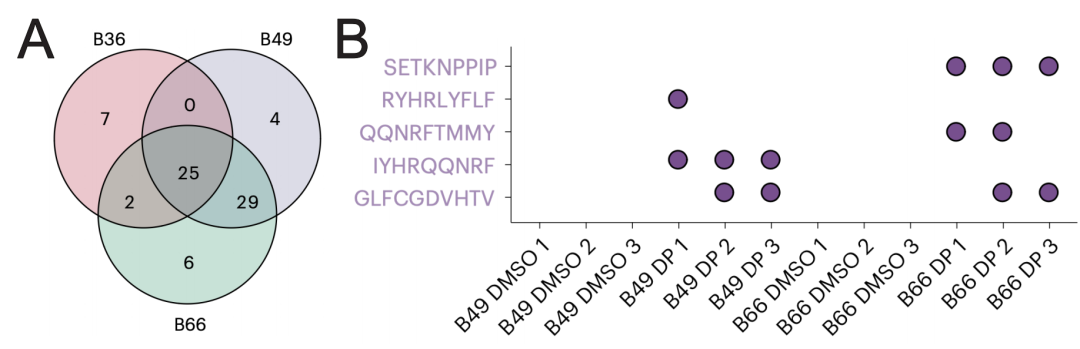
图2.(A)表观遗传疗法激活的转座子-基因嵌合转录本在三个肿瘤干细胞系中的分布(B)从质谱数据中检测到的转座子新抗原在两个肿瘤干细胞系中的分布(Credit: Nature Genetics)
参考文献:
1. June, C. H., O’Connor, R. S., Kawalekar, O. U., Ghassemi, S. & Milone, M. C. CAR T cell immunotherapy for human cancer. Science 359, 1361–1365 (2018).
2. Laumont, C. M. et al. Noncoding regions are the main source of targetable tumor-specific antigens. Sci. Transl. Med. 10, eaau5516 (2018).
3. Burns, K. H. Transposable elements in cancer. Nat. Rev. Cancer 17, 415–424 (2017).
4. Liang, Y., Qu, X., Shah, N. M. & Wang, T. Towards targeting transposable elements for cancer therapy. Nat. Rev. Cancer 24, 123–140 (2024).
5. Babaian, A. & Mager, D. L. Endogenous retroviral promoter exaptation in human cancer. Mob. DNA 7, 24 (2016).
6. Jang, H. S. et al. Transposable elements drive widespread expression of oncogenes in human cancers. Nat. Genet. 51, 611–617 (2019).
7. Shah, N. M. et al. Pan-cancer analysis identifies tumor-specific antigens derived from transposable elements. Nat. Genet. 55, 631–639 (2023).
8. Brocks, D. et al. DNMT and HDAC inhibitors induce cryptic transcription start sites encoded in long terminal repeats. Nat. Genet. 49, 1052–1060 (2017).
9. Charoentong, P. et al. Pan-cancer Immunogenomic Analyses Reveal Genotype-Immunophenotype Relationships and Predictors of Response to Checkpoint Blockade. Cell Rep. 18, 248–262 (2017).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


