研究探索植物SWI/SNF亚复合体的组装过程及机制
来源:网络 2023-05-06 11:33
该研究系统研究了三种不同的拟南芥SWI/SNF亚复合体(SAS、MAS和BAS)的靶基因结合机制并初步探索了它们的组装机制。通过全面比较三个亚复合体的染色质结合图谱,发现SAS、MAS和BAS亚复合体
Switch defective/sucrose non-fermentable(SWI/SNF)复合体是一个进化上保守的由多亚基组成的ATP依赖性染色质重塑复合体。它通过ATP水解提供能量重塑染色质结构, 从而对基因转录进行调控。植物SWI/SNF亚基在细胞分化、发育和对各种环境信号的反应中起着关键作用,它们的突变导致叶片发育、根干细胞维持、花形态形成、胚胎发育以及营养体到成体阶段的转变等方面的严重缺陷。然而,这些亚基的突变如何改变植物SWI/SNF复合体在染色质上的功能并导致发育受损和对环境刺激响应缺陷的分子机制仍然未知。此外,植物的SWI/SNF亚复合体的活性和基因组靶向是否以及如何相互协调以调节染色质结构和基因表达也知之甚少。最后,植物SWI/SNF亚复合体的组装的过程及机制也完全不清楚,同样也阻碍了对植物SWI/SNF复合体功能的理解和认识。
近日,中山大学生命科学学院李陈龙课题组分离及鉴定了植物SWI/SNF亚复合体、解析了它们的靶基因结合异同及相互调控机制、初步探索了它们的亚基组装过程及机制。
为了表征三种不同的植物SWI/SNF复合体并确定其亚基组成的差异是否会导致不同的基因组靶向,该研究通过染色质免疫沉淀结合高通量测序(ChIP-seq),绘制了SAS、MAS和BAS亚复合体的全基因组靶基因图谱。发现三种植物SWI/SNF亚复合体在染色质上既有重叠也有特异性的基因组靶向性,另外三种SWI/SNF亚复合体在靶基因上的结合位置也存在差异。同时该研究也发现SAS亚复合体的突变会导致BAS亚复合体的结合往上游启动子区域偏移,揭示了BAS亚复合体的基因组定位依赖于SAS亚复合体;另外,BAS亚复合体的突变会导致SAS亚复合体在它们共靶基因上的结合强度下降(图1)。

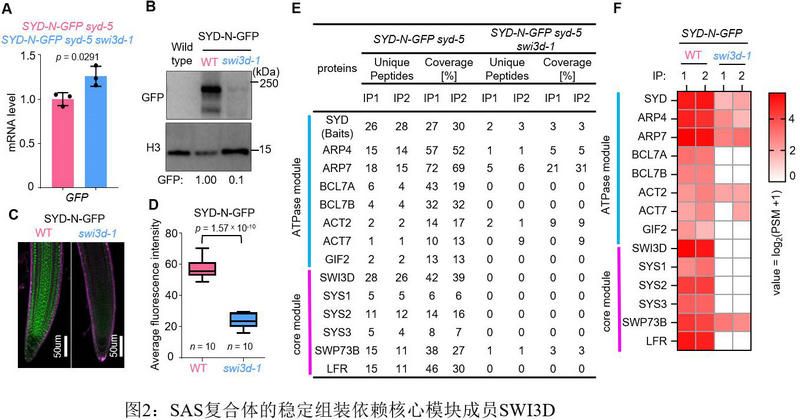
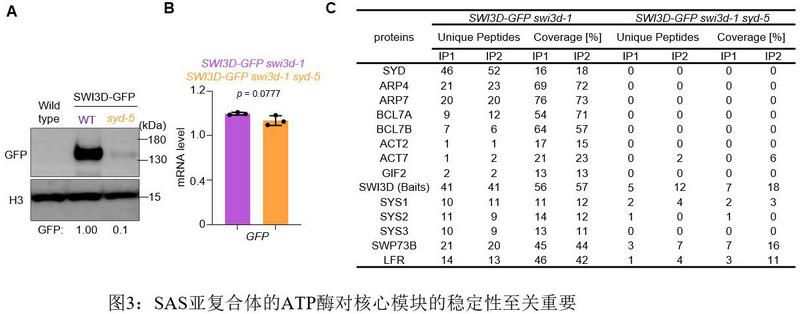
同时,该研究通过研究SAS亚复合体的组装情况,证明了SAS亚复合体核心模块亚基SWI3D在SAS亚复合体稳定中的关键作用(图2)。SWI3D丢失使SAS复合体解体,同时会破坏SYD在靶基因上的结合;另一方面,删除SWI3D 的SANT结构域会破坏SWI3D-SYD之间的相互作用从而导致SWI3D蛋白质丰度的降低. 因此,推测可能与哺乳动物SWI/SNF复合体组装不一致,植物中的ATP酶催化亚基可能稳定核心模块的组装。为了验证这种可能性,将syd-5突变体引入SWI3D-GFP转基因植株中。令人惊讶的是,发现在缺少SYD催化亚基的情况下,SWI3D-GFP蛋白水平显著降低,这表明SAS亚复合体中的ATP酶对核心模块的稳定起着至关重要的作用(图3)。
综上所述,该研究系统研究了三种不同的拟南芥SWI/SNF亚复合体(SAS、MAS和BAS)的靶基因结合机制并初步探索了它们的组装机制。通过全面比较三个亚复合体的染色质结合图谱,发现SAS、MAS和BAS亚复合体在靶基因上显示出不同的靶基因结合模式,并定位于具有不同组蛋白修饰的染色质以及可能具有潜在的复合体特异功能。此外,SAS亚复合体与BAS亚复合体在靶基因结合上相互调控,为理解不同SWI/SNF亚复合体的基因组靶向如何协调染色质结构调控提供了新的见解。最后,该工作发现SAS的核心模块和ATP酶模块在它们的组装中相互依赖,共同维持植物复合体的完整性。总之,这些发现证明了SWI/SNF染色质重塑复合体在真核生物进化过程中的差异性,并为全面理解植物SWI/SNF复合体的组装、基因组靶向和功能奠定了基础。
近日,该研究以“Organization, genomic targeting, and assembly of three distinct SWI/SNF chromatin remodeling complexes in Arabidopsis”为题在The Plant Cell发表。该研究得到了国家自然科学基金,博士后创新人才支持计划以及广东省基础与应用基础研究联合基金的资助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


