David Baker最新Science论文:AI从头设计蛋白,用作生物传感器
来源:生物世界 2024-07-22 14:59
,这项发表于 Science 的研究展示了通过深度学习模型来设计基于假环肽的、具有模块化重复结构和中央结合口袋的小分子结合蛋白,对靶标小分子化合物具有高结合亲和力,并可以实现下游传感。
人工设计蛋白领域先驱 David Baker 教授(安琳娜博士等人为第一作者)在 Science 期刊发表了题为:Binding and sensing diverse small molecules using shape-complementary pseudocycles 的研究论文。
该研究利用人工智能从头设计了一种带有中心空腔的、小分子量的假环肽,可以通过改变重复单元的数量来扩大或缩小,以匹配目标配体的大小。研究团队展示了这种假环肽的两种应用:1)作为一个结构域结合到一个更大的、全新设计的蛋白质中;2)用于小分子传感的纳米孔融合以及用于化学诱导二聚化的分裂蛋白 。这些发现将为蛋白质从头设计和生物传感开辟新的研究方向。

目前,蛋白质设计的一个主要挑战是创造高选择性和高亲和力的小分子结合蛋白。高亲和力需要与大多数可用原子接触的高形状互补袋,因此相比于蛋白质结合物,小分子结合蛋白设计更加困难,因为蛋白质与小分子化合物之间可以进行相互作用的原子更少。
此前,在小分子结合蛋白设计方面的成功使用基于物理和基于深度学习扩散的方法,主要集中在刚性疏水靶标上,并且通常需要大量的实验优化才能将结合亲和力从微摩尔(μM)提高到纳摩尔(nM)水平。除了结合之外,一个普遍的未解决的挑战是将结合事件转导为下游信号,即生物传感。
在这项最新研究中,David Baker 团队着手开发一种通用的方法来设计具有高形状互补性的小分子结合蛋白,并可应用于下游传感。研究团队假设,与基于固定支架的方法相比,从鉴定与靶向小分子具有高度形状互补性的蛋白质支架开始的设计方法将能够实现更高的亲和力结合,并能够与柔性和极性靶标小分子结合。
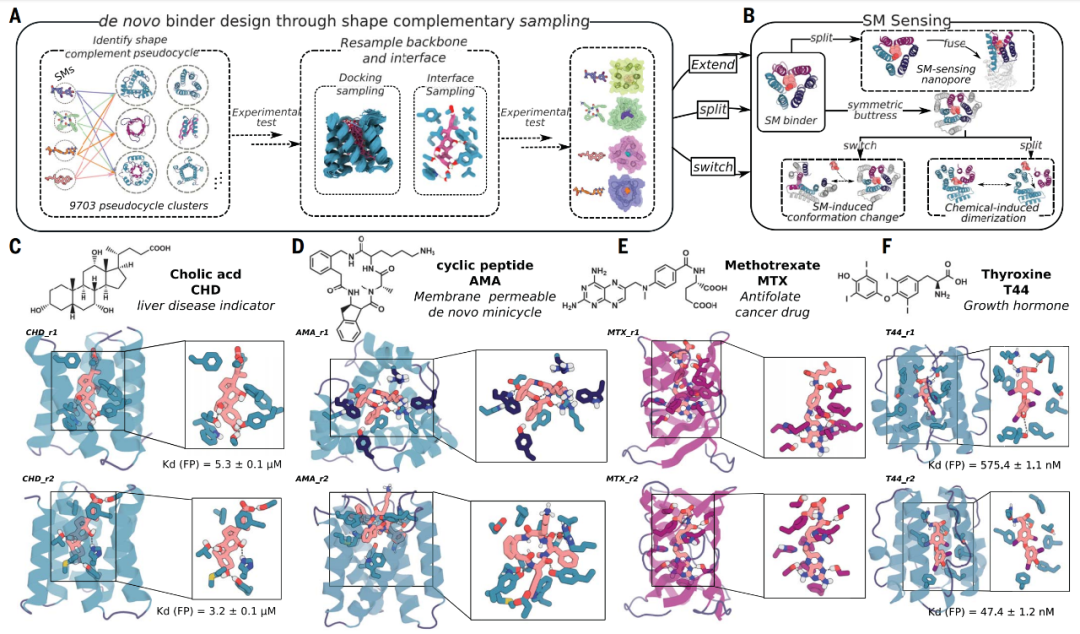
设计策略
由此出发,研究团队开发了一种集成深度学习和基于Rosetta能量设计的通用方法,为任何所需的小分子生成高形状互补的、基于假环肽的结合蛋白。研究团队选择了四种小分子作为结合靶点——胆酸(CHD)、甲氨蝶呤(MTX)、甲状腺素(T44)以及一种全新设计的细胞渗透性环四肽(AMA)。CHD是主要的胆汁酸,检测其游离形态对判断肝脏疾病有重要意义;MTX是一种抗叶酸癌症治疗剂,需要定期血液监测以减少患者的不良后果;T44是一种调节能量使用和其他功能的人体激素,家庭监测游离T44水平可用于甲状腺疾病诊断。
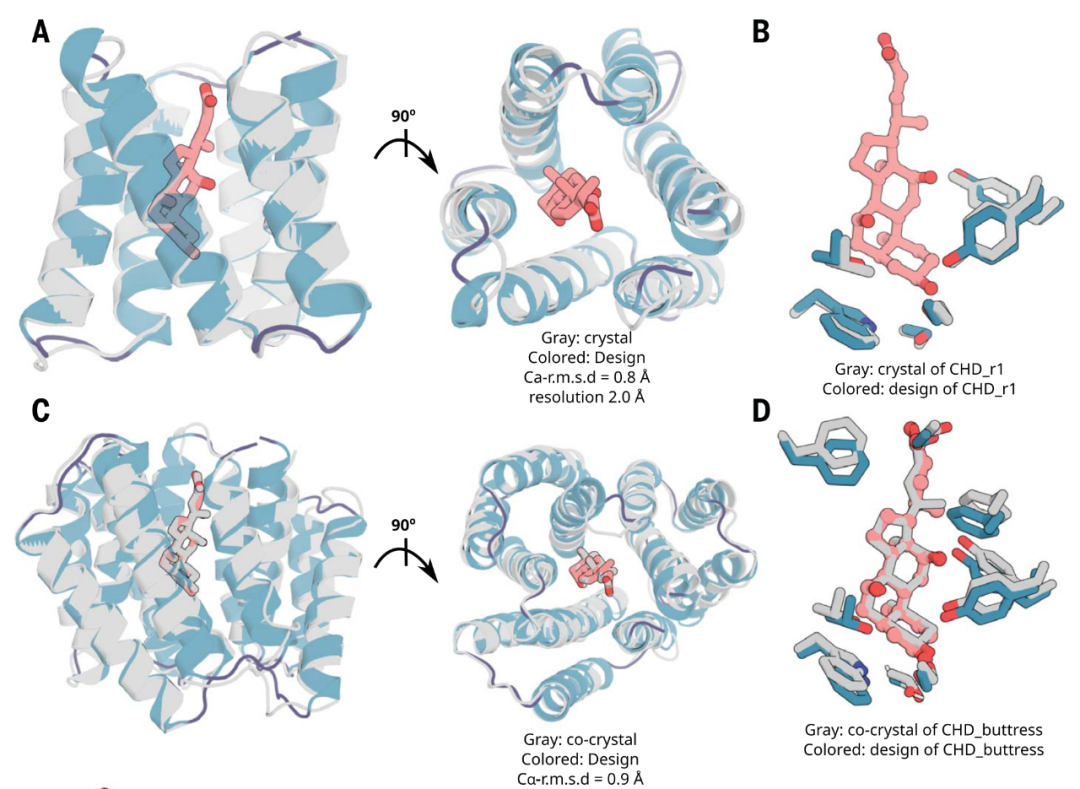
X射线晶体学证明了设计方法的准确性
基于深度学习技术,David Baker 团队生成了具有重复结构单元的假环肽,这些结构单元围绕着中心形成口袋状结构,其形状取决于重复单元的几何形状和数量。紧接着,研究团队将上述四种小分子对接到这些假环肽的口袋状结构中,设计高结合亲和力的相互作用表面,并通过实验筛选确定具有最高亲和力的设计。
具体来说,研究团队将目标小分子的不同构象对接到包含大量中心口袋的深度学习生成的假环肽中,其界面序列经过优化可进行高亲和力结合。其中,排名靠前的设计进行实验测试,并对最佳对接的骨架进行广泛的重新采样。序列设计完成后,对排名靠前的第二轮设计进行实验检验。
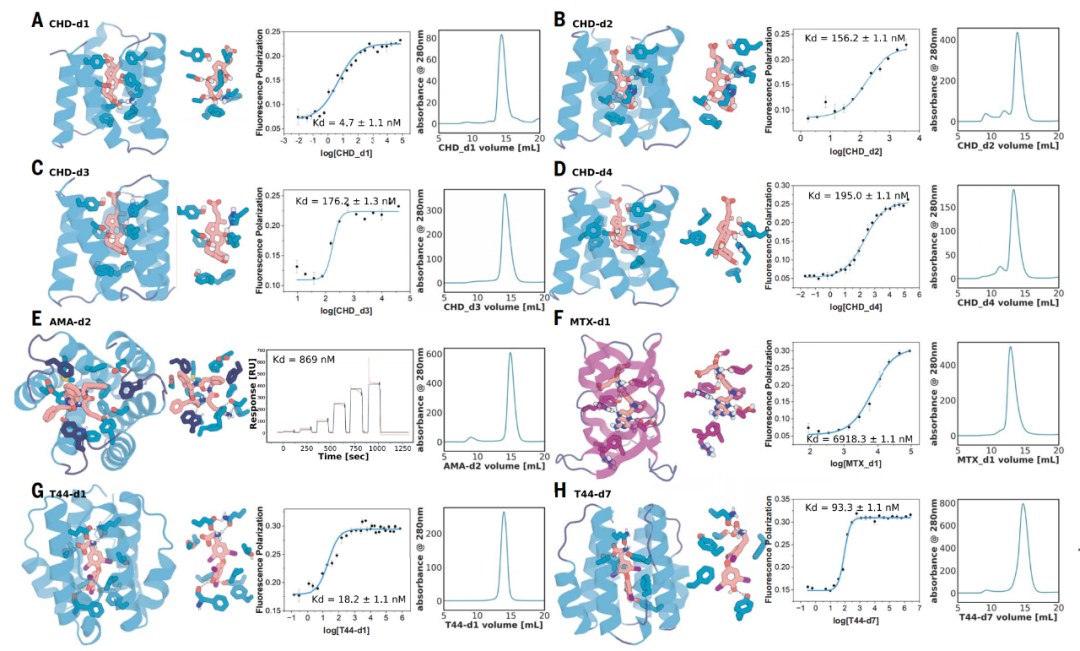
对选定的第二轮设计的实验表征
David Baker 团队通过X射线晶体学证明了设计方法的准确性,并获得了对这4种化合物具有高亲和力的小分子结合蛋白。不仅如此,由于这些人工设计假环肽是多个独立折叠的结构域,并依赖与靶标小分子结合驱动关联整合,因此这些人工设计的小分子结合蛋白可以很容易地整合到配体门控通道和化学诱导二聚化(CID)系统中,最终转化为生物传感器。
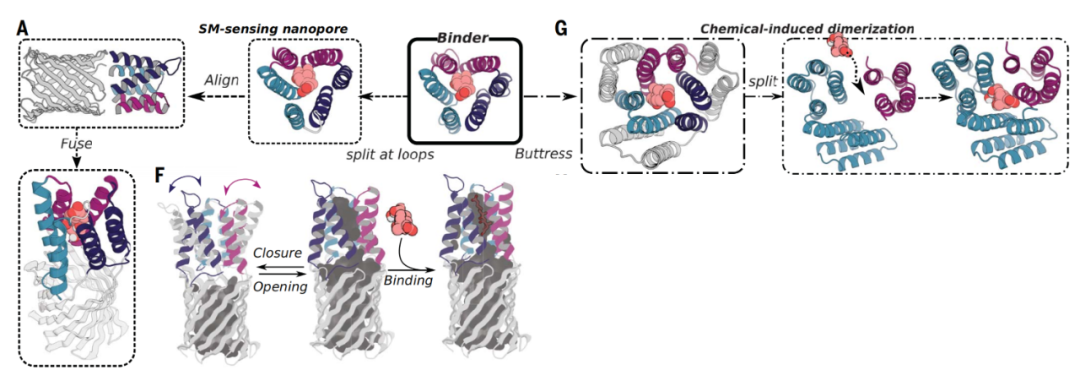
基于假环肽的小分子结合蛋白可以很容易转化为配体门控通道和CID系统
总而言之,这项发表于 Science 的研究展示了通过深度学习模型来设计基于假环肽的、具有模块化重复结构和中央结合口袋的小分子结合蛋白,对靶标小分子化合物具有高结合亲和力,并可以实现下游传感。
David Baker 团队表示,这种基于形状互补的蛋白质从头设计方法能够为各种生物学感兴趣的分子生成强大的、配体响应的门控通道和生物传感器。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


